Понятие о выравнивании. Эволюционное выравнивание
1.Построение вручную выравнивания фрагментов последовательности двух родственных белков
1) Скопировала пару коротких последовательностей из таблицы (которая дана в заданиях) в файл shortseqs.fasta. Последовательности соответственно в fasta-формате.
2) Запустила программу GeneDoc и импортировала этот файл.
3) Сделала выравнивание последовательности, стараясь, чтобы было сопоставлено максимальное число одинаковых букв. Неожиданно оказалось, что самым оптимальным является выравнивание, вообще не содержащее гэпов (то есть n-ая буква одной последовательности соответствует n-ой букве второй). Если Bас терзают смутные сомнения, то Вы можете проверить это сами :).
 Следующие работы показали, что такое "выравнивание" на самом деле является наилучшим, если учитывать вес выравнивания!
Следующие работы показали, что такое "выравнивание" на самом деле является наилучшим, если учитывать вес выравнивания!
4) Сохранила выравнивание под именем alignment1.msf.
5) Рассчитаем процент идентичности двух последовательностей:
число колонок с одинаковыми буквами - 10;
общее число колонок выравнивания - 20;
процент идентичности - (10 / 20) * 100% = 50%
Пользуясь матрицей сходства BLOSUM62, рассчитаем процент сходства двух последовательностей:
число колонок с одинаковыми буквами - 10;
число колонок с буквами, соответствующими сходным остаткам, - 1;
общее число колонок выравнивания - 20;
процент идентичности - ((10 + 1) / 20) * 100% = 55%
2. Пользуясь возможностями Excel построение карты локального сходства последовательностей из задания 1.
мой файл эксель
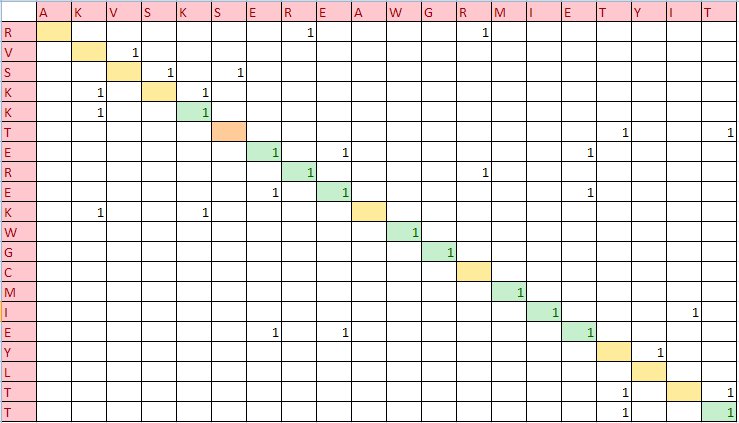
Зеленым отмечены ячейки, где "пересекаются" одинаковые буквы; желтым - любые, рыжим - сходные. Таким образом, путь выравнивания получается просто диагональным, так как в выравнивании нет гэпов.
3. Пользуясь программой bl2seq сделала выравнивание первого фрагмента из задания 1 с последовательностью моего белка.
Было найдено два выравнивания, в одном из которых последовательности полностью совпадают(координаты выравния 82 - 101 в моем белке). Также найден второй участок, который похож на искомую последовательность(координаты 2-18 в обеих последовательностях, идентичность 41%).
Query start position Subject start position
Score = 43.9 bits (102), Expect = 3e-12, Method: Compositional matrix adjust.
Identities = 20/20 (100%), Positives = 20/20 (100%), Gaps = 0/20 (0%)
Query 82 AKVSKSEREAWGRMIETYIT 101
AKVSKSEREAWGRMIETYIT
Sbjct 1 AKVSKSEREAWGRMIETYIT 20
Score = 11.2 bits (17), Expect = 1.6, Method: Compositional matrix adjust.
Identities = 7/17 (41%), Positives = 8/17 (47%), Gaps = 0/17 (0%)
Query 2 KVTKSEIVISAVKPEQY 18
KV+KSE E Y
Sbjct 2 KVSKSEREAWGRMIETY 18
4. Пользуясь сервисом bl2seq выровняйте последовательность вашего белка с последовательностью гомологичного (родственного) белка
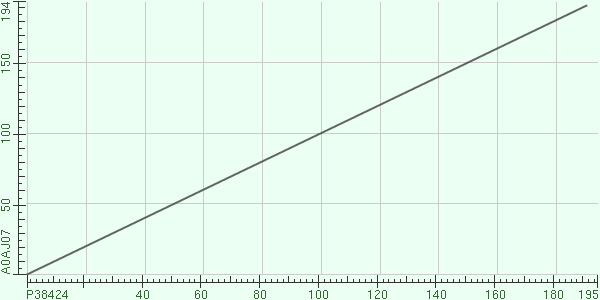
Я сравнила два белка: свой(ID ENGB_BACSU, AC P38424) с другим: ID ENGB_LISW6, AC A0AJ07.
Мой белок взят из организма bacillus subtilis, а белок ENGB_LISW6 взят из организма Listeria welshimeri.
Процент идентичности - 72%, сходства - 85%, гэпов в выравнивании нет (разрывов и гэповых колонок, соответственно, тоже). Координаты выравнивания - 1-191 (в обеих последовательностях).
Карта локального сходства
©Melnichuk Anastasia

