Ключевые слова
Herbaspirillum seropedicae, геном, протеом
Резюме
В настоящей работе представлены результаты анализа генома бактерии Herbaspirillum seropedicae. Изучение проводилось для получения основных статистических данных о геноме и протеоме: длина и нуклеотидный состав генома, встречаемость к-меров, сравнение длин кодируемых белков и распределение генов по цепочкам молекулы ДНК. Получены они были в ходе работы с файлом генома в формате fasta и таблицей генов (содержит названия генов, их местоположение на хромосоме, продукты и пр.). Результаты также представлены в виде таблиц.
Введение
Изучение генома бактерий необходимо для увеличения информационной базы о работе конкретных генов. Бактерии часто содержат белки, необходимые для грамотной адаптации к среде, в том числе – к экстремальным условиям. Причины и способы таких приспособлений можно проследить в геноме через последовательность генов и состав белков, которые они кодируют. В дальнейшем появится возможность задавать клетке необходимые функции и свойства через изменение генома по данным, полученным в ходе подобных анализов.
Род Herbaspirillum относится к протеобактериям, типу, объединяющему грам-отрицательные бактерии, как патогенные, так и азотфиксирующие.
Данный вид относится к азотфиксирующим бактериям и вступает в симбиоз с такими растениями как кукуруза, рис, сорго, сахарный тростник и т.д. При чем Herbaspirillum seropedicae - представитель эндофитных бактерий, то есть он колонизирует межклеточное пространство тканей растения и вступает в азотфиксирующие ассоциации с растением. Исследования показали, что отдельно от растений, непосредственно в почве, H. seropedicae имеет низкую выживаемость [1]. Что характерно, H. seropedicae не наносит растениям ущерб и не является патогеном [2]. На основе этого можно предположить, что отношения бактерия-растение взаимовыгодные, хотя до конца механизмы химического взаимодействия между ними не изучены
Начинается колонизация растения с попадания в корни, преимущественно в зону корневых волосков, по средству хемотаксиса. Большая часть бактерий остаётся в этой части растения, но некоторые проникают внутрь через неровности эпидермиса, занимая межклеточные полости. Также бактерии могут проникать выше, колонизируя листья и побеги. Передвигаются они преимущество по ксилеме [3].
Геном Herbaspirillum seropedicae, а точнее штамма SmR1, был секвенирован. В общей сложности кольцевая молекула ДНК этой бактерии содержит 4804 гена [4]. Были обнаружены и изучены гены, участвующие в фиксации азота. Но привлекла внимание ученых бактерия не этим. Как и у большинства эндофитов, у Herbaspirillum seropedicae многие жизненно важные клеточные процессы завязаны на железе. Чтобы эффективно усваивать железо, бактерия производит siderophores serobactins (серобактины). Однако были обнаружены и другие гены, также участвующие в приобретении этого металла, что натолкнуло исследователей на изучение альтернативных методов взаимодействия бактерии и железа, а также более подробный анализ работы некоторых генов [5].
В настоящее время Herbaspirillum seropedicae наиболее известна именно как объект генных исследований, так как, во первых, важна для сельскохозяйственных культур (например, в симбиозе с сельскохозяйственными культурами, колония бактерий может увеличить урожайность почти на 50% [4]), а во-вторых, обладает сложными метаболическими схемами, как в отношении фиксации азота, так и при нахождении в стрессовых ситуациях нехватки железа.
Материалы и методы
Как было отмечено выше, данные были взяты из двух основных файлов: генома бактерии формата fasta [6] и таблицы генов [7].
Подробную информацию о геноме Herbaspirillum seropedicae (в том числе и сам секвенированный геном) можно найти по ссылке:
Ссылка на геномПомимо этого, в ходе работы составлялись дополнительные таблицы, которые можно найти в сопровождающих материалах в двух категориях: геном и протеом.
Основные вычисления и анализ генома были сделаны на языке программирования Python. В сопроводительных материалах в категории «использованные программы» можно найти ссылку на них.
Алгоритм и использование данных программ подробнее описаны в соответствующих пунктах «Результатов и обсуждения».
Результаты и обсуждение
1 Анализ генома
1.1 Основные данные о геноме
Геном Herbaspirillum seropedicae представлен одной кольцевой хромосомой, общее число нуклеотидов – 5509723. Это было посчитано с использованием программы «standart_data.py». Алгоритм предельно простой: последовательность считывается посимвольно, после чего каждый символ идентифицируется как нуклеотид и считается их сумма. Параллельно идет подсчет количества нуклеотидов каждого типа (подробнее в следующем абзаце).
Также был рассчитан GC-состав ДНК. GC-состав последовательности дает представление о доли гуанина и цитозина в молекуле и считается по следующей формуле:
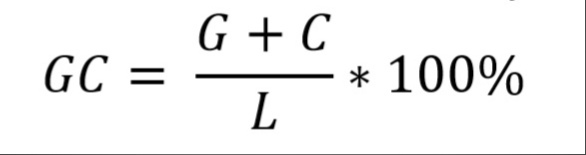
где G и C обозначено количество нуклеотидов гуанина и цитозина соответственно, L – общая длина последовательности, а GC – искомый GC-состав в процентах.
В случае Herbaspirillum seropedicae, GC-состав имеет вид:
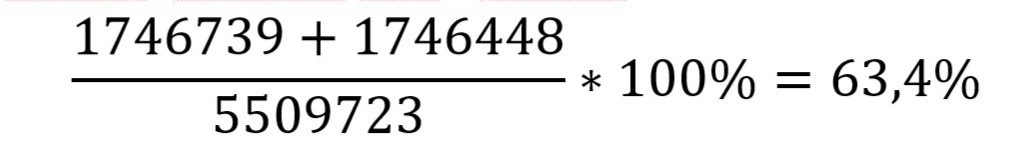
GC-содержание ДНК (или РНК) показывает, насколько устойчива данная молекула к денатурации в растворе. Это объясняется формой связи между азотистыми основаниями: цитозин и гуанин образуют три связи, а аденин и тимин – две. Поэтому чем выше доля пар гуанин-цитозин, тем молекула устойчивее.
Полученное процентное содержание говорит о том, что молекула довольно прочная, показатель не максимальный, но выше среднего, значит, быстрой денатурации в растворе не происходит
1.2 Нуклеотидный состав
Помимо содержания гуанина и цитозина, было рассчитано содержание аденина и тимина. Данные были получены с помощью программы «standart_data.py». Результаты представлены в таблице 1:
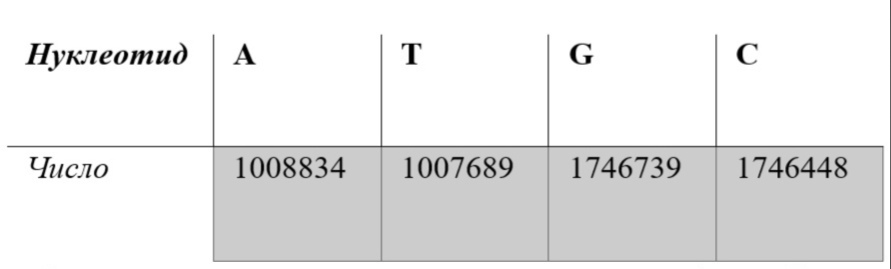
Однако, если сложить все значения и сравнить с общим числом нуклеотидов (5509723), станет очевидно, что значения не совпадают. Это объясняется наличием в последовательности несеквенированных нуклеотидов.
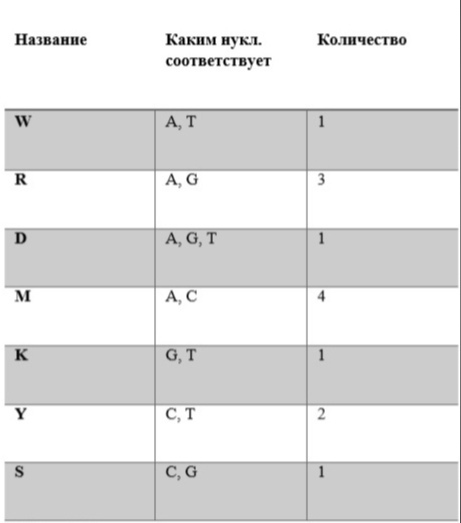
Несеквенированные нуклеотиды также имеют однобуквенные обозначения. Ими обозначают нуклеотид, который не удалось определить. Например, W обозначение для аденина или тимина, D – аденина, гуанина или тимина, и т.д. [8]
Встречаемость несеквенрованных нуклеотидов представлена в таблице 2. Эти данные были получены в ходе работы программы «other_nuclioides.py»
Из таблицы 1 можно заметить, что число нуклеотидов с аденином приблизительно равно числу нуклеотидов с тимином (разница составляет всего 1145), а гуанина – цитозину (разница всего 291 нуклеотид). Значит, выполняется второе правило Чаргаффа, о равенстве частот встречаемости нуклеотидов на одной цепи ДНК.
1.3 Анализ статистики k-меров в геноме для фиксированного k
Анализировалось встречаемость k-меров с k, равным трем. Важно отметить, что здесь встречаемость k-меров не совпадает со встречаемостью кодонов, так как при подсчете числа k-меров не учитывается свойство неперекрываемости генетического кода.
Программой «k-mers.py» было посчитано количество вхождений для каждого k-мера. Результаты можно увидеть на гистограмме 1.
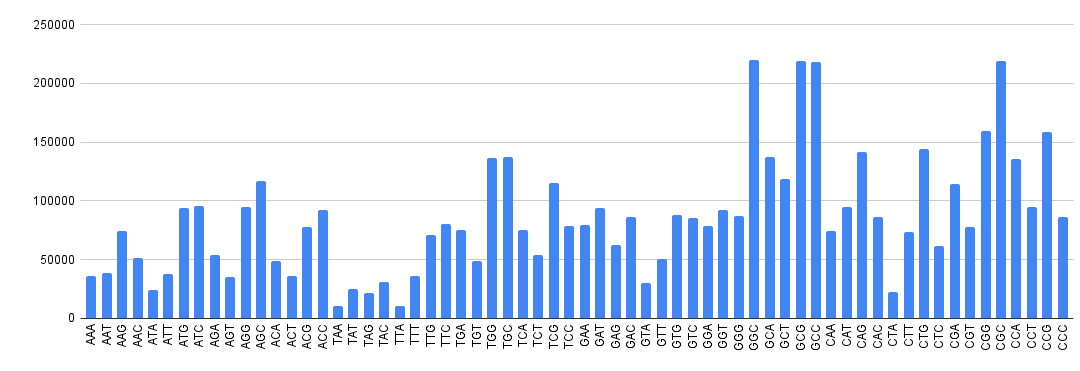
Можно заметить, что наибольшее количество вхождений у k-меров, содержащих только гуанин и цитозин, таких как: GGC, GCG, GCC и CGC. Это подтверждает вычисления GC-состава генома, приведенные выше.
Однако такое представление не самое понятное для восприятия и не очень удобное в плане статистики.
Поэтому для всех k-меров была рассчитана ожидаемая и реальная частоты встречаемости (в программах «expected_k-mers.py» и «real_k-mers.py» соответственно). Ожидаемая встречаемость считалась на основе встречаемости каждого входящего в k-мер нуклеотида. Встречаемость нуклеотидов в свою очередь оценивалась по данным из таблицы 1, по формуле:
Число нуклеотидов данного типа / Общее число нуклеотидов
Таким образом получили следующие данные:
встречаемость А = встр.Т = 0.18
встречаемость G = встр. С = 0.317
Далее была получена гистограмма с ожидаемой встречаемостью различных k-меров (гистограмма 2).
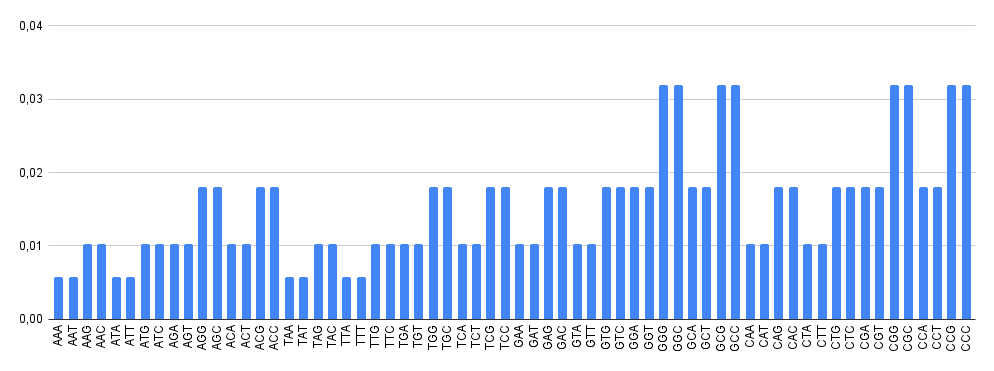
Однако полученная реальная встречаемость выглядит несколько иначе (гистограмма 3).
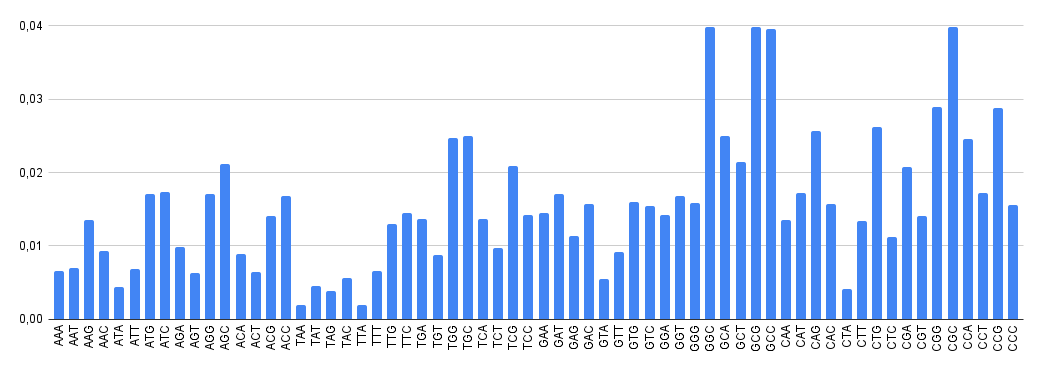
Заметно, что общий рисунок сохраняется (наибольшую встречаемость ожидаемо сохраняют k-меры с гуанином и цитозином в составе), хотя локальные изменения все же есть (например, резко сниженное в сравнении с ожидаемым число k-меров GGG или повышенное содержание CGC или АТС).
Для более четкого представления различия между ожидаемыми и реальными значениями был рассчитан композиционный сдвиг (или Compositionsal Bias). Вычисляется данный сдвиг по следующей формуле:
cd = Наблюдаемая встречаемость / Реальная встречаемость
По полученным данным была построена гистограмма cd для различных k-меров (гистограмма 4).
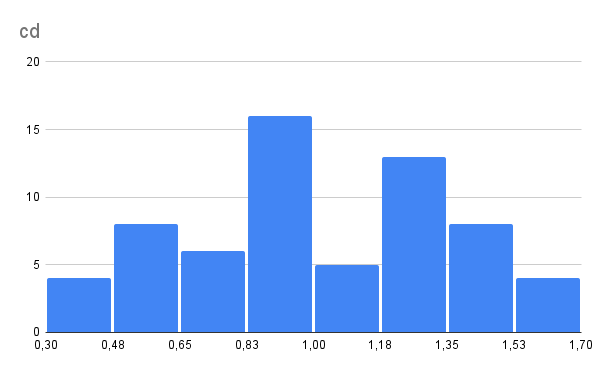
По гистограмме видно, что наиболее частый сдвиг – немного меньше единицы (диапазон 0,83 – 1), то есть наибольший процент k-меров имеют реальную встречаемость чуть меньше ожидаемой. Но при этом следующий по встречаемости диапазон – 1.18-1.35, k-меры, у которых реальная встречаемость выше. Таким образом, у большинства нуклеотидов различие между реальной и ожидаемой встречаемостью есть, но незначительное.
Однако есть также экстремальные варианты, попадающие в диапазоны 0.3-0.48 и 1.53-1.7. Минимальное значение cd наблюдается у ТАА – 0,326, а максимальное у АТС – 1,693. В первом случае реальная встречаемость оказывается в три раза меньше ожидаемой. Вероятно, это связано с тем, что в норме кодон ТАА (UAA) является стоп кодоном, преимущественно встречающимся в митохондриальной ДНК, то есть объяснить его встречаемость встречаемостью составляющих нуклеотидов нельзя. Другой экстремальный случай – k-мер АТС. Его встречаемость можно связать с тем, что кодон АТС комплементарен стоп кодону UAG. Надо отметить, что встречаемость k-мера GAT (GAU) также очень высокая, cd = 1,666.
2. Анализ протеома
2.1 Гистограмма длин белков
На основе таблицы Genome table [7] была построена гистограмма длин кодируемых белков (гистограмма 5).
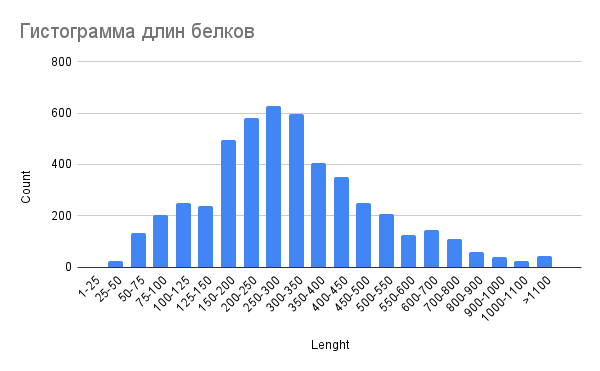
Можно заметить, что наибольшая доля белков имеет длину в диапазоне 200-350 (примерно 1800 различных белков попадают в этот карман). Медиана составляет 295, а среднее значение – 337,29.
Максимальные и минимальные значения составляют 7910 и 29 соответственно.
2.2 Распределение генов белков по цепям ДНК
По данным таблицы Genome table, были получены следующие данные (таблица 3):
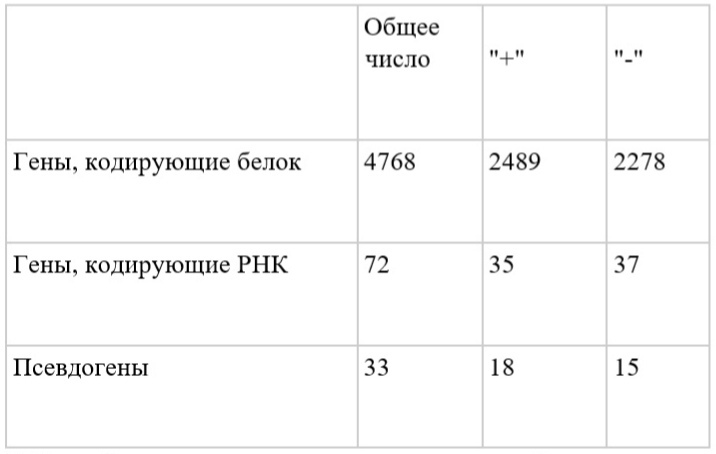
Из полученных данных следует, что распределение по цепям примерно равномерное. В случае белок кодирующих генов на «+» цепи 0,52 от всех генов, а на «-» цепи – 0,48. Такое распределение возможно получить случайно.
Заключение
Результатом данного исследования является оформленная статистическая информация о геноме и протеоме бактерии Herbaspirillum seropedicae. Были сделаны некоторые предположения относительно данных, отличающихся от ожидаемых (например, о природе аномально низкой встречаемости k-мера АТС), однако они требуют дальнейшей проверки и изучения.
Подводя итог проделанной работе, стоит отметить, что Herbaspirillum seropedicae является интересным объектом для дальнейшего изучения. К примеру, можно уделить внимание описанному несоответствию ожидаемой и реальной встречаемости k-мера ТАА в геноме, определить наиболее часто встречающиеся стоп и старт кодоны.
Сопроводительные материалы
Дополнительные таблицы:
- Геном: Ссылка
- Протеом: Ссылка
Использованные программы:
СсылкаСсылки на литературу
[1] - Baldani JI, Pot B, Kirchhof G, Falsen E, Baldani VLD, et al.(1996) Emended description of Herbaspirillum; inclusion of (Pseudomonas) rubrisubalbicans, a mild pathogen, as Herbaspirillum rubrisubalbicans comb. nov., and classification of a group of clinical isolates (EFgroup 1) as Herbaspirillum species 3. Int J Syst Bacteriol 46: 802– 810.
[2] - Пиментел Дж. П., Оливарес Флорида, Питард Р. М., Уркиага С., Акиба Ф. и др. (1991) Динитрогеновая фиксация и инфицирование листьев травы Pseudomonas rubrisubalbicans и Herbaspirillum seropedicae . Почва для растений 137: 61–65.
[3] - Herbaspirillum-plant interactions: microscopical, histological and molecular aspects Rose Adele Monteiro, Eduardo Balsanelli, Roseli Wassem, Anelis M. Marin, Liziane C. C. Brusamarello-Santos, Maria Augusta Schmidt, Michelle Z. Tadra-Sfeir, Vânia C. S. Pankievicz, Leonardo M. Cruz, Leda S. Chubatsu, Fabio O. Pedrosa & Emanuel M. Souza
[4] - Genome of Herbaspirillum seropedicae Strain SmR1, a Specialized Diazotrophic Endophyte of Tropical Grasses Fábio O. Pedrosa ,Rose Adele Monteiro,Roseli Wassem,Leonardo M. Cruz, Ricardo A. Ayub,Nelson B. Colauto,Maria Aparecida Fernandez,Maria Helena P. Fungaro,Edmundo C. Grisard,Mariangela Hungria,Humberto M. F. Madeira,Rubens O. Nodari,Clarice A. Osaku
[5] - Herbaspirillum seropedicae Differentially Expressed Genes in Response to Iron Availability María F. Trovero, Paola Scavone, Raúl Platero, Emanuel M. de Souza, Elena Fabiano and Federico Rosconi Bardet, G. (1920) Sur un syndrome d’obesite infantile avec polydactylie etretinite pigmentaire (contribution a l’etude des formes cliniques de l’obesite hypophysaire). PhD Thesis, name of institution, Paris, France.
[6] - ссылка на файл с геномом
[7] - Ссылка на таблицу генов
[8] - Здесь можно найти полную таблицу названий несеквенированных нуклеотидов