Исследуемый белок можно найти у различных штаммов бактерии Bradyrhizobium diazoefficiens.
Бактерии рода Bradyrhizobium грамотрицательные, обитающие в почве и часто вступающие в симбиоз с растениями. Некоторые виды способны к фиксации атмосферного азота. Предположительно, данный вид также приспособлен к фиксации азота, так как найдены подтверждения его симбиотических отношений с соей[1].
Долгое время существовала путаница в таксономии. Так, например, исследуемый вид Bradyrhizobium diazoefficiens был выделен в отдельный вид только в 2013 году, а до этого времени его относили как штамм к виду Bradyrhizobium japonicum[2].

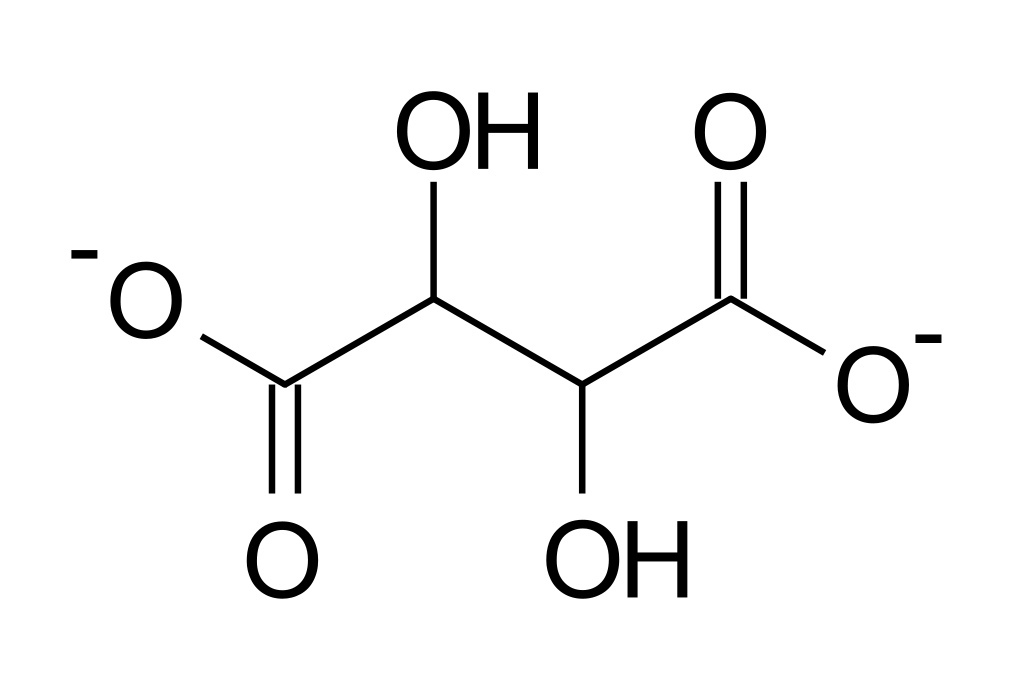
Русский эквивалент названия - дегидратаза D-тартрата.
Основная информация была взята из базы данных UniProtKB. Ознакомиться со страницей белка можно здесь: https://www.uniprot.org/uniprot/Q89FH0
Белок D(-)-tartrate dehydratase в клетке бактерии катализирует реакцию дегидратации D-тартрата до оксалоацетата. По классификации ферментов относится к классу лиаз.
В общем случае тартраты являются производными винной кислоты (соли и эфиры). Тартраты используются в пищевой промышленности в качестве антиоксидантов. В химии тартраты используются как хиральные лиганды в комплексообразовании[3].
Через расширенный поиск UniProt было обнаружено, что тем же геном tarD кодируется 6 не бактериальных белков, однако их нет в базе Swiss-Prot. Можно сделать вывод, что такой метаболический процесс есть только у прокариотических организмов.
Кофактором белка является ион Mg2+, причем соблюдается соотношение: молекула белка : ион кофактора = 1:1. Также через расширенный поиск было установлено, что у этой бактерии еще 10 (по базе Swiss-Prot) различных лиаз связываются с ионом магния в качестве кофактора. То есть магний играет довольно значимую роль в метаболизме. Вероятно, Mg2+, как и другие ионы металлов, бактерия получает от симбионта – растения.
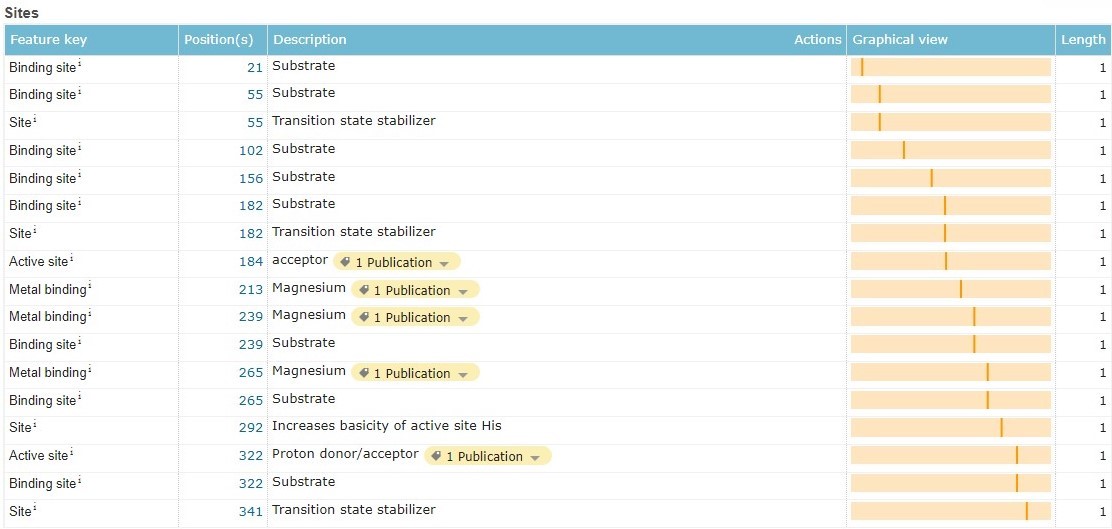
Связывание с магнием происходит в трех активных центрах (сайтах). Всего сайтов у D(-)-tartrate dehydratase 17, из которых 8 отвечают за связывание непосредственно с субстратом.
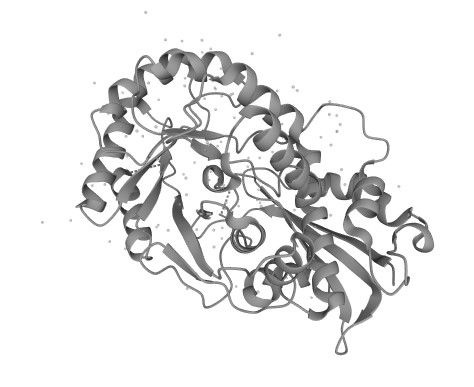
У белка изучена третичная структура и есть ее 3D-модель:
По мимо этого было изучено влияние мутаций в последовательности аминокислот на третичную структуру. Как можно заметить из таблицы, мутации преимущественно приводят к прекращению или снижению каталитической активности.
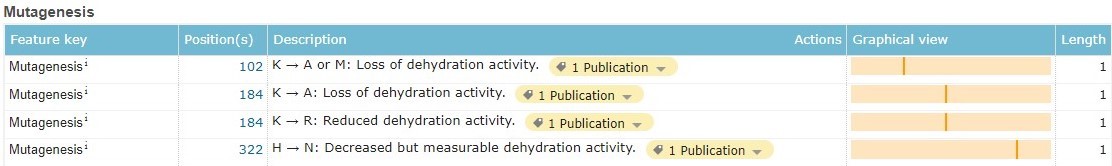
С одной стороны, это довольно очевидный факт, а с другой – не было выявлено других функциональных мутаций, при которых, например, прекращалось бы связывание с магнием, или белок не сворачивался бы вовсе. Скорее всего, такие мутации есть, просто их сложнее детектировать: проще заметить, что белок перестал выполнять свои функции, чем уменьшение способности связываться с кофактором.
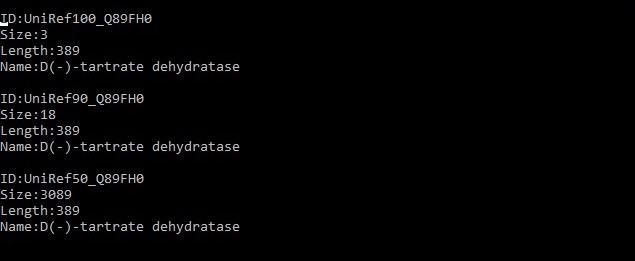
Изучение кластеров проводилось по трем значениям точности: 100%, 90% и 50%, с соответствующими запросами UniRef*_Q89FH0, где вместо "*" - 100, 90 и 50. D(-)-tartrate dehydratase относится к кластеру D(-)-tartrate dehydratase со всеми проверяемыми значениями. Краткая информация о кластерах:
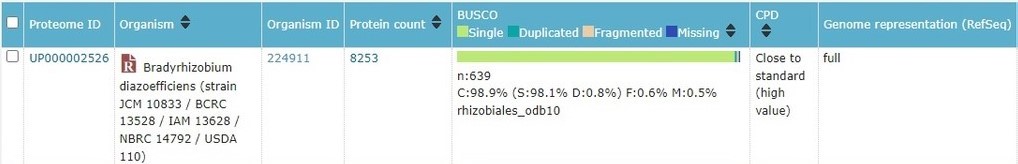
При общем рассмотрении протеома бактерии Bradyrhizobium diazoefficiens можно заметить, что он референсный (то есть был отобран в качестве лучшего из доступных геномов данной таксономической группы), состоит из 8253 различных белков, при чем согласно алгоритму BUSCO, процент сходства или наличия представителей референсных ортологических групп генов составляет 98.1%. Это говорит о высокой точности определения белков протеома. По системе сравнения размеров протеома с близкородственными группами (CPD) ему был присвоен статус Close to standard.
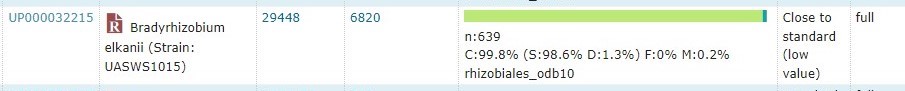
В качестве контрольного протеома я взяла протеом другого вида бактерий того же рода: Bradyrhizobium elkanii. Этот вид также является азотфиксирующим и может вступать в эндосимбиоз с соей. Однако только этот вид вызывает обесцвечивание листьев или же заболевание, известное как 'микросимбионт-индуцированный листовой хлорозис' [4]. На основании устойчивости к некоторым антибиотикам и отличиях в составе жирных кислот, в 1988 году Bradyrhizobium elkanii был присвоен статус вида (до этого он рассматривался как штамм B. Japonicum) [5].
В протеоме этой бактерии суммарно 6820 белков, геном полностью представлен в RefSeq, высокие показатели точности BUSCO.
Сравнение протеомов проводилось по нескольким параметрам. Во-первых, сравнивалась доля различных групп белков. Результат можно увидеть в таблице ниже:

Запросы в UniProt имели следующий вид:
Трансмембранные: annotation:(type:transmem) AND organism:[название вида] AND proteome:[id протеома]
Ферменты: ec:* AND organism:[название вида] AND proteome:[id протеома]
Азот фиксирующие: annotation:(type:function 'nitrogen fixation') AND organism:[название вида] AND proteome:[id протеома]
Легко заметить, что доля трансмембранных белков и ферментов примерно одинакова. Основное различие - в наличии белков, участвующих в фиксации азота у B. Diazoefficiens. Выше упоминалось, что в теории бактерии вида B. Elkanii могут иметь способность к азот фиксации, однако отсутствие необходимых белков говорить о том, что конкретно у рассматриваемого штамма UASWS1015 такой способности нет. Логично предположить, что вещества для азотистого обмена бактерия берет от растения. Сопоставляя отсутствие способности к фиксации азота и болезнь листьев, вызываемую данным видом, можно сказать, что данный штамм является паразитическим.
Другой критерий сравнения протеомов – начальная аминокислота белков. Как правило, белки начинаются с метионина, но он может убираться, например, в ходе посттрансляционной модификации.[6]
Для сравнения была написана программа на python, которая поэтапно считывала информацию о белках из файла протеома, находила строки с последовательностью аминокислот и проверяла первую букву. Файлы протеома были скачаны через командную строку следующими запросами:
wget 'https://www.uniprot.org/uniprot/?query=organism:"Bradyrhizobium diazoefficiens (strain JCM 10833 / BCRC 13528 / IAM 13628 / NBRC 14792 / USDA 110) [224911]" AND proteome:up000002526&format=txt&compress=yes' -O up000002526.swiss.gz
wget 'https://www.uniprot.org/uniprot/?query=organism:"Bradyrhizobium elkanii [29448]" AND proteome:up000032215&format=txt&compress=yes' -O up000032215.swiss.gz
Для изучаемого и контрольного образцов соответственно. Код программы можно посмотреть здесь: https://drive.google.com/file/d/1tRz9cf52brJkZVGWS31IWsnHXu7vKvAV/view?usp=sharing
Для изучаемой бактерии Bradyrhizobium diazoefficiens программа дала нулевой результат – все белки протеома начинаются с метионина. Интереснее вышла ситуация с Bradyrhizobium elkanii: программа выдала целый кластер последовательностей, которые начинались с других аминокислот (целый спектр, включающий треонин, серин, аргинин и т.д.). Для определения названия этих белков программа 1 была доработана, вторую версию можно посмотреть здесь:https://drive.google.com/file/d/1fZPY7jX0zy891irG_uTh4qyehF0Px3SV/view?usp=sharing
По итогу мы получили ряд белков с ID вида: A0A0D1****_BRAEL. По этим ID на UniProt можно найти записи с их описанием – изменение начальной аминокислоты может быть связано с функциями белка. Была теория, что белки связаны с болезнетворной деятельностью этого штамма, так как с изучаемым образцом эти виды близкородственные, и главное различие заключается в том, что Bradyrhizobium elkanii вызывает болезнь растений, а Bradyrhizobium diazoefficiens – нет.
После проверки части белков из полученного списка, теория была опровергнута – функционально белки между собой не связаны. Однако общая черта все-таки есть – последовательности созданы автоматически на базе генома и являются предварительными данными. Так что скорее всего данная аномалия является продуктом систематической компьютерной ошибки и к биологии отношения не имеет.
Таким образом, сравнение протеомов двух близкородственных видов дало информацию об отсутствии способности к фиксации азота у паразитического штамма, других кардинальных изменений в составе белков не обнаружилось.
[1] -Rivas R., Martens M., De Lajudie P., Willems A. Multilocus sequence analysis of the genus Bradyrhizobium // Systematic and Applied Microbiology. — 2009. — Vol. 32. — P. 101–10. — doi:10.1016/j.syapm.2008.12.005 — PMID 19201125
[2] -Delamuta JR, Ribeiro RA, Ormeno-Orrillo E, Melo IS, Martinez-Romero E, Hungria M. Polyphasic evidence supporting the reclassification of Bradyrhizobium japonicum group Ia strains as Bradyrhizobium diazoefficiens sp. nov. Int J Syst Evol Microbiol 2013; 63:3342-3351 https://pubmed.ncbi.nlm.nih.gov/23504968/
[3] -Evolution of enzymatic activities in the enolase superfamily: D-tartrate dehydratase from Bradyrhizobium japonicum. Yew W.S., Fedorov A.A., Fedorov E.V., Wood B.M., Almo S.C., Gerlt J.A. http://dx.doi.org/10.1021/bi061688g
[4] -T. E. Devine, L. D. Kuykendall & J. J. O’Neill. DNA homology group and the identity of bradyrhizobial strains producing rhizobitoxine-induced chlorosis on soybeans (англ.) // Crop Science : journal. — 1988. — Vol. 28. — P. 939—941.
[5] -L. D. Kuykendall, M. A. Roy, J. J. O’Neill & T. E. Devine. Fatty acids, multiple antibiotic resistance, and DNA homology groups of Bradyrhizobium japonicum (англ.) // International Journal of Systematic and Evolutionary Microbiology (англ.)рус. : journal. — 1988. — Vol. 37. — P. 358—361.
[6] -Lodish H, Berk A, Zipursky SL, et al. (2000). "17.6, Post-Translational Modifications and Quality Control in the Rough ER". Molecular Cell Biology (4th ed.). New York: W. H. Freeman. ISBN 978-0-7167-3136-8.