
В прошлом практикуме я работала с Гавайским вороном (Corvus hawaiiensis) но при поиске через ftp.ncbi оказалось, что у него нет аннотированного CDS. Посколько вороны по-прежнему классные, для этого практикума я взяла его не очень далекого родственника - Американского ворона (Corvus brachyrhynchos). Другое его название - короткоклювый ворон. Он меньше своих сородичей, обитает в Северной Америке. Больше всего известен за распространение лихорадки Западного Нила.

В таблице скаффолдов GCA_000691975.1_ASM69197v1 я искала скаффолды, которые были бы короче 10 000 нуклеотидов и при этом содержали CDS. Но таких не нашлось, поэтому я взяла scaffold700 длиной 15524 нуклеотидов. Он относится к unplaced-scaffold.
Его идентификатор NW_008235546.1. По этому идентификатору на ncbi было найдено графическое отображение этого скаффолда
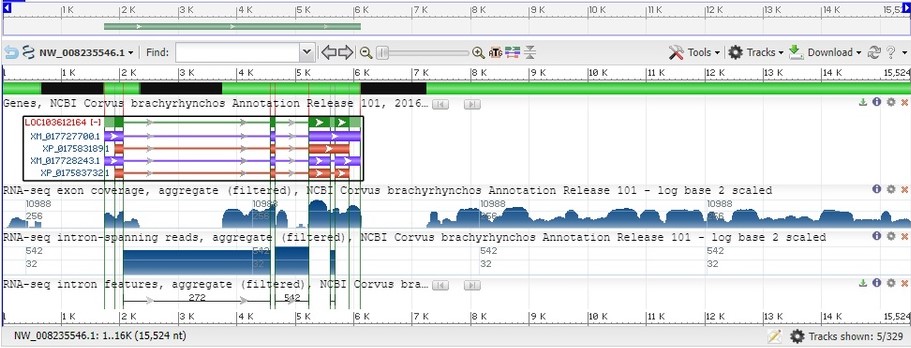
На верхней панели видно бледно-зеленый отрезок, который показывает положение гена LOC103612164 в положении 1,740-6,117. Ниже зеленым показаны экзоны, а черным - интроны. В рамке ниже фиолетовым показаны последовательности mRNA и красным - белок кодирующие. Этот ген кодирует zinc finger protein 239-like (белок, похожий на белок цинковых пальцев, который встречается у людей) в двух изоформах. Тонкие стрелки между блоками - куски, которые потом вырежутся.
Последовательность слишком длинная, поэтому я взяла ее отрезок из первых 10 000 нуклеотидов. Обрезанный скаффолд можно посмотреть в файле:
short_scaffold700.fastaМожно заметить, что там есть большие участки с N повторами.
blastn
Из поиска я исключила хордовых (taxid:7711) и изменила следующие параметры: максимальное число находок - 500, длина слова - 15 (максимальная). Это было сделано для того, чтобы найти максимально похожие последовательности. Исключение хордовых сильно сузило круг поиска, так как при исключении только рода Corvus почти все находкт были предсказанными генами различных птиц (лучшие значение были у зебровой амадины, зарянки и тибетской ложносойки). При заданных параметрах максимальный процент покрытия составил 10%. Предсказанных находок стало значительно меньше, в основном нашлись хромосомы. Всего находок 407, наибольшее совпадение с Phaedon cochleariae, это вид жуков-листоедов.
megablast
Для нахождения последовательностей с наибольшим совпадением, я исключила из поиска птиц (taxid: 8782), так как предполагаю, что у совсем дальних родственников вероятность найти гомологичную последовательность гораздо ниже*. Длина слова стандартная - 28. Всего находок было 157 и только три из них были не предсказанными. Интересно, что все три относились к рыбам - две у Salmo trutta (кумжа) и одна у Coregonus sp. (обыкновенный сиг).
* Для сравнения, результат выравнивания с исключением позвоночных это пять предсказанных последовательностей ZFP3 like белка китайской белой креветки.
blastx
С надеждой, что данный белок есть только у хордовых, я исключила их из запроса. Но результатов все еще было больше 1000 и ncbi не дает посмотреть их все. Лучшие совпадения были с разными видами рода Drosophila. E-value лучшей находки 7.7E-37.
tblastx
Поиск велся с исключением рода Corvus (taxid:30420). Ничего не нашлось, tblastx устал:(
blastn используется для поиска по нуклеотидной базе, когда нужно найти гомологов или аналогов к данной последовательности. Алгоритм не самый точный, поэтому для поиска идентичных или практически идентичных последовательностей используется megablast. С его помощью можно найти избыточные записи или гомологичные гены у близкородственных видов, т.е. последовательности с минимальными отличиями. megablast также как и blastn работает только в нуклеотидных базах. Для поиска в белковых базах данных есть алгоритм blastx. Он получает на вход последовательность нуклеотидов, транскрибирует ее и затем производит поиск гомологичных белков. Такой алгоритм может понадобится в ситуации, когда есть кодирующая часть ДНК, но первичная последовательность белка еще не аннотирована. tblastx использует похожий принцип действия, только в конечном итоге поиск ведется в нуклеотидной базе данных. То есть после получения белковой последовательности, tblastx затем переводит ее обратно в нуклеотидную, чтобы искать не гомологичные белки, а гены, кодирующие эти белки.
Индексация генома была сделана с помощью makeblastdb:
makeblastdb -in GCA_000691975.1_ASM69197v1_genomic.fna -dbtype nucl
Поиск последовательностей, кодирующих рРНК - конкретно 16s и 23s субъединицы - был сделан через следующие команды:
blastn -task blastn -query ecoli-16s.fasta -db GCA_000691975.1_ASM69197v1_genomic.fna -out blast16.out
blastn -task blastn -query ecoli-23S.fasta -db GCA_000691975.1_ASM69197v1_genomic.fna -out blast23.out
1. 16s и 23s рРНК это характерные рибосомальные РНК прокариот. Это малая и большая субъединицы соответственно. Поскольку мы смотрим геном эукариота, логично ожидать, что основные рибосомы у него будут характерными для эукариот - 18S, 5S и 28S. Есть вероятность найти интересующие нас гены в митохондриальной ДНК, так как по теории симбиогенеза митохондрии имеют бактериальное происхождение.
2. Использовался поиск через blastn, потому что мы сравниваем две нуклеотидные последовательности.
3. Для 16s р РНК было найдено 17 скаффолдов, но они все относились к isolate BGI_N302 unplaced genomic scaffold. Аннотации у них нет. Лучшая находка имела значение e-value 4e-05, остальные - 0.78 и выше. Для 23s рРНК всего 11 находок, из которых только одна является контигом - isolate BGI_N302 contig12029 - а остальные, как и в прошлом запросе, скаффолды. Лучшее значение e-value 3e-10. Аннотаций также нет.
Ссылки на файлы вывода: blast16.out, blast23.out.