Метаболизм лактозы у прокариот регулируется ее концентрацией в среде. Выделяют четыре случая:
-В среде много глюкозы и нет лактозы
-В среде есть и глюкоза, и лактоза
-В среде есть только лактоза
-В среде нет ни того, ни другого
Транскрипция генов лактозного метаболизма определяется лактозным опероном. Оперон состоит из промотора, оператора и терминатора, а также трех генов:
lacZ кодирует фермент β-галактозидазу, которая расщепляет дисахарид лактозу на глюкозу и галактозу
lacY кодирует β-галактозидпермеазу, мембранный транспортный белок, который переносит лактозу внутрь клетки
lacA кодирует β-галактозидтрансацетилазу, фермент, переносящий ацетильную группу от ацетил-КoA на бета-галактозиды
Первыми работу лактозного оперона описали ученые Ф. Жакобом и Ж. Моно [1].
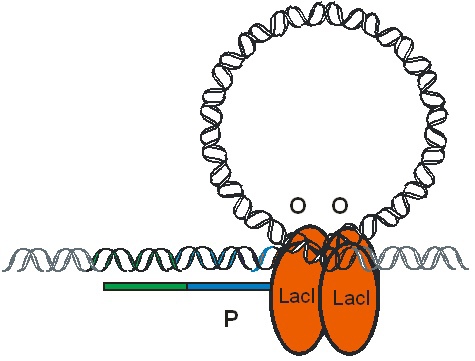
Непосредственно регуляция осуществляется с помощью Lac-репрессора, который связывается с оператором и тормозит транскрипцию, если в среде есть глюкоза, и трансактиватором CRP, который при появлении в клетке цАМФ (признак окислительного стресса) связывается с промотором и активирует транскрипцию. На схемах ниже можно увидеть, как взаимодействуют белки с промотором в описанных выше ситуациях:

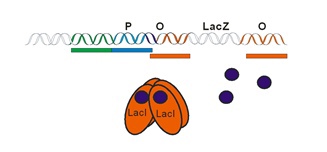
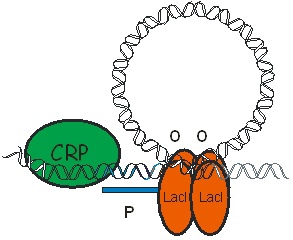
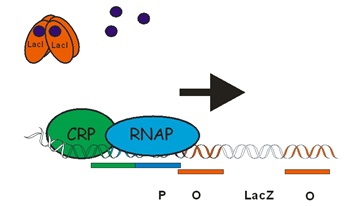
[1] - Jacob F; Monod J. Genetic regulatory mechanisms in the synthesis of proteins PMID 13718526
Рисунки были взяты из презентаций курса молекулярной биологии Королева С.П.
У эукариот в геноме есть окрестность старт кодона AТG, также называемая последовательностью Козак. Эта последовательность включает 4-6 нуклеотидов, предшествующих старт-кодону, и один-два нуклеотида непосредственно после старт-кодона. У млекопитающих оптимальный контекст имеет вид GCCRCCAUGG ( Kozak M. At least six nucleotides preceding the AUG initiator codon enhance translation in mammalian cells).
Обработка данных была выполнена с помощью программы на python: Ссылка.
На вход программа принимает таблицу, содержащую гены человека и их позиции в геноме, в формате .tsv.
В результате работы получаются следующие файлы:
- Файлы для построения весовой позиционной матрицы с последовательностями для обучения (kozak-learn.fasta), тестирования (kozak-test.fasta) и негативного контроля (pseudokozak1.fasta)
- Позиционная весовая матрица (result.csv)
- Гистограммы с распределением весов (hist.svg и hist.png)
- Результаты проверки (check.csv)
- Матрица инфомационного содержания (ic.csv)
Файлы kozak-learn.fasta, kozak-test.fasta и pseudokozak1.fasta содержат по 500 последовательностей, на основе которых была построена следующая позиционная весовая матрица (Таблица 1). При вычислении логарифма отношения использовались pseudocounts, равные 0.1 для всех букв. Это было сделано во избежание получения нулевых частот.
| letter | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A | -0.45 | -0.46 | -0.46 | -0.25 | 0.29 | -0.04 | -0.38 | 1.22 | -6.60 | -6.60 | -0.32 | -0.15 | -0.67 |
| T | -0.34 | -0.49 | -0.40 | -0.57 | -1.16 | -0.79 | -0.95 | -6.60 | 1.22 | -6.60 | -0.69 | -0.50 | -0.35 |
| G | 0.28 | 0.63 | 0.39 | 0.28 | 0.63 | 0.14 | 0.38 | -6.24 | -6.24 | 1.58 | 0.88 | -0.14 | 0.60 |
| C | 0.48 | 0.20 | 0.42 | 0.48 | -0.45 | 0.52 | 0.63 | -6.24 | -6.24 | -6.24 | -0.34 | 0.64 | 0.28 |
Таблица 1: Позиционная весовая матрица
С помощью приведенного выше скрипта также была построена гистограмма на основе вычисленных весов последовательностей (Рис.5).
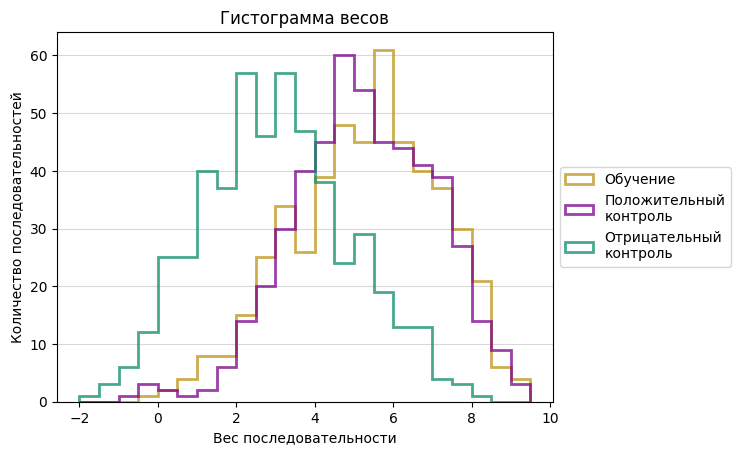
Из гистограммы можно заметить, что порог веса находится около 4, так как при весе выше 4 находка считается удачной. Гистограмма отрицательного контроля сдвинута влево относительно графиков распределения для последовательностей обучения и тестирования практически на четыре деления (то есть довольно заметно).
На основе этих данных была построена таблица результатов проверки (Таблица 2) с порогом 4.
| Обучение | Положительный контроль | Отрицательный контроль | |
|---|---|---|---|
| Сигнал(+) | 290 (58.0%) | 276 (55.2%) | 82 (16.4%) |
| Сигнал(-) | 210 (42.0%) | 224 (44.8%) | 418 (83.6%) |
Таблица 2: Таблица с результатами проверки находок
Из таблицы видно, что вероятность ошибочно принять отрицательный результат за положительный довольно значительна - 44.8%, при этом с вероятностью 16.4% положительный сигнал будет принят за отрицательный . А значит почти в 30% случаев находка будет оценена неверно. Это объясняется большой областью перекрытия негативного и положительного контроля на гистограмме.
Также с помощью приведенного выше кода была построена матрица информационного содержания (Таблица 3).
| letter | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A | -0.12 | -0.12 | -0.12 | -0.08 | 0.16 | -0.02 | -0.11 | 1.76 | 0.00 | 0.00 | -0.10 | -0.05 | -0.15 |
| T | -0.10 | -0.13 | -0.11 | -0.14 | -0.15 | -0.15 | -0.16 | 0.00 | 1.76 | 0.00 | -0.15 | -0.13 | -0.10 |
| G | 0.11 | 0.35 | 0.17 | 0.11 | 0.35 | 0.05 | 0.16 | 0.00 | 0.00 | 2.29 | 0.62 | -0.04 | 0.32 |
| C | 0.23 | 0.07 | 0.19 | 0.23 | -0.09 | 0.26 | 0.35 | 0.00 | 0.00 | 0.00 | -0.07 | 0.36 | 0.11 |
| IC(j) | 0.11 | 0.17 | 0.12 | 0.12 | 0.27 | 0.14 | 0.25 | 1.76 | 1.76 | 2.29 | 0.30 | 0.14 | 0.18 |
Таблица 3: Матрица информационного содержания
С помощью сервиса WebLOGO по файлу для обучения (kozak-learn.fasta) было построено LOGO (Рис. 6)
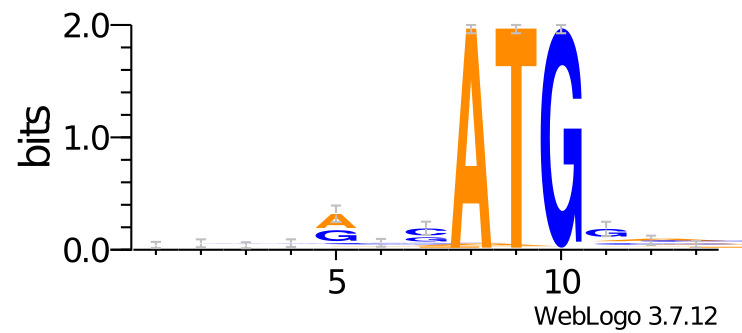
Помимо высоко консервативного участка 8-10, заметно преобладание пуринов на позициях 5, 7 и 11.
Для анализа был выбран референсный геном бактерии Escherichia coli O157:H7 str. Sakai DNA (NC_002695.2). Геном содержит 5,329 генов с GC-составом 50,5%.
С помощью программы на python, были получены следующие данные:
| GC-совтав генома | 0.5053704794221342 |
|---|---|
| Вероятность буквы A на i-той позиции | 0.2473147602889329 |
| Вероятность буквы T на i-той позиции | 0.2473147602889329 |
| Вероятность буквы G на i-той позиции | 0.2526852397110671 |
| Вероятность буквы C на i-той позиции | 0.2526852397110671 |
| Вероятность GAATTC на позициях i..i+5 | 0.00023886905692885308 |
| Ожидаемое число GAATTC в геноме | 1313.4387080953975 |
| Реальное число GAATTC в геноме | 787 |
| p-value | 9.219809820757712e-56 |
Таблица 4: Информация о геноме нашей бактерии
Из таблицы мы видим, что реальное количество сайтов рестрикции GAATTC в геноме бактерии практически в два раза ниже ожидаемого.
В работе были использованы программы Муравьева Г. (Ссылка на страницу).