Для анализа было выбрано семейство белков теплового шока - HSP70 (PF00012). Это группа АТФ-зависимых шаперонов, облегчающих сворачивание сложных белков и стабилизирующих частично свернутые белки, а также способствующих их транспорту через мембрану. Все белки этой группы состоят из следующих структур: N-конец представляет собой домен АТФазы, а С-конец представляет собой область связывания субстрата.
HSP помогают клетке переживать физиологические стрессы, в том числе тепловые. При стрессе резко повышается экспрессия генов этих белков, при чем фактор теплового шока (HSF) вызывает гиперэкспрессию. Белки теплового шока разделяют по размеру на три группы: Hsp60, Hsp70 и Hsp90. Выбранная группа Hsp70 содержит белки размером 70кДа. Самые изученные из них - HSP70, Bip, и DnaK.
Белки Hsp70 обнаружены в клетках практически всех живых организмов,в том числе - у бактерий и человека.
Поиск в базе Pfam велся по ключевому слову - AMP-binding protein. Выравнивание seed содержит 27 последовательностей.
В качестве мотива была выбрана белковая последовательность со следующим паттерном: N[DE][PTV]TA[AV][AGS].[AS]YG. Она встречается в 26 из 27 последовательностей, при чем в каждой только один раз. На уровне совпадения 100% был найден наиболее консервативный участок - [175; 185]. На рисунке ниже (Рис. 1) видно, что даже на уровне совпадения 85% участок остается консервативным практически для всех последовательности.
В результате поиска через сервис MyHints в базе данных Swiss-Prot было найдено 1045 белков, содержащих данных домен, из которых 866 принадлежат к группе Heat shock protein, а еще 179 находок относится к группе гомологов шаперона HscA. Из этого можно сделать вывод, что выбранный мотив отвечает за связывание с белками, чью структуру меняет шаперон.
Алгоритмом NJ было построено филогенетическое дерево (Рис. 2).
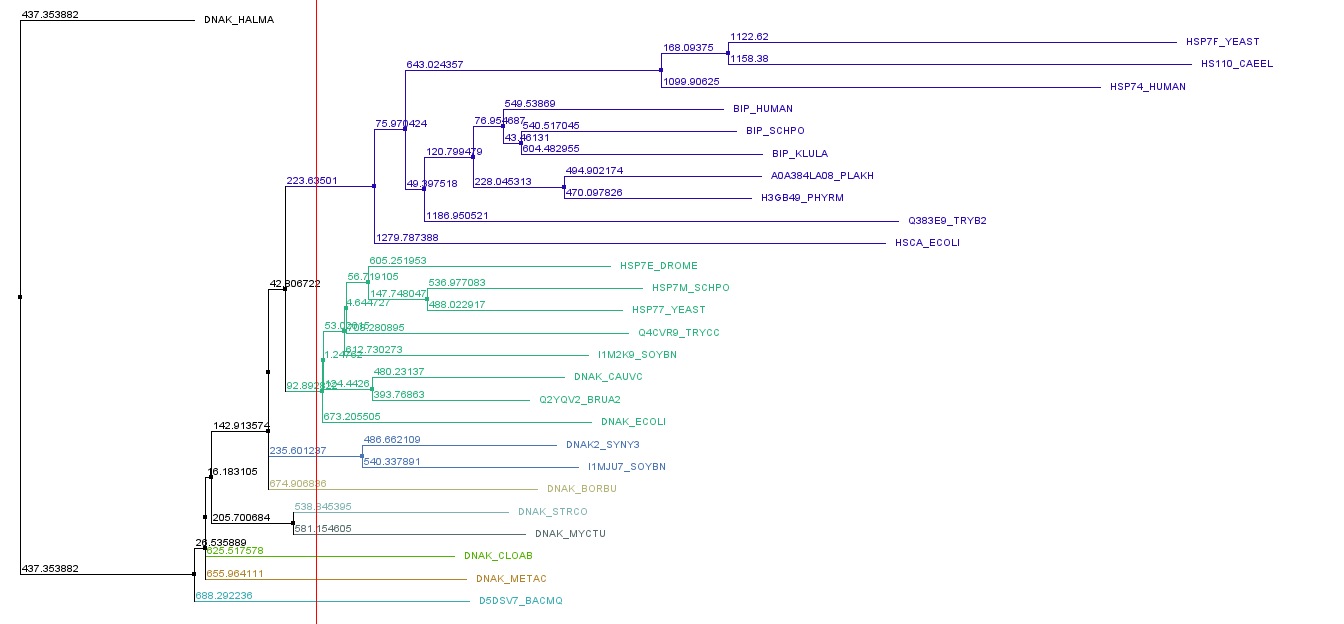
Была взята зеленая клада, содержащая 8 последовательностей. В ней был выбран высоко консервативный участок [95; 100] с паттерном [NT]GDAW[VL]. В других кладах такой паттерн не встретился, а в шести последовательностях на участке [95;100] только гэпы.
В остальном все белки семейства имеют довольно схожую последовательность, так что данный паттерн можно считать специфичным для клады.
Для работы с PSI-BLAST был взят белок с АС Q7VDL2.1 - Probable septum site-determining protein MinC. Он был выделен из цианобактерии Prochlorococcus marinus.
После четвертой итерации число последовательностей выше порога не менялось. Полученные данные представлены в таблице ниже (Таблица 1).
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
|---|---|---|---|---|---|
| 1 | 146 | Q9AG20.1 | 0.005 | A8GFG7.1 | 0.006 |
| 2 | 188 | B6JKX0.1 | 7e-08 | - | - |
| 3 | 188 | Q9ZM51.1 | 2e-12 | A7H8E6.1 | 0.014 |
| 4 | 189 | A8MHK8.1 | 0.001 | A7H8E6.1 | 0.013 |
| 5 | 189 | A8MHK8.1 | 4e-10 | A7H8E6.1 | 0.009 |
Таблица 1: Результат работы PSI-BLAST
На третьей итерации формируется семейство, у которого худшая находка имеет довольно хорошиц e-value. На следующей стадии добавляется белок A8MHK8.1 с пограничным e-value. Можно было бы сделать вывод, что белок либо посторонний, либо очень дальнеродственный, но уже на следующей итерации его e-value уменьшается практически на семь порядков. Предположительно, такое резкое изменение является артефактом, но тем не менее разница между худшей подходящей находкой и лучшей неподходящей довольно большая, а значит, можно считать выделенное семейство довольно обособленным.
В прошлом практикуме использовался геном бактерии Escherichia coli O157:H7 str. Sakai DNA (NC_002695.2).
С помощью следующего кода были получены содержания А и Т в геноме:
| А | 1361525 | 24,8% |
|---|---|---|
| Т | 1358234 | 24,7% |
Таблица 2: АТ содержание в геноме бактерии
Также был расчитан теоритический TA состав - 336 277,6. С помощью скрипта был вычислен реальный TA состав - 254 399. А значит в реальности количество динуклеотидов ТА в 1,32 раза ниже, чем в теории (примерно 75%).