Занятие 10. Макромолекулярный докинг¶
В этом практикуме пытаемся сделать предсказание о связывании фрагмента антитела (VHH domain) верблюда (camelid.pdb) c панкреатической альфа-амилазой (amylase.pdb) с помощью программы ZDOCK.
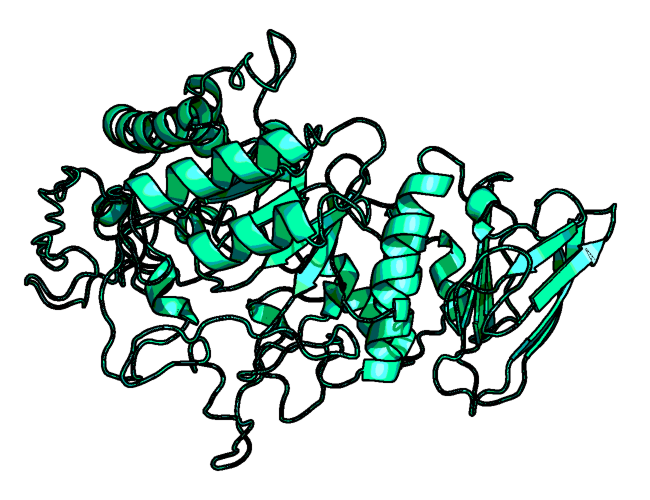 Рисунок 1а. Структура панкреатической альфа-амилазы.
Рисунок 1а. Структура панкреатической альфа-амилазы.
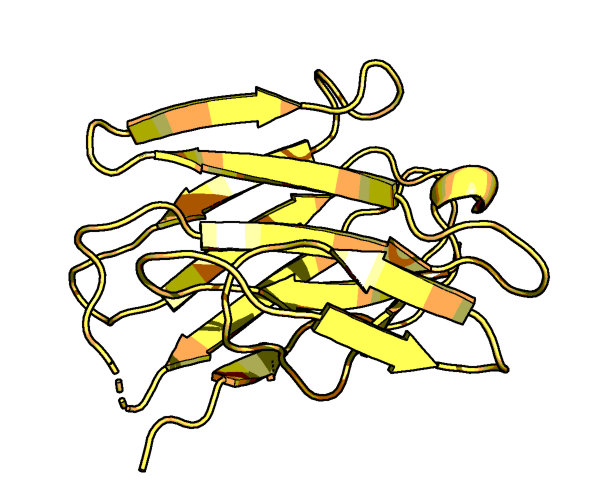 Рисунок 1b. Структура VHH домена антитела верблюда.
Рисунок 1b. Структура VHH домена антитела верблюда.
С помощью pdb2gmx добавляем к исходным pdb-файлам водороды. Затем с помощью mark_sur отмечаем остатки на поверхности белка. mark_sur препроцессирует pdb-файлы перед запуском докинга, добавляя значения заряда, радиуса и типа атома.
%%bash
export LD_LIBRARY_PATH=${LD_LIBRARY_PATH}:/home/preps/golovin/progs/lib
export PATH=${PATH}:/home/preps/golovin/progs/bin
pdb2gmx -f amylase.pdb -o amylase_h.pdb -p -ignh
pdb2gmx -f camelid.pdb -o camelid_h.pdb -p
mark_sur amylase_h.pdb amylase_hm.pdb
mark_sur camelid_h.pdb camelid_hm.pdb
zdock -R amylase_hm.pdb -L camelid_hm.pdb
%%bash
zrank zdock.out.cp 1 2000
sort -n -k2 zdock.out.cp.zr.out | head
Топ-5 структур представлены на рисунке 2.
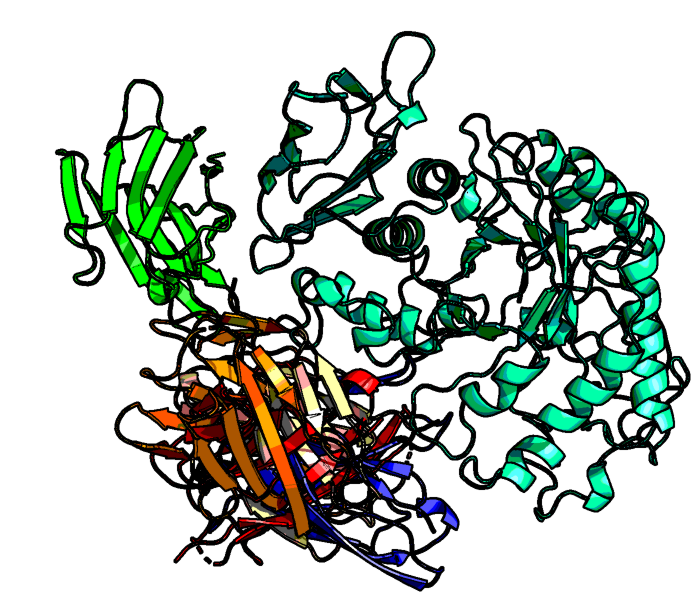 Рисунок 2. 5 лучших структур комплекса амилазы и антитела.
Рисунок 2. 5 лучших структур комплекса амилазы и антитела.
Для анализа качества полученных структур сравним их с известной структурой комплекса из PDB.
%%capture
# Camelid VHH Domains in Complex with Porcine Pancreatic alpha-Amylase
!wget http://www.pdb.org/pdb/files/1kxt.pdb
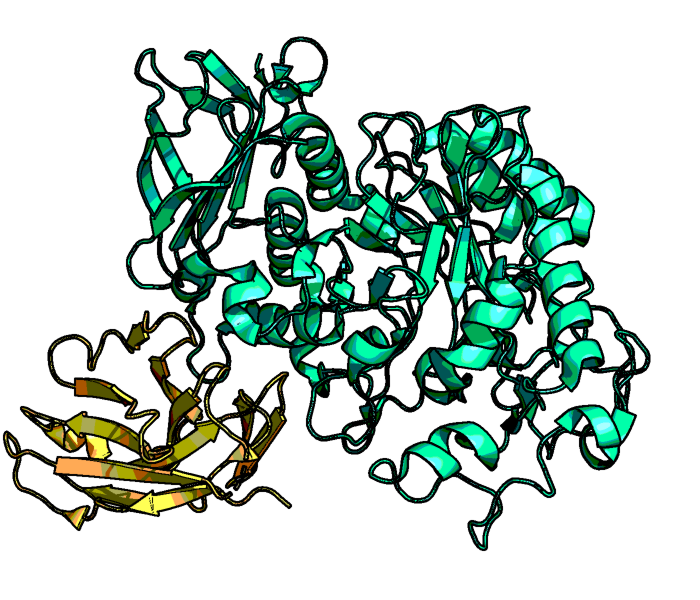 Рисунок 3. Стурктура 1KXT комплекса VHH домена (желтый) и альфа-амилазы (зеленый) из PDB.
Рисунок 3. Стурктура 1KXT комплекса VHH домена (желтый) и альфа-амилазы (зеленый) из PDB.
Видно, что антитело связывается в разных местах и в разных положениях с амилазой (рисунок 2). В целом, место связывания определено верно в 4 структурах из 5 (рисунок 3).
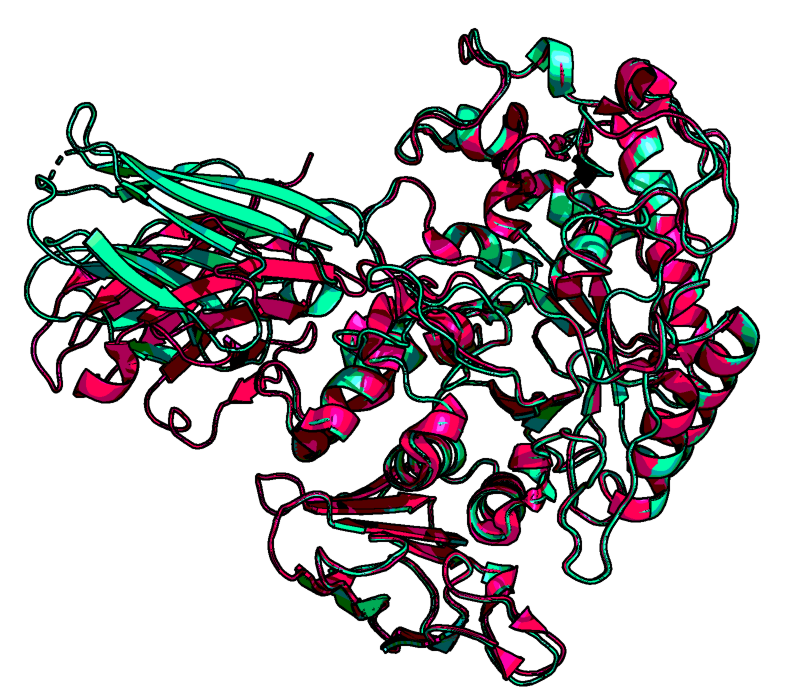 Рисунок 4. Наложение структуры 1KXT (розовый) на лучшую из полученных структур (№1560, голубой).
Рисунок 4. Наложение структуры 1KXT (розовый) на лучшую из полученных структур (№1560, голубой).
Структура амилазы в обоих комплексах практически совпадает. Антитело связывается в том же сайте, но в несколько другом положении.
Теперь оценим качество полученных структур по их сходству с известным комплексом (по RMSD между C$\alpha$-атомами). RMSD посчитаем с помощью g_rms.
for i in range(1,2001):
!grep CA complex.{i}.pdb > {i}.pdb
!echo 0 0 | g_rms -s orig.pdb -f {i}.pdb -o {i}.xvg
!tail -n 1 {i}.xvg >> rmsd
!rm {i}.*
!rm complex.{i}.pdb
!head rmsd.txt
#файл orig.pdb - CA атомы цепей A и B из 1kxt.pdb
#rmsd.txt - файл rmsd с номерами структур (1-2000), отсортированный по увеличению rmsd (в нм)
Наименьшим RMSD оказалось для комплекса №2.
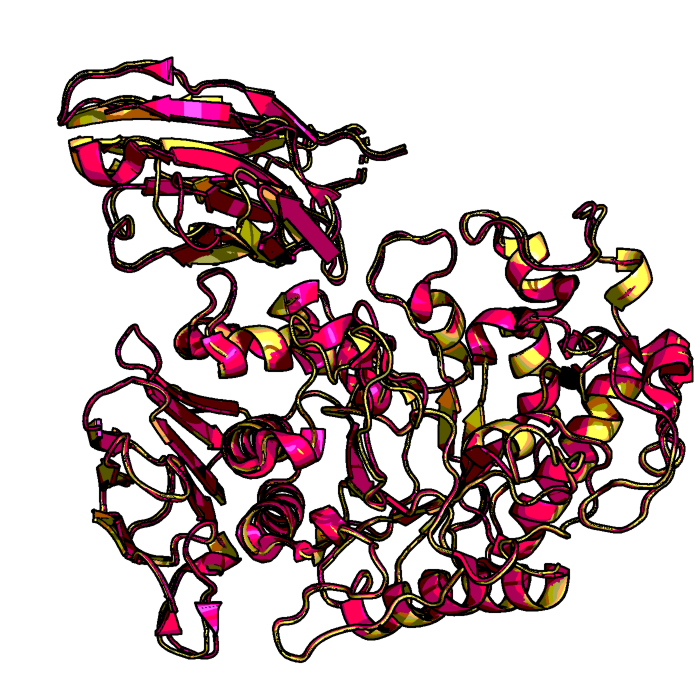 Рисунок 5. Наложение структуры 1KXT (розовый) и комплекса №2 с наименьшим RMSD = 1.45 ангстрем (желтый).
Рисунок 5. Наложение структуры 1KXT (розовый) и комплекса №2 с наименьшим RMSD = 1.45 ангстрем (желтый).