Алгоритмы множественного выравнивания, эволюционные домены белков
1. Описание информации, доступной в Pfam по одному домену
| Название | ID | AC | Число последовательностей (full) | Число последовательностей в выравнивании seed | Число доменных архитектур | Число 3D структур доменов из разных последовательностей |
|---|---|---|---|---|---|---|
| Ankyrin repeat | Ank | PF00023 | 12100 | 1063 | 2689 | 28 |
Анкириновые повторы – один из наиболее распространенных доменов, встречающийся чаще всего в эукариотических белках (у эукариот 10085 белков с этим доменов, у бактерий – 799, у вирусов – 95). Эти повторы были обнаружены в белках с разнообразными функциями: они встречаются в инициаторах транскрипции, регуляторах клеточного цикла, ионных каналах и сигнальных белках. Домены, часто встречающиеся с данным, – Ion_trans, PGG, Ank_2, Ank_4. Дата создания HMM профиля выравнивания – 5 августа 2018 г., воскресенье, 03:11:49, число позиций – 32.
2.Анализ выравнивания из Pfam
В Sunburst был выбран род Streptomyces, содержащий 25 видов и 30 последовательностей. Эти последовательности были выровнены в Jalview, выравнивание отредактировано там же. Консервативный вертикальный блок (21-26 – LLAAGA или LVAAGA), консервативный блок, включающий не все последовательности (30-32 – AQD), и блок, в котором нет оснований предполагать гомологичность фрагментов (16-17), были найдены при помощи Genedoc (Рисунок 1).
Ссылка на проект Jalview
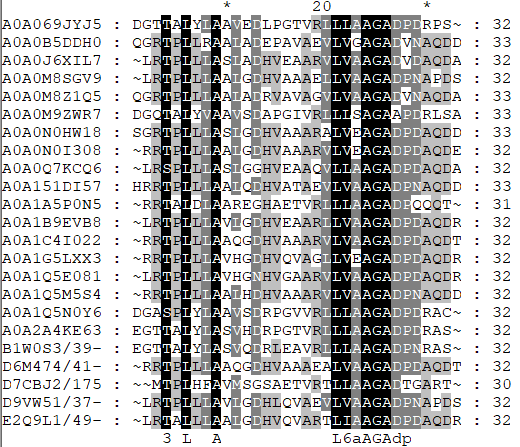 Рисунок 1. Выравнивание, раскрашенное в Genedoc.
Рисунок 1. Выравнивание, раскрашенное в Genedoc.
3. Поиск всех белков с данным доменом Pfam
По запросу database:(type:pfam pf00023) в UniProt было обнаружено 49942 записей, 194 из которых в Swiss-Prot. Число белков с доменной архитектурой PF00023;PF12796; – 22689. У эукариот 41356 белков с данным доменом, у бактерий – 7253, у вирусов – 1055, у архей – 127. Это не согласуется с данными Pfam. Выбранному домену Pfam соответствует домен PROSITE PS50297.
Ссылка на таблицу
