| На главную |
|
Мои проекты | Ссылки | Обо мне | Мои заметки |
Множественное выравнивание
Создание репрезентативной выборки гомологов MntR Bacillus Subtilis
Сначала был определен филум, родной для изучаемого белка. Это Firmicutes. Для поиска гомологов с помощью программы BLAST использовалось ограничение по таксонам в программе BLAST - не филум Firmicutes и не домен Eukaryota. По причине выдачи в числе результатов нескольких некорректных (принадлежащих к "родному" филуму белка), они удалялись вручную. Параметры запросов представлены в таблице 1. FASTA-файл, содержащий последовательности гомологов, можно посмотреть здесь . Затем был осуществлен поиск гомологов белка у эукариот.
Таблица 1. Параметры поиска гомологов| Поиск | Алгоритм BLAST | Название базы данных | Ограничения по таксонам | Порог E-value | Максимальное количество хитов |
| По прокариотам | BLASTP | Reference proteins (refseq_protein) | не Firmicutes не Eukaryota |
1е-05 | 5000 |
| По эукариотам | BLASTP | Reference proteins (refseq_protein) | только Eukaryota | 1 | 2 |
Для эукариот находится только 2 хита, с E-value 0,46 и 0,79, но и они являются всего лишь двумя записями одного и того же белка. Поэтому, в дальнейшем, я не рассмотривала их в качестве гомологов изучаемого белка.
Ген этого белка локализуется в ядре.
Для 500 хитов было построено дерево (рисунок 1), из которого и была сделана выборка гомологов. Их соотношение по таксонам представлено в таблице 2. Всего отобрано 26 гомологов.
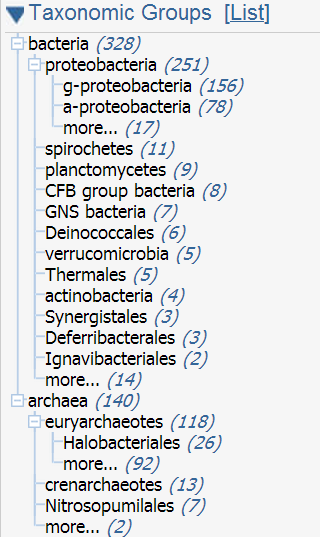
Таблица 2. Распределение отобранных гомологов по таксонам. Светло-голубым цветом отмечено положение узучаемого белка MntR Bacillus subtilis.
| Домен | Филум/Царство | Название организма | Количество белков |
| Archaea | Cenarchaeales | Cenarchaeum symbiosum A | 1 |
| Crenarchaeotes | Hyperthermus butylicus DSM 5456 | 1 | |
| Euryarchaeotes | Natrialba taiwanensis | 1 | |
| Halorubrum tebenquichense | 1 | ||
| Methanocaldococcus villosus | 1 | ||
| Methanoplanus limicola | 1 | ||
| Thermococcus litoralis | 1 | ||
| Nitrosopumilales | Candidatus Nitrosopumilus sp. AR2 | 1 | |
| Nitrososphaerales | Candidatus Nitrososphaera gargensis Ga9.2 | 1 | |
| Bacteria | CFB group bacteria | Polaribacter irgensii | 1 |
| Deferribacterales | Flexistipes sinusarabici DSM 4947 | 1 | |
| Deinococcales | Deinococcus peraridilitoris DSM 19664 | 1 | |
| Firmicutes | Bacillus subtilis | 1 | |
| GNS bacteria | Oscillochloris trichoides | 1 | |
| Ignavibacteriales | Melioribacter roseus P3M | 1 | |
| Planctomycetes | Planctomyces maris | 1 | |
| Proteobacteria | Salinisphaera shabanensis | 1 | |
| Yokenella regensburgei | 1 | ||
| Acetobacter tropicalis | 1 | ||
| delta proteobacterium NaphS2 | 1 | ||
| Mesorhizobium sp. STM 4661 | 1 | ||
| Spirochetes | Treponema vincentii | 1 | |
| Treponema phagedenis | 1 | ||
| Synergistales | Anaerobaculum mobile DSM 13181 | 1 | |
| Thermales | Thermus scotoductus SA-01 | 1 | |
| Thermotogales | Mesotoga sp. PhosAc3 | 1 | |
| Verrucomicrobia | Coraliomargarita akajimensis DSM 45221 | 1 |
Множественное выравнивание гомологов белка MntR Bacillus subtilis
Последовательности всех отобранных гомологов и исходного белка были выровнены в программе Jalview с помощью Muscle. Полная картина выравнивая представлена на рисунке 2.
Рисунок 2. Множественное выравнивание для белка MntR Bacillus subtilis и его отобранных гомологов из разных таксонов На этом рисунке и далее: фиолетовый цвет - гэпы, красный - отрицательно заряженные аминокислотные остатки(АК), зеленый - положительно заряженные АК, синий - полярные незаряженные АК, желтый - неполярные незаряженные АК. Строки аннотации в порядке снизу вверх: Secondary - особенности вторичной структуры белка (альфа-спирали (красные), бета-листы (зеленые), места поворота цепи), Ligand - отмечены аминокислотные остатки, принимающие участи в связывании лиганда. В данном случае, это Glu11, His77, Glu102. Blocks - отмечены красным цветом участки возможных блоков
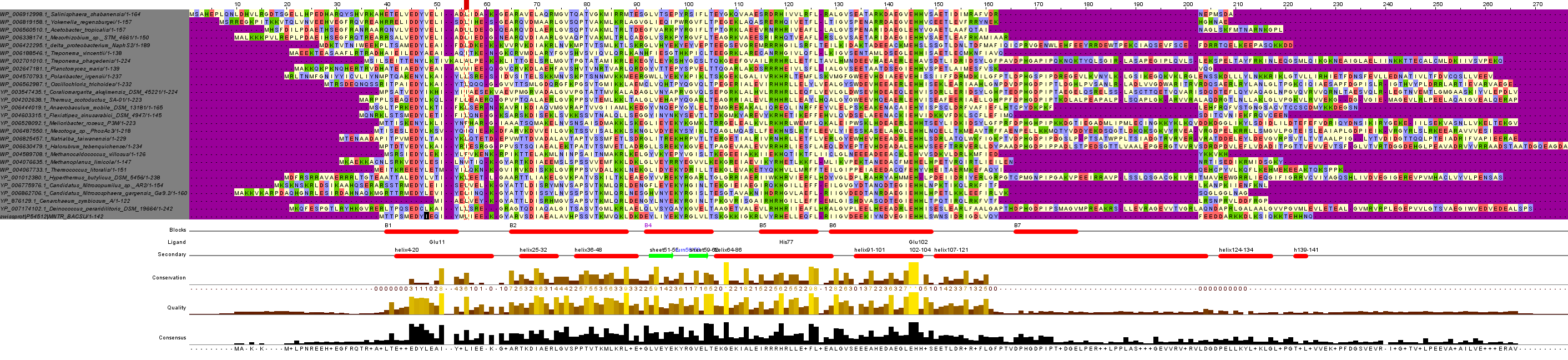
Затем были выделены более насыщенным цветом только те участки, которые имеют консервативность более 25%. Остальные участки остались белыми. Это выравнивание представлено на рисунке 3.
Рисунок 3. Множественное выравнивание для белка MntR Bacillus subtilis и его отобранных гомологов из разных таксонов с учетом консервативности участков.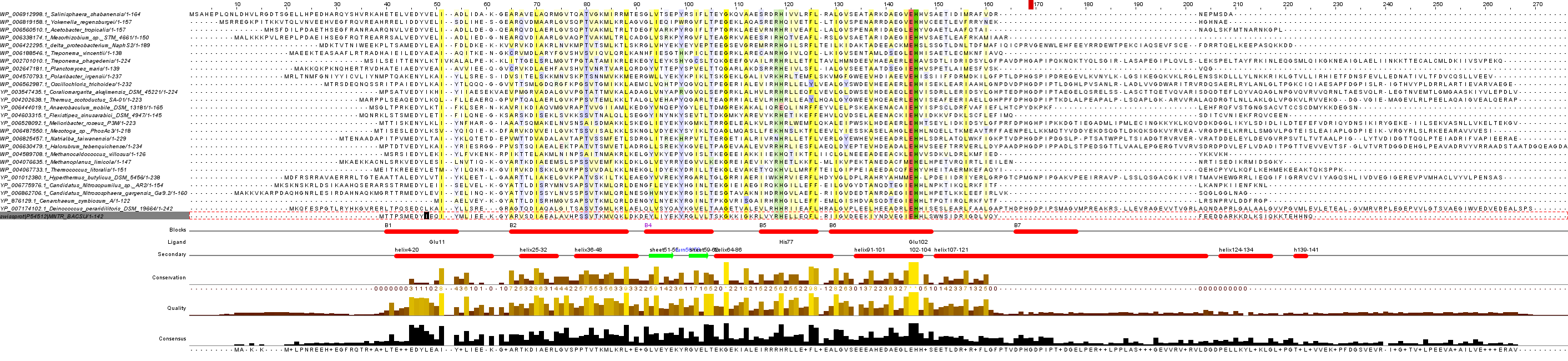
Результаты анализа множественного выравнивания гомологов белка MNTR_BACSU
Выравнивание в целом нельзя оценить как высоко консервативное, возможно, это связано с особенностями выборки, потому что при её составлении, согласно заданию, преследовалась цель
охватить как можно большее разнообразие таксонов в первую очередь, и лишь во вторую учитывалось E-value.
В выравнивании наиболее консервативны отдельные участки альфа-спиралей, очень высокую консерваривность показали участки связывания с лигандом, кроме глутамина11, его консервативность на среднем уровне.
Глутамин102 и гистидин103 - абсолютно (100%) консервативны для всех белков. Видимо, эти остатки играют ключевую роль в связывании лиганда.
Самое плохое выравнивание наблюдается в "петлях" белков большего размера, чем изучаемый (однако, есть участки в петлях крупных белков, которые хорошо выравниваются друг с другом. Участки последовательностей
их в этих местах очень схожи. Можно обратить внимание на петлю на месте альфа-спирали 107-121. В ней часто встрачается последовательность DPHGDPIP.), и на концевых участках белка. Видимо эти участки не несут столь значимой функции - не отвечают за внутрибелковые связи и связь с лигандом.
Колонки гэпов соответствуют чаще неструктурированным участкам или петле больших белков на месте крупной альфа-спирали исходного.
Выделенные блоки либо объединяют пару небольших альфа-спиралей, либо дробят крупную, для расположенных близко бета-листов был выделен один блок. Критерием выбора служило сходство по свойствам остатков и самим отстакам большей части белков из выборки.
Аминокислотные остатки со схожими свойствами условно принимались за одно и то же.
Как уже упоминалось выше консервативность осттатков, связывающих лиганд достаточно высоко. Это иллюстрирует рисунок 4.
Рисунок 4. Консервативность оттатков белка на его трехмерной структуре. Лиганд - ярко-зеленый, насыщенность цвета аминокислотных остатков прямо пропорциональна их консервативности.
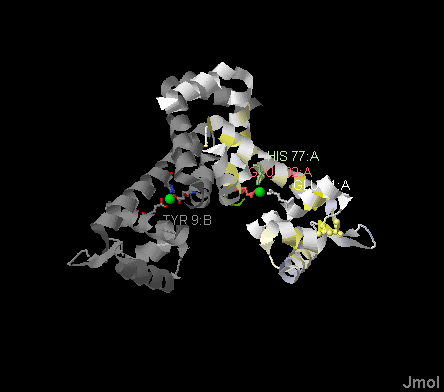
Наименее консервативный глутамин11 часто заменяется на лизин, редко на валин и треонин. На основе выравнивая можно сказать, что некоторые гомологи смогут связать тот же лиганд, благодаря полной идентичности центра связывания.