| На главную |
|
Мои проекты | Ссылки | Обо мне | Мои заметки |
Эволюционные домены. Банки Pfam и InterPro
1. Доменная структура белка CLPQ_BACSU по данным Pfam
Таблица 1. Доменная структура белка CLPQ_BACSU по данным PfamCхема из Pfam: |
|||||
| Пояснения к схеме |
|||||
| № | Pfam AC | Pfam ID | Полное название семейства доменов |
Положение в последовательности белка CLPQ_BACSU | Клан |
| 1. | PF01325 | Fe_dep_repress | Iron dependent repressor (названо по названию железо-зависимого репрессора транскрипции у бактерий и архей) | 1-60 | CL0123 (Helix-turn-helix clan) |
| 2. | PF02742 | Fe_dep_repr_C | Iron dependent repressor (названо по названию железо-зависимого репрессора транскрипции у бактерий и архей) | 63-132 | CL0123 (Helix-turn-helix clan) |
Данные о домене Fe_dep_repress
Домен входит в 14 белковых архитектур. Последовательность известна для 3362 белков, содержащих этот домен.Пространственная структура определена для 6ти из них. Выравние seed для этих белком можно посмотреть здесь. Как можно увидеть по выравниванию, белки содержат гомологичный домен.
Представленность доменов PF01325 и PF02742 в организмах различных таксонов
Работа велась в белками следующей архитектуры:
Таблица 2. Домен PF01325 в организмах различных таксонов
| Таксон | Количество белков с доменом PF00227. | |
| Эукариоты | Зеленые растения | 0 |
| Грибы | 0 | |
| Животные | 0 | |
| Остальные эукариоты | 0 | |
| Археи | 257 | |
| Бактерии | 3099 | |
| Вирусы | 0 | |
Как видно из данной таблицы, изучаемый домен характерен исключельно для прокариот, что может подтверждаться данными поиска гомологов белка с использованием
алгоритма BLAST. Алгоритм не нашел ни одного удовлетворительного гомолога среди эукариот.
Наибольшей встречаемости достигает среди бактерий.
Таблица 3. Домен PF02742 в организмах различных таксонов
| Таксон | Количество белков с доменом PF00227. | |
| Эукариоты | Зеленые растения | 0 |
| Грибы | 0 | |
| Животные | 0 | |
| Остальные эукариоты | 0 | |
| Археи | 252 | |
| Бактерии | 3131 | |
| Вирусы | 0 | |
Для второго домена наблюдаем идентичную ситуацию, что, впрочем, неудивительно, учитывая функциональную близость доменов.
Сравние описания мотивов в разных банках семейств, по данным InterPro.
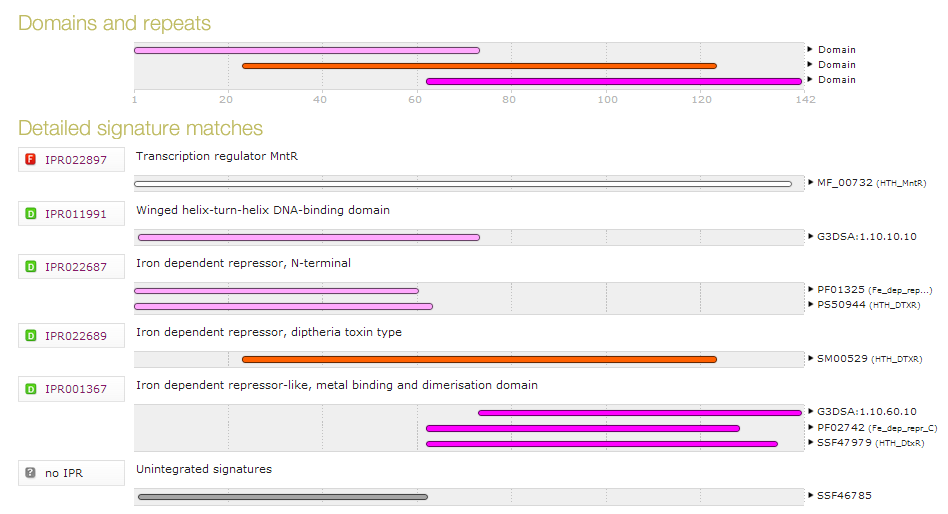
Самый короткий мотив PF01325 (Fe_dep_repress) из банка данных Pfam (60 остатков)
Самый длинный мотив MF_00732 (HTH_MntR) из банка данных HAMAP (140 остаттков)
В InterPro интегрированы домены:
Winged helix-turn-helix DNA-binding domain (G3DSA:1.10.10.10 из банка GENE3D); 2-73 остатки
Iron dependent repressor, N-terminal (PF01325 (Fe_dep_repress) из банка Pfam); 1-60 остатки
HTH_DTXR (PS50944)- его профиль из банка Prosite (HTH_DTXR); 1-63 остатки
Iron dependent repressor, diptheria toxin type - из банка SMART; 24-123 остатки;
Iron dependent repressor-like, metal binding and dimerisation domain из банков данных Gene3D (G3DSA:1.10.60.10), Pfam (PF02742 (Fe_dep_repr_C)), SUPERFAMILY (SSF47979).
Номера остатков соответственно 74-141, 63-128, 63-136.
Границы элементов Pfam чуть уже, чем у структурных элеметнов. Это может объясняться, тем, что конечные аминокислоты доменов могут быть очень неконсеквативны, поэтому не учитываться в банке данных Pfam.