| На главную |
|
Мои проекты | Ссылки | Обо мне | Мои заметки |
A- и В-формы ДНК, структура РНК
Задание 1
Были построены модели структур A-, B- и Z-формы ДНК с помощью инструментов пакета 3DNA (команда fiber). Получены следующие файлы:
А-форма ДНК
B-форма ДНК
Z-форма ДНК
Задание 2
Упр.1. Научиться выделять разные атомы и химические группировки, используя средства JMol.
На примере А-формы ДНК покажем выделение сахарофосфатного остова (рисунок 1), всех нуклеотидов (рисунок 2), 7го атома азота в гуанинах (рисунок), все аденины (рисунок 4).
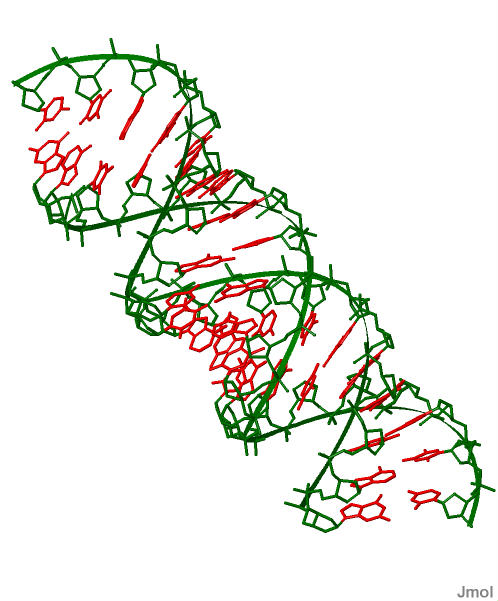 Рисунок 1. A-форма ДНК, зеленым выделен сахарофофатный остов, красный - азотистые основания. |
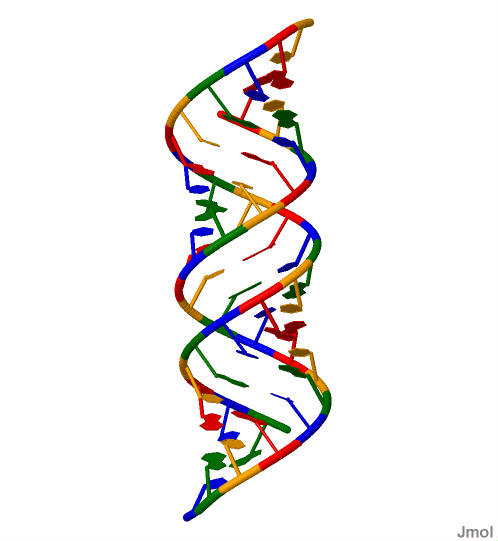 Рисунок 2. А - форма ДНК, цветом выдедены различные нуклеотиды. Красный - аденин, оранжевый - тимин, зеленый - гуанин, синий - цитозин. |
 Рисунок 3. А - форма ДНК. Оранжевый цвет - аденин, красный - все остальные, зеленый - сахаро-фосфатный остов. |
 Рисунок 4.а A-форма ДНК, гуанин выделен синим цветом, его седьмой атом азота - желтым. |
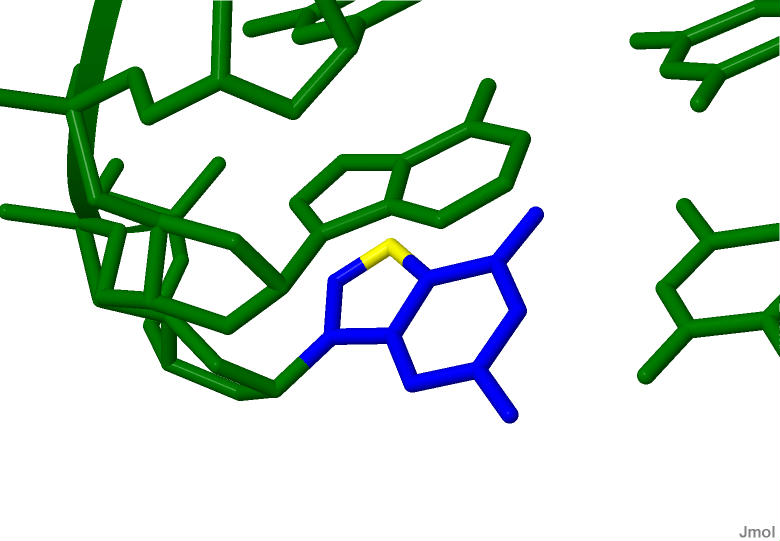 Рисунок 4.b A-форма ДНК, гуанин выделен синим цветом, его седьмой атом азота - желтым. Увеличенный масштаб. |
 Рисунок 5. Отдельно показанная цепь РНК. Видно, что разрывов в структуре нет. |
 Рисунок 6. Отдельно показанная цепь ДНК, разрывов в структуре нет. |
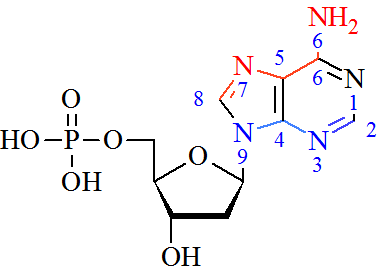 Рисунок 7. Адениновый нуклеотид, на котором красным цветом отмечены атомы, ображенные в сторону большой борозки, синим цветом отмечены атомы, обращенные в сторону малой бороздки. |
Упр.2. Сравнить параметры большой и малой бороздок для различных форм ДНК.
Результаты измерения представлены в таблице 1. На рисунках 8-13 представлены соответсвенно для А, В и Z-форм проводимые изверения бороздок.
| A-форма | B-форма | Z-форма | |
| Тип спирали (правая или левая) | Правая | Правая | Левая |
| Шаг спирали (Å) | 28,03 | 33,75 | 36,35 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 16,81 | 17,21 | 7,2 |
| Ширина малой бороздки | 7,98 | 11,69 | 15,17 |
 Рисунок 8. Измерение шага спирали A-формы |
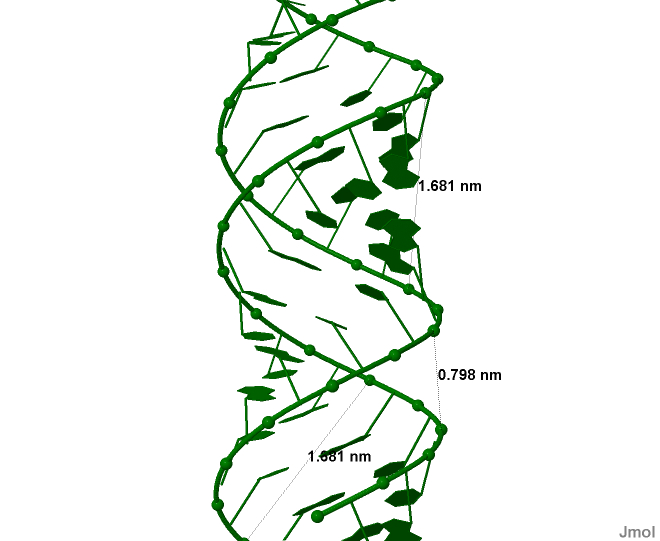 Рисунок 9. Измерение ширины большой и малой бороздок A-формы Рисунок 9. Измерение ширины большой и малой бороздок A-формы |
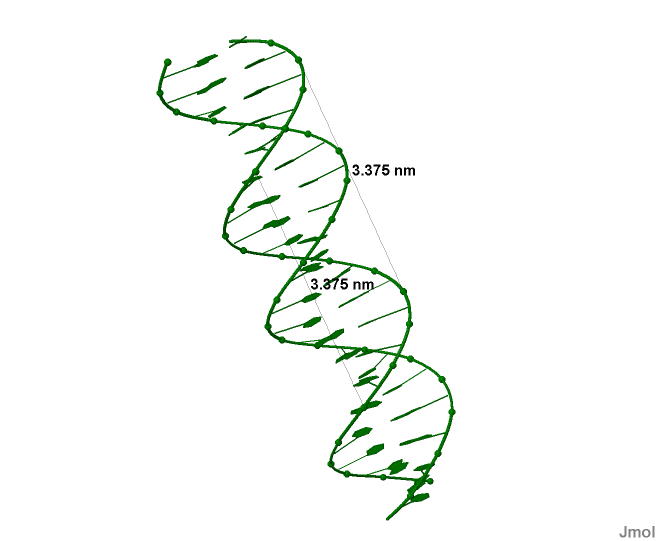 Рисунок 10. Измерение шага спирали В-формы |
 Рисунок 11. Измерение ширины большой и малой бороздок В-формы Рисунок 11. Измерение ширины большой и малой бороздок В-формы |
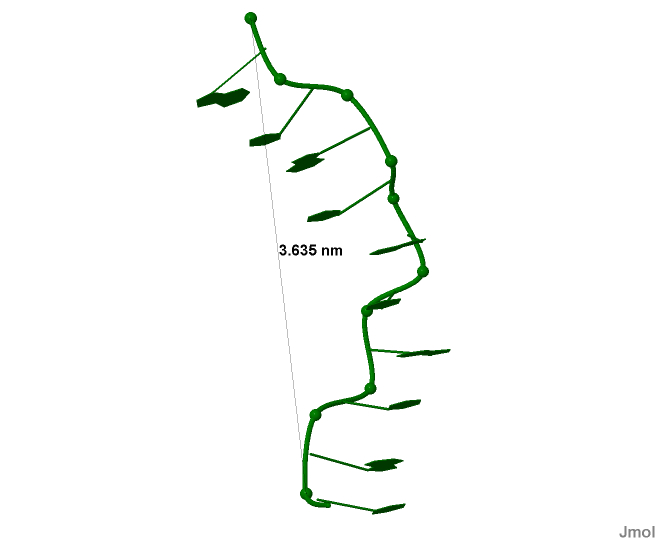 Рисунок 12. Измерение шага спирали Z-формы Рисунок 12. Измерение шага спирали Z-формы |
 Рисунок 13. Измерение ширины большой и малой бороздок Z-формы Рисунок 13. Измерение ширины большой и малой бороздок Z-формы |
Упр. 3. Сравнить торсионные углы в структурах А- и В-форм.
Приведем данные, полученные с помощью JMol и данные из презентации в одной таблице 2.
| угол | α | β | γ | δ | ε | ξ | χ |
| A-форма ( из презетации) | 62 | 173 | 52 | 88 или 3 | 178 | -50 | -160 |
| A-форма (Jmol) | 64.1 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157,2 |
| B-форма ( из презентации) | 63 | 171 | 54 | 123 или 131 | 155 | -90 | -117 |
| B-форма (Jmol) | 85.9 | 136.3 | 31.2 | 143.3 | -140.8 | -160.5 | -98.0 |
Задание 4: Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA
Упр. 1. Определение торсионеых углов нуклеотидов
С помощью программ пакета 3DNA find_pair и analyze определяли торсионные углы для заданной структуры ДНК из PDB файла 1odh
Таблица 3. Торсионные углы в заданной структуре ДНК, нижняя строчка - среднее значение торсионных углов без учета краевых нуклеотидов.
base alpha beta gamma delta epsilon zeta chi 1 C --- --- 11.5 125.3 --- --- -77.4 2 A --- 180.0 90.0 180.0 --- --- -90.0 3 G --- 160.5 -30.0 180.0 --- --- -101.5 4 G --- 0.0 -0.0 168.5 -100.9 150.0 -150.0 5 G -90.0 164.2 41.8 161.6 --- --- -153.4 6 T --- -160.5 30.0 161.6 --- --- -143.3 7 C --- 180.0 -90.0 135.0 125.3 -30.0 0.0 8 A 180.0 60.0 180.0 180.0 --- --- 180.0 9 T --- --- -90.0 135.0 144.7 -150.0 0.0 10 G 19.1 -143.3 -54.7 -0.0 0.0 -54.7 -90.0 11 C 0.0 154.8 43.3 146.1 153.4 180.0 0.0 12 A 25.2 121.5 54.7 90.0 --- --- -131.8 13 C --- 180.0 0.0 180.0 180.0 -135.0 -131.8 14 C -0.0 -180.0 0.0 135.0 --- --- -144.7 15 C --- -125.3 0.0 180.0 180.0 -116.6 0.0 16 A -39.2 144.7 45.0 135.0 -160.5 -138.2 -0.0 17 T -26.6 180.0 54.7 144.7 161.6 -138.2 -90.0 18 C -54.7 -160.5 30.0 180.0 --- --- -90.0 Среднее 0,30 61,11 17,18 144,53 75,96 -48,08 -65,41
Нуклеотиды с самыми отклоняющимися от среднего значениями торсиооных углов - 2й аденин и 7й цитозин.
Упр. 2. Определение структуры водородных связей.
Для вторичной структуры заданной тРНК характерны следующие особенности:
1. Антикодоновый стебель: [938-944] - [932-926]
2. Акцепторный стебель: [901-907] - [972-966]
3. Т-стебель: [949-953] - [965-961]
4. D-стебель: [910-913] - [925-922]
Водородные связи в молекуле:
(----- for WC bp, + for isolated bp, x for helix change)
Strand I Strand II Helix
1 (0.003) C:.901_:[..U]U-----A[..A]:.972_:C (0.005) |
2 (0.004) C:.902_:[..C]C-----G[..G]:.971_:C (0.009) |
3 (0.004) C:.903_:[..C]C-----G[..G]:.970_:C (0.011) |
4 (0.012) C:.904_:[..G]G-----C[..C]:.969_:C (0.004) |
5 (0.008) C:.905_:[..U]U-*---G[..G]:.968_:C (0.010) |
6 (0.006) C:.906_:[..G]G-----C[..C]:.967_:C (0.008) |
7 (0.006) C:.907_:[..A]Ax----U[..U]:.966_:C (0.004) |
8 (0.017) C:.949_:[5MC]c-----G[..G]:.965_:C (0.008) |
9 (0.005) C:.950_:[..G]G-----C[..C]:.964_:C (0.005) |
10 (0.006) C:.951_:[..G]G-----C[..C]:.963_:C (0.006) |
11 (0.012) C:.952_:[..G]G-----C[..C]:.962_:C (0.005) |
12 (0.009) C:.953_:[..G]G----xC[..C]:.961_:C (0.008) |
13 (0.012) C:.954_:[5MU]u-**-xA[..A]:.958_:C (0.007) |
14 (0.043) C:.955_:[PSU]Px**+xG[..G]:.917_:C (0.005) x
15 (0.005) C:.938_:[..C]C-*---P[PSU]:.932_:C (0.044) |
16 (0.006) C:.939_:[..G]G-----C[..C]:.931_:C (0.006) |
17 (0.003) C:.940_:[..U]U-*---G[..G]:.930_:C (0.009) |
18 (0.010) C:.941_:[..G]G-----C[..C]:.929_:C (0.004) |
19 (0.010) C:.942_:[..C]C-----G[..G]:.928_:C (0.004) |
20 (0.003) C:.943_:[..C]C-----G[..G]:.927_:C (0.005) |
21 (0.005) C:.944_:[..A]Ax*---G[..G]:.926_:C (0.005) |
22 (0.009) C:.910_:[..G]G-*---U[..U]:.925_:C (0.004) |
23 (0.003) C:.911_:[..U]U-----A[..A]:.924_:C (0.009) |
24 (0.007) C:.912_:[..U]U-----A[..A]:.923_:C (0.007) |
25 (0.044) C:.913_:[PSU]Px*--xG[..G]:.922_:C (0.005) |
26 (0.003) C:.908_:[..U]Ux**-xA[..A]:.946_:C (0.005) |
27 (0.003) C:.914_:[..A]A-*--xA[..A]:.921_:C (0.008) |
28 (0.004) C:.915_:[..A]A-**+xU[..U]:.948_:C (0.003) |
29 (0.115) C:.916_:[H2U]ux**+xU[..U]:.959_:C (0.006) x
30 (0.021) C:.918_:[..G]Gx---xC[..C]:.956_:C (0.007) +
Мы видим несколько неканонических оснований - псевдоуридин, 5-метилцитозин, 2-гидроксиуридин, а так же неканонические взаимодействия между нуклеотидами (они отмечены звездочками).
Нуклеотиды, образующие комплементарные пары вне стеблей:
13 (0.012) C:.954_:[5MU]u-**-xA[..A]:.958_:C (0.007) | 14 (0.043) C:.955_:[PSU]Px**+xG[..G]:.917_:C (0.005) x 27 (0.003) C:.914_:[..A]A-*--xA[..A]:.921_:C (0.008) | 28 (0.004) C:.915_:[..A]A-**+xU[..U]:.948_:C (0.003) | 29 (0.115) C:.916_:[H2U]ux**+xU[..U]:.959_:C (0.006) x 30 (0.021) C:.918_:[..G]Gx---xC[..C]:.956_:C (0.007) +
Упр. 3. Стекинг-взаимодействия.
Площадь перекрывания:
step i1-i2 i1-j2 j1-i2 j1-j2 sum
1 UC/GA 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 2.52( 1.02) 2.52( 1.02)
2 CC/GG 0.00( 0.00) 0.00( 0.00) 0.27( 0.00) 4.35( 3.12) 4.62( 3.12)
3 CG/CG 0.07( 0.00) 0.00( 0.00) 4.34( 1.58) 0.00( 0.00) 4.41( 1.58)
4 GU/GC 6.93( 4.29) 0.00( 0.00) 0.00( 0.00) 6.95( 3.82) 13.88( 8.11)
5 UG/CG 0.04( 0.00) 0.00( 0.00) 4.43( 1.56) 0.00( 0.00) 4.47( 1.56)
6 GA/UC 3.95( 2.44) 0.00( 0.00) 0.00( 0.00) 1.52( 0.32) 5.47( 2.76)
7 Ac/GU 3.57( 0.85) 0.00( 0.00) 0.00( 0.00) 6.18( 3.23) 9.75( 4.09)
8 cG/CG 0.00( 0.00) 0.00( 0.00) 3.86( 1.41) 0.39( 0.01) 4.24( 1.42)
9 GG/CC 2.27( 0.85) 0.00( 0.00) 0.26( 0.00) 1.27( 0.37) 3.80( 1.22)
10 GG/CC 5.13( 3.77) 0.00( 0.00) 0.11( 0.00) 0.38( 0.00) 5.62( 3.77)
11 GG/CC 3.90( 2.52) 0.00( 0.00) 0.65( 0.00) 0.00( 0.00) 4.55( 2.52)
12 Gu/AC 8.16( 3.29) 0.00( 0.00) 0.00( 0.00) 5.49( 2.41) 13.66( 5.70)
13 uP/GA 5.69( 1.65) 0.00( 0.00) 0.00( 0.00) 6.67( 3.61) 12.36( 5.26)
14 PC/PG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
15 CG/CP 0.64( 0.30) 0.00( 0.00) 0.00( 0.00) 3.57( 1.51) 4.21( 1.81)
16 GU/GC 7.38( 3.96) 0.00( 0.00) 0.00( 0.00) 7.11( 4.09) 14.49( 8.06)
17 UG/CG 0.00( 0.00) 0.00( 0.00) 6.00( 3.08) 0.00( 0.00) 6.00( 3.08)
18 GC/GC 5.84( 2.75) 0.00( 0.00) 0.00( 0.00) 5.42( 2.57) 11.26( 5.33)
19 CC/GG 0.05( 0.00) 0.00( 0.00) 0.55( 0.00) 1.95( 0.55) 2.55( 0.55)
20 CA/GG 0.38( 0.00) 0.00( 0.00) 1.45( 0.51) 1.18( 0.18) 3.01( 0.69)
21 AG/UG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.38( 0.14) 0.38( 0.14)
22 GU/AU 6.73( 3.84) 0.00( 0.00) 0.00( 0.00) 5.58( 4.52) 12.31( 8.36)
23 UU/AA 1.31( 0.02) 0.00( 0.00) 0.00( 0.00) 2.24( 1.93) 3.55( 1.96)
24 UP/GA 1.76( 0.24) 0.00( 0.00) 0.00( 0.00) 3.67( 2.38) 5.43( 2.63)
25 PU/AG 2.99( 0.73) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 2.99( 0.73)
26 UA/AA 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 3.31( 2.31) 3.31( 2.31)
27 AA/UA 5.89( 3.47) 0.00( 0.00) 0.00( 0.00) 4.87( 2.39) 10.76( 5.86)
28 Au/UU 5.28( 3.84) 0.00( 0.00) 0.00( 0.00) 4.70( 0.64) 9.98( 4.48)
29 uG/CU 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
30 GC/GC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
31 CC/GG 0.00( 0.00) 0.00( 0.00) 0.27( 0.00) 3.38( 2.02) 3.65( 2.02)
32 CG/CG 0.00( 0.00) 0.00( 0.00) 4.91( 2.11) 0.00( 0.00) 4.91( 2.11)
33 GU/GC 7.25( 4.31) 0.00( 0.00) 0.00( 0.00) 6.58( 3.48) 13.84( 7.79)
34 UG/CG 0.00( 0.00) 0.00( 0.00) 5.98( 3.15) 0.00( 0.00) 5.98( 3.15)
35 GA/UC 3.17( 1.29) 0.00( 0.00) 0.00( 0.00) 1.50( 0.18) 4.66( 1.47)
36 Ac/GU 5.06( 1.54) 0.00( 0.00) 0.00( 0.00) 7.01( 4.34) 12.08( 5.88)
37 cG/CG 0.00( 0.00) 0.00( 0.00) 4.39( 1.52) 0.14( 0.00) 4.53( 1.53)
38 GG/CC 3.13( 1.64) 0.00( 0.00) 0.44( 0.00) 0.48( 0.07) 4.05( 1.71)
39 GG/CC 4.49( 2.86) 0.00( 0.00) 0.53( 0.00) 0.00( 0.00) 5.02( 2.86)
40 GG/CC 3.22( 1.74) 0.00( 0.00) 0.82( 0.00) 0.00( 0.00) 4.04( 1.74)
41 Gu/AC 8.53( 3.99) 0.00( 0.00) 0.00( 0.00) 5.60( 2.62) 14.13( 6.61)
42 uP/GA 6.11( 1.84) 0.00( 0.00) 0.00( 0.00) 7.23( 4.69) 13.34( 6.52)
43 PC/PG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
44 CG/CP 1.12( 0.64) 0.00( 0.00) 0.00( 0.00) 3.64( 1.58) 4.76( 2.22)
45 GU/GC 6.81( 3.55) 0.00( 0.00) 0.00( 0.00) 5.82( 2.76) 12.63( 6.31)
46 UG/CG 0.00( 0.00) 0.00( 0.00) 5.72( 2.85) 0.00( 0.00) 5.72( 2.85)
47 GC/GC 6.67( 3.64) 0.00( 0.00) 0.00( 0.00) 7.00( 3.98) 13.67( 7.62)
48 CC/GG 0.83( 0.04) 0.00( 0.00) 0.12( 0.00) 2.10( 0.67) 3.04( 0.71)
49 CA/GG 0.42( 0.00) 0.00( 0.00) 1.33( 0.60) 1.21( 0.17) 2.95( 0.77)
50 AG/UG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.07( 0.01) 0.07( 0.01)
51 GU/AU 6.65( 3.90) 0.00( 0.00) 0.00( 0.00) 5.12( 4.12) 11.77( 8.02)
52 UU/AA 1.05( 0.01) 0.00( 0.00) 0.00( 0.00) 2.84( 2.43) 3.88( 2.44)
53 UP/GA 1.86( 0.20) 0.00( 0.00) 0.00( 0.00) 2.99( 1.58) 4.85( 1.78)
54 PU/AG 3.09( 0.55) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 3.09( 0.55)
55 UA/AA 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 4.02( 2.90) 4.02( 2.90)
56 AA/UA 6.10( 3.99) 0.00( 0.00) 0.00( 0.00) 6.46( 3.76) 12.56( 7.75)
57 Au/UU 4.65( 2.82) 0.00( 0.00) 0.00( 0.00) 2.76( 0.05) 7.41( 2.86)
58 uG/CU 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
Получим стандартные изображения стекинг-взаимодействий с наибольшей и наимменьшей прощадью перекрывания.
 Рисунок 14. Стекинг-взаимодействия с наибольшим перекрыванием |
 Рисунок 15. Стекинг-взаимодействия с наименьшим перекрыванием |
© Ходыкина Наталья,2012