| На главную |
|
Мои проекты | Ссылки | Обо мне | Мои заметки |
Программы пакета EMBOSS
Программа getorf
С помощью команды entret сделали файл с записью D89965 из банка EMBL.
Затем, командой getorf embl:d89965 -minsize 90 -find 1 получили открытые рамки считывания, удовлетворяющие следующим условиям: длина не менее 90 аминокислот (-minsize 90);
начинаются со старт-кодона и заканчиваются стоп-кодоном (-find 1). Оказалось, что третьья рамка полностью соответсвует последователности белка, указанного в записи d89965.
Теперь обратимся к бакну данных SwissProt, чтобы посмотреть на какую запись в этом банке ссылается EMBL. Оказалось, что в банке SwissProt та же самая последовательность указана, как принадлежащая
E.coli , в то время как по данным EMBL это белок крысы. Чтобы разобраться в ситуации можно вспомнить, что EMBL не реферируемый банк данных, в отличии от SwissProt. Значит,
скорее всего, в определении принадлежности этого белка крысе была ошибка. Возможно, ученые, которые выделяли этот белок, по ошибке при выделении генетического материала из кишечника крысы
выделили мРНК E.coli , в больших количествах живущую в кишечнике крыс.
Файлы-списки
С помощью программы seqret пакета EMBOSS скачали в файл adh.fasta в fasta-формате все доступные в Swissprot последовательности алкогольдегидрогеназ.
Командой infoseq adh.fasta -only -usa получили файл-список.
Затем, с помощью команды infoseq adh.fasta -only -usa -out stdout|grep -f organisms.txt > org_list.txt получили файл со списком всех алкогольдегидрогеназ
заданных организмов.
Последовательности всех алкогольдегидрогеназ из базы данных SwissProt были получены командой seqret @org_list.txt org_adh.fasta
и их можно посмотреть здесь
EnsEMBL
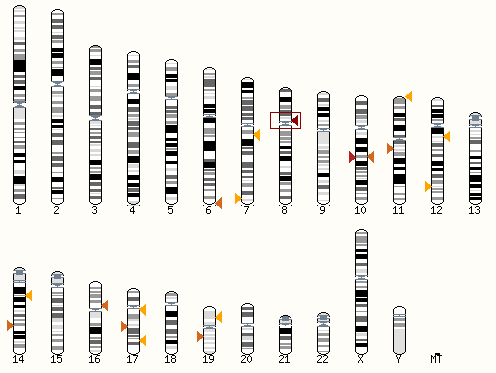
Ген человека, используемый ранее kat6a, был дан для работы программе EnsEMBL. На странице резульата в разделе Alignment Locations vs. Karyotype мы видим, что наш ген находится
в восьмой хромосоме ближе в центромере, чем к концу хромосомы. Так как это ген ацетилтрасферазы гистонов, который, скорее всего, должен сохранять кончервативность, то его располоожение
там логично.
На сайте можно найти экзон-интронную структуру данного гена и число возможных транскриптов. В экзон-интронной структуре расписаны фланкирующие последовательности, нестранслируемые
регионы, координаты каждого экзона и интрона.

Есть подробное выравнивание с указанием возможных замен и значения этого места в последовательности (экзон, интрон, нестранслируемая область и т.д.) Ниже представлен фрагмент
этого выравнивания.

В списке находок можно для каждой перейти ссылке "Contig view", где есть более конкретное указание региона хромосомы и делаkьная информация о регионе. С какими генами соседствует ген
исследуемого белка, что кодируют эти гены. Для каждого хита можно посмотреть выравнивание отдельно.

В целом, сервис очень информативен и позволяет узнать много информации об интересующем нас гене. Интерфейс вполне дружесвенный, местами сам себя дублирующий.