| На главную |
|
Мои проекты | Ссылки | Обо мне | Мои заметки |
Реконструкция филогенетических деревьев
1. Taxonomy browserТаксономия выбранных бактерий:
| Название | Мнемоника | Таксономия (домен; тип; класс; порядок; семейство; род; группа, если указана) |
| Bacillus subtilis | BACSU | Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus subtilis group |
| Clostridium tetani | CLOTE | Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium |
| Geobacillus kaustophilus | GEOKA | Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Geobacillus |
| Lactobacillus acidophilus | LACAC | Bacteria; Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus |
| Lactococcus lactis | LACLM | Bacteria; Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Lactococcus |
| Listeria monocytogenes | LISMO | Bacteria; Firmicutes; Bacilli; Bacillales; Listeriaceae; Listeria |
| Staphylococcus aureus | STAA1 | Bacteria; Firmicutes; Bacilli; Bacillales; Staphylococcaceae; Staphylococcus |
Соответствующее филогенетическое дерево

Все отобранные бактерии относятся к типу Firmicutes, 6 из 7 бактерий относятся к классу Bacilli. Бактерия Clostridium tetani относится к классу Clostridia - верхняя одинокая ветвь дерева CLOTE.
4 из 6 бактерий класса Bacilli относятся к порядку Bacillales - нетривиальная ветвь {BACSU, GEOKA, LISMO, STAA1} vs {CLOTE, LACAC, LACLM}. 2 из 6 относятся к порядку Lactobacillales - нетривиальная ветвь {LACAC, LACLM} vs {CLOTE, BACSU, GEOKA, LISMO, STAA1}.
2 из 4 бактерий порядка Bacillales относятся к одному семейству Bacillaceae - нетривиальная ветвь {BACSU, GEOKA} vs {CLOTE, LACAC, LACLM, LISMO, STAA1}. Другие 2 бактерии относятся к разным семействам порядка Bacillales - ветви LISMO и STAA1.
Бактерии порядка Lactobacillales принадлежат разным семействам - ветви LACAC и LACLM.
Из списка функций белков выбрала функцию Фактор элонгации трансляции Ts (EFTS), по белкам соответствующего семейства далее реконструировала филогенетическое дерево.
С помощью Uniprot получила из Swiss-Prot последовательности нужных белков из отобранных бактерий. Использовала идентификаторы вида EFTS_BACSU. Поместила последовательности в файл seq.fasta и отредактировала названия последовательностей, оставила только мнемонику видов. Порядок последовательностей соответствует приведенному в таблице.
Получила выравнивание.
Получаю изображение выравнивания в "блочной" форме (Format - Wrap), с раскраской по проценту идентичности.

Проект сохранен тут и получено выравнивание в fasta-формате.
4.
Реконструкция филогенетического дерева четырьмя методами JalView. Каждое дерево сохранено в Newick-формате.
Получила изображения деревьев, сделанные программой Mega.
Average Distance Using % Identity

Новые ветви: нет.
Отсутствующие ветви: нет.
Neighbour Joining Using % Identity
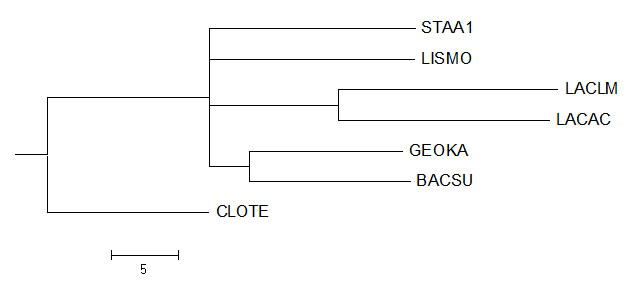
Новые ветви:
{LACLM, LACAC, GEOKA, BACSU} vs {STAA1, LISMO, CLOTE},
{LISMO, LACLM, LACAC, GEOKA, BACSU} vs {STAA1, CLOTE}.
Отсутствующие ветви:
{BACSU, GEOKA, LISMO} vs {CLOTE, LACAC, LACLM, STAA1},
{BACSU, GEOKA, LISMO, STAA1} vs {CLOTE, LACAC, LACLM}.
Average Distance Using BLOSUM62

Новые ветви:
{STAA1, LISMO} vs {CLOTE, GEOKA, BACSU, LACLM, LACAC}.
Отсутствующие ветви:
{BACSU, GEOKA, LISMO} vs {CLOTE, LACAC, LACLM, STAA1}.
Neighbour Joining Using BLOSUM62
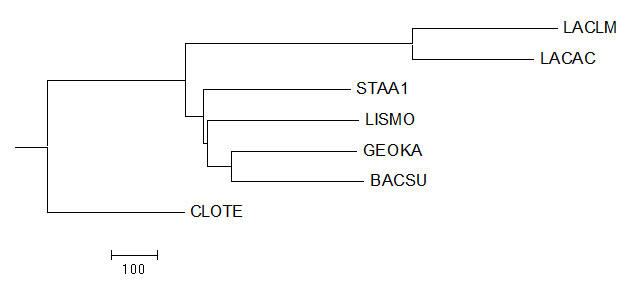
Новые ветви: нет.
Отсутствующие ветви: нет.
5.
Импортирую выравнивание (файл в fasta-формате) в программу Mega (при импорте выбираю "Analyze"). Реконструирую дерево методом "Maximum Parsimony" (кнопка Phylogeny). Укореняю дерево.
Полученное изображение

Новые ветви:
{LISMO, STAA1} vs {GEOKA, BACSU, LACAC, LACLM, CLOTE},
{LISMO, STAA1, GEOKA} vs {BACSU, LACAC, LACLM, CLOTE}.
Отсутствующие ветви:
{BACSU, GEOKA} vs {CLOTE, LACAC, LACLM, LISMO, STAA1},
{BACSU, GEOKA, LISMO} vs {CLOTE, LACAC, LACLM, STAA1}.