| На главную |
|
Мои проекты | Ссылки | Обо мне | Мои заметки |
Алгоритмы реконструкции деревьев
Укоренение в среднюю точку.
Неукорененное дерево из предыдущего задания (метод neighbor-joining):
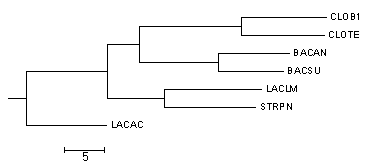
Это же дерево, укороненное в среднюю точку с помощью программы retree:
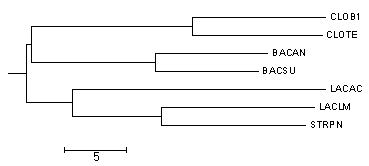
(укоренение в ветвь {CLOTE,CLOB1,BACSU,BACAN} vs {LACAC,STRPN,LACLM})
Правильное дерево:
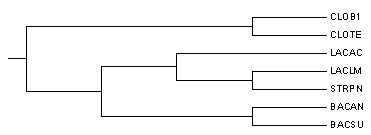
(укоренение в ветвь {CLOTE,CLOB1} vs {BACSU,BACAN,LACAC,STRPN,LACLM})
Видно, что укоренение в среднюю точку не дало правильного результата.
Не имеет смысла укоренять таким образом деревья, построенные методом UPGMA, так как этот метод, в отличие от neighbor-joining, предполагает молекулярные часы.
Использование внешней группы.
Деревья, построенные методом максимальной экономии ("Maximum parsimony") нельзя укоренить в среднюю точку, так как этот метод не реконструирует длины ветвей.
Укоренение с помощью внешней группы
Выровняем исходные белки вместе с белком из E. coli (внешняя группа); построим в MEGA дерево методом максимальной экономии; укореним его в ветвь, идущую к E. coli. Получившееся дерево: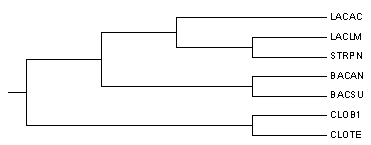
Дерево совпадает с правильным.
Бутстрэп.
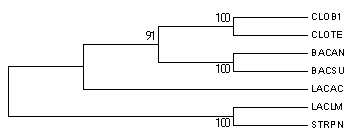
Результат бутстрэп-анализа филогении белков:
Original tree:
Bootstrap consensus tree:
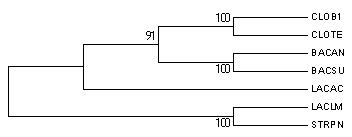
Оба дерева по топологии совпадают с правильным.