| На главную |
|
Мои проекты | Ссылки |
Совмещение структуры GAPDH кролика с ее структурными гомологами
Cтруктурные гомологи нашла при помощи сервиса PDBeFold, lowest acceptable match по умолчанию 70%. В таблице с находками выбрала 6 разных белков с RMSD от 0,8 до 2,5 ангстрем и N_align от 50% до 90%. Сервис сам показывает картинку совмещения структур(Рис. 7). На первый взгляд структуры очень похожи.
Рис.7. Совмещение структуры 1J0X и ее пяти структурных гомологов.
Выравнивание последовательностей по совмещению структур; совмещение структур.
Выравнивание последовательностей Tcoffee.
Посмотрим на петлю 3hja:D 102-105. В структурном выравнивании она не совпадает с другими структурами( см. рис.8).
Из выравниваний (Рис.9) видно, что аминокослоты 106-109 для последней последовательностью плохо выравниваются с любым участком из куска 102-109,
поэтому все равно куда их выровнять, следовательно, сложно сказать, где правильное выравнивание. В целом, выравнивания очень похожи.

Рис.8. Совмещение структуры 1J0X и ее пяти структурных гомологов, выпетливание невыравнивающегося участка.

Рис.9. Выравнивания структурных гомологов структуры 1J0X (JalView, раскраска Clustalx по консервативности 30%).
Совмещение структур альфа и бета доменов константной части T-клеточного рецептора
Была выбрана структура 1OGA: альфа-цепь region d:118-202, бета-цепь region e:119-245.
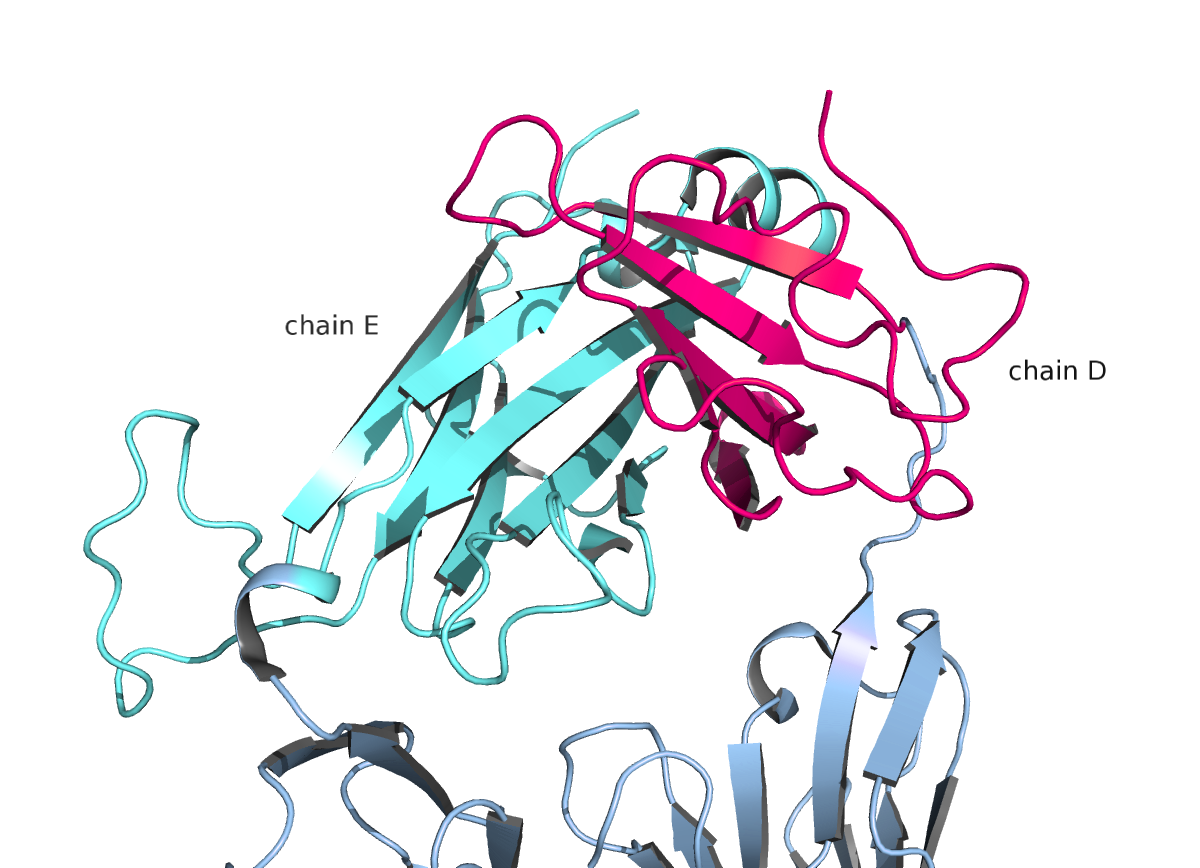
Рис.10. Альфа и бета домены структуры 1OGA.
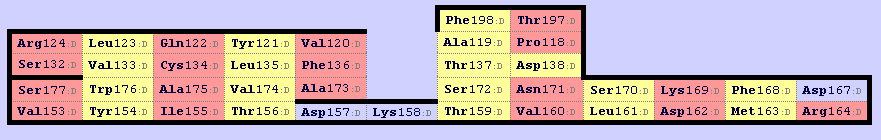
Рис.11. SheeP карта бета-листа для альфа-цепочки.
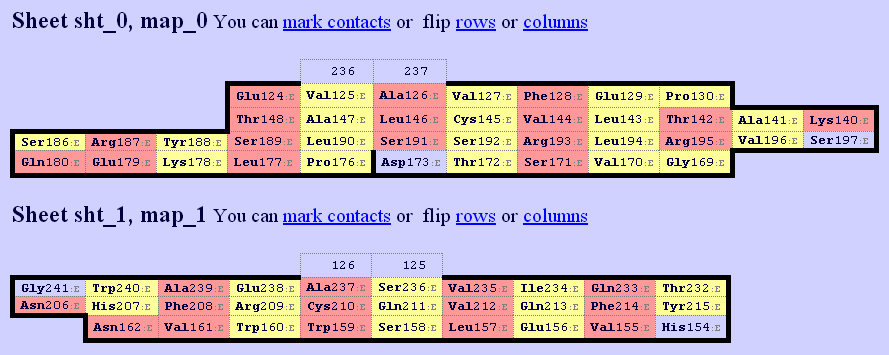
Рис.12. SheeP карта бета-листа для бета-цепочки.
Два консервативных цистеина Cys134:D (альфа-домен) и Cys145:E (бета-домен) отмечены синим. Выравнивание структур бета-листа альфа-домена и бета-листов бета-домена приведено на рисунках 13 и 14. Для выравнивания взяли девять СА атомов вокруг цистеина (включая его СА атом). Чтобы было проще сравнивать возьмем только бета-тяжи, так как петли являются менее регулярными и более подвижными элементами структуры.
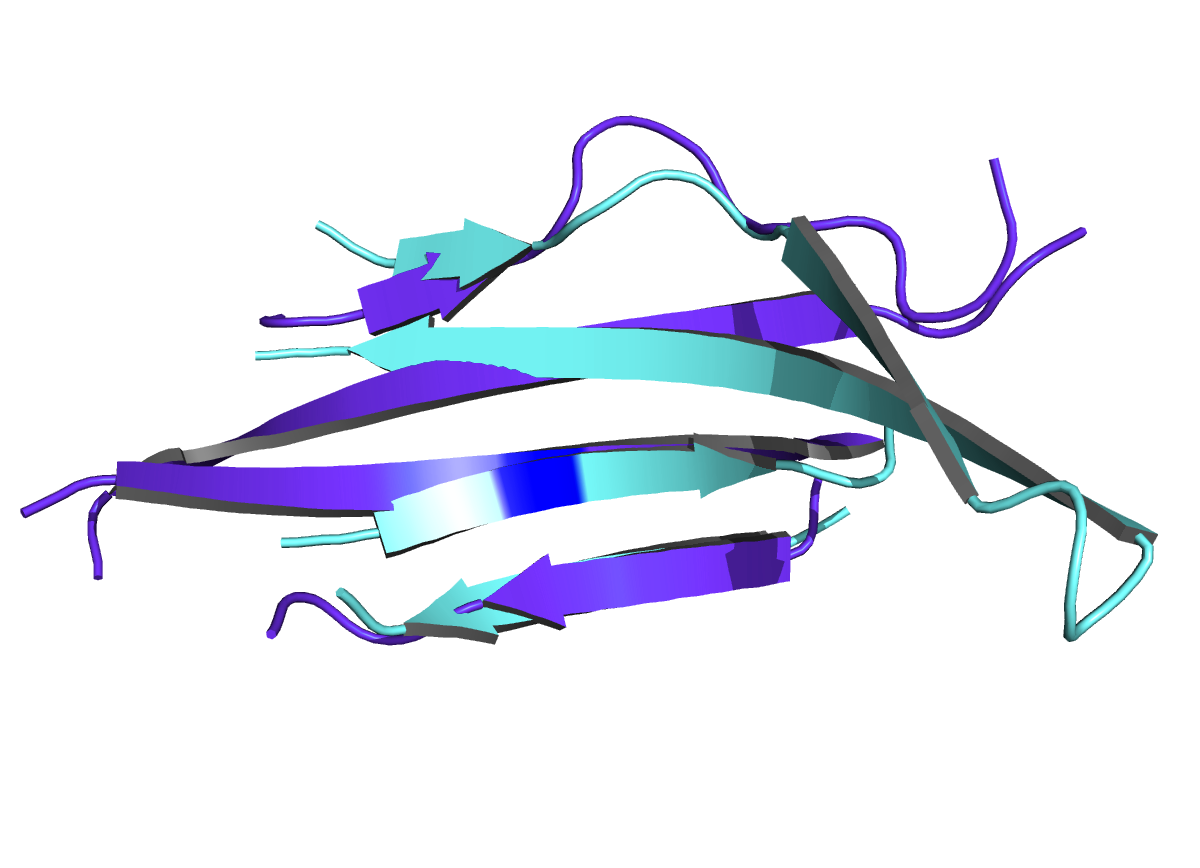
Рис.13. Выравнивание структур бета-листа альфа-домена и первого (ближе к альфа-домену) бета-листа бета-домена.
pair_fit alpha_list///121-123+133-135+174-176/CA, beta_1///126-128+144-146+191-193/CA

Рис.14. Выравнивание структур бета-листа альфа-домена и второго (дальше от альфа-домена) бета-листа бета-домена.
pair_fit alpha_list///121-123+133-135+174-176/CA, beta_2///236-238+209-211+158-160/CA
RMSD первого наложения 0,485, второго 0,548, по визуальным признакам со вторым листом бета-домена выравнивается лучше. Сходство топологий наблюдается скорее во втором случае, так как различие в качестве наложения не столь велико, но количество и направление бета-тяжей совпадает.