1. ВВЕДЕНИЕ
Таксономическое положение изучаемой бактерии Thermusbrockianus [1]:
| Таксономический ранг | Название таксона |
|---|---|
| Надцарство | Bacteria |
| Тип | Deinococcota |
| Класс | Deinococci |
| Порядок | Thermales | Семейство | Thermaceae |
| Род | Thermus |
| Вид | Thermus brockianus |
Thermus brockianus - термофильная, аэробная, грамотрицательная неподвижная бактерия, впервые была обнаружена в геотермальной зоне Гейсир, Исландия. Thermus brockianus имеет высокую оптимальную температуру около 70°C Разлагающие ферменты, выделенные из этих бактерий, используются в различных целях в промышленности, так как имеют высокую устойчивость к повышенным температурам.. Геном бактерии состоит из хромосомы, одной мегаплазмиды и одной плазмиды [2].
2 МАТЕРИАЛЫ И МЕТОДЫ
2.1 Материалы
Материалами для данного мини-обзора выступили:
1. Таблица особенностей генома бактерии Thermus brockianus: *ССЫЛКА*
2. Таблица CDS (кодирующих последовательностей) бактерииThermus brockianus: *ССЫЛКА*
3. Полный геном бактерии Thermus brockianus: *ССЫЛКА*
4. Протеом бактерии Escherichia coli K12 для сравнение его спротеомом бактерии Thermus brockianu: *ССЫЛКА*
5. Первые три файла были взяты из базы данных NCBI длябактерии Thermus brockianus: *ССЫЛКА*
6. Последний файл был взят из базы данных NCBI для бактерииEscherichia coli K12: *ССЫЛКА*
2.2 Методы
2.2.1 Эектронные таблицы
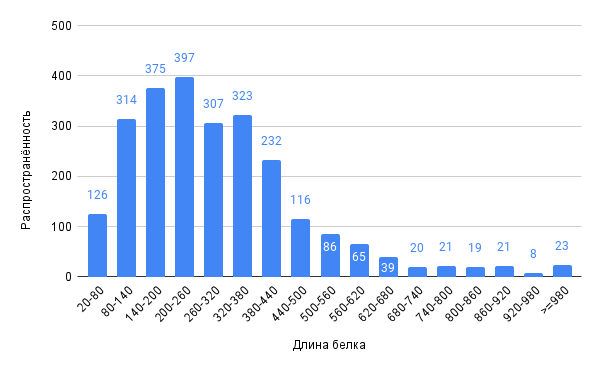
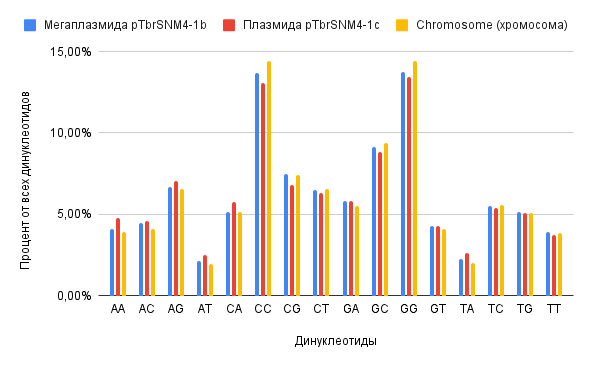
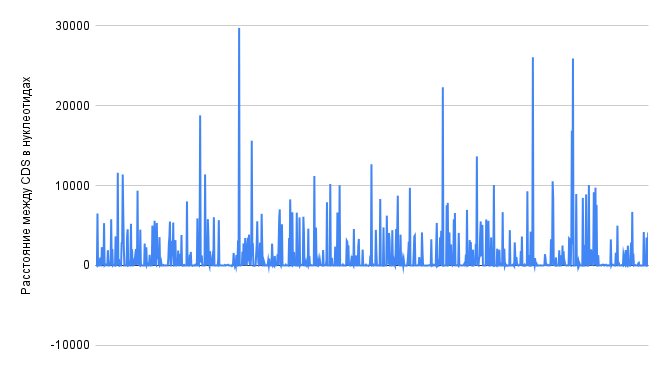
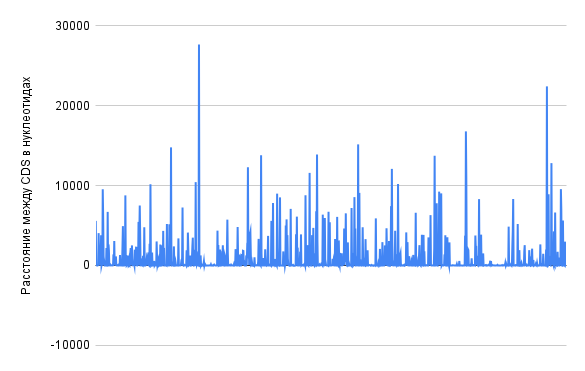
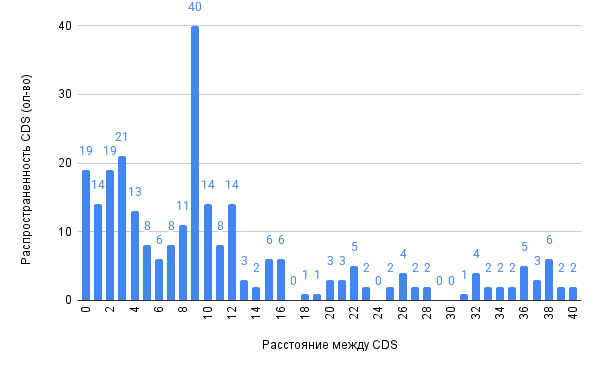
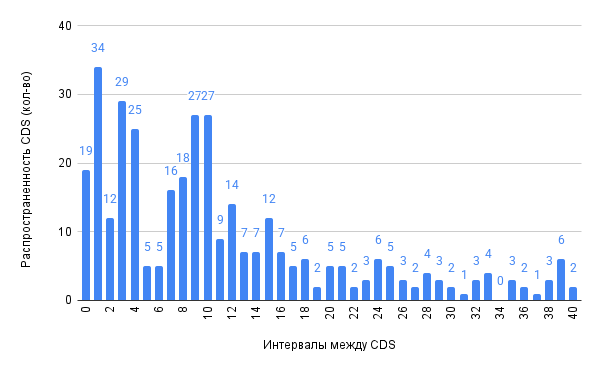
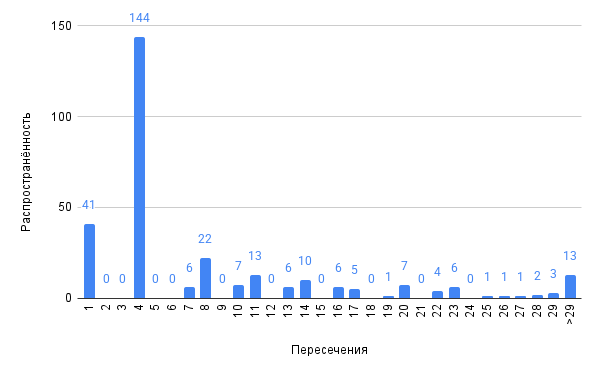
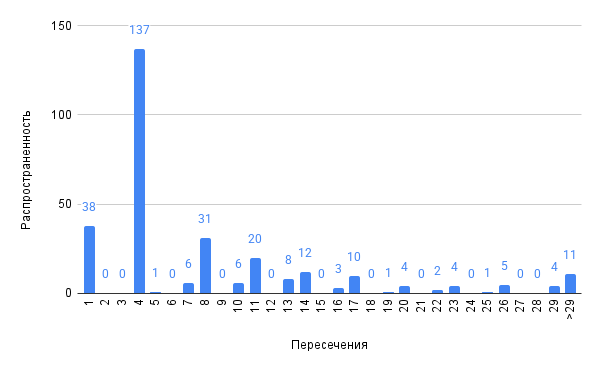
Для обработки данных из таблиц особенностей генома и кодирующих последовательностей бактерии Thermus brockianus они импортировались в электронные таблицы. При обработке использовались функции: =COUNTIF, =COUNTIFS, =MIN, =MAX, =AVERAGE – также накладывались фильтры для избирательного выбора данных (3.5 “Анализ генов репликонов”; 3.7 “Интервалы между CDS и их пересечения”; 3.8 “Исследование особенностей бактерии на чужеродные гены”). Также использовались инструменты электронных таблиц для создания диаграмм, представляющих данные в удобном для просмотра виде (Диаграмма длин белков, диаграмма доли гуанина, диаграмма распределения динуклеотидов по репликонам, диаграммы интервалов между CDS на плюс и минус цепях и диаграммы пересечений CDS на плюс и минус цепях).
Электронные таблицы также были использованы для обработки данных, полученных при помощи программ, написанных на языке программирования Python 3.0, и при помощи команд командной оболочки Bash.