Семейство белковых доменов - DEAD. PFAM.
Задание 1. Краткое описание домена DEAD
Ссылка на PFAM
Краткое описание: Семейство белкового домена DEAD/DEAH box helicase входит в клан P-loop_NTPase. Белки этого семейства, являясь АТФ-зависимыми РНК-хеликазами, участвуют в процессе раскручивания двуцепочечных молекул РНК на одноцепочечные. Белки данного домена принимают участие в метаболизме РНК и необходимы при транскрипции, сплайсинге мРНК, инициации трансляции, сборка рибосом, распаде РНК и экспресии генов органелл.
| Информация | |
|---|---|
| AC pfam |
PF00270
|
| ID pfam | DEAD/DEAH box helicase |
| #SEED | 181 белок |
| #All | 708k белков | #SW | 1715 белков |
| #architectures | 6978 архитектур доменов |
| #3D | 650 белков |
| Taxonomy | 707584 белков 35982 вида |
| #eukaryota |
278230 белков 5257 видов |
| #archaea |
19792 белка 1718 видов |
| #bacteria |
398130 белков 28166 видов |
Задание 2. Карта локального сходства двух белков - Q5YRB2_NOCFA и CAS3_ECOLI
| Информация | Комментарии | |
|---|---|---|
| Доменная архитпектура 1 | Cas3_HD - DEAD - Cas3-like_C_2 - Cas3_C | PF18019 - PF00270 - PF22590 - PF18395 |
| Белок с архитектурой 1 | Q5YRB2_NOCFA – Helicase Cas3 |
AC: Q5YRB2 Length: 914 |
| Доменная архитпектура 2 | DEAD - Cas3-like_C_2 - Cas3_HD | PF00270 - PF22590 - PF18019 |
| Белок с архитектурой 2 | CAS3_ECOLI – CRISPR-associated endonuclease/helicase Cas3 |
AC: P38036 Length: 888 |
Для построения карты локального сходства я выбрал два белка: Helicase cas3 (AC:Q5YRB2) из Nocardia farcinica (вид граммположительных инфекционных бактерий) и CRISPR-associated endonuclease/helicase Cas3 (AC:P38036) из Escherichia coli (вид граммотрицательных бактерий).
Данные cas3 белки выполняют функции антивирусной защиты, они связываются с двуцепочечной ДНК, расплетают её с затратой АТФ на одноцепочечные цепи (хеликазная активность), совершают отжиг ДНК с РНК, а после гидролизуют фосфодиэфирные связи в цепи ДНК (эндонуклеазнаяя активность). Интересно то, что кроме ДНК/ДНК и ДНК/РНК нуклеазных активностей присущих всем белкам cas3, данные ещё и обладают РНК/РНК нуклеазной активностью.
Белок Q5YRB2 (первый белок) имеет доменную архитектуру:
Cas3_HD - DEAD - Cas3-like_C_2 - Cas3_C
(10-221) - (295-476) - (550-657) - (792-905)

Белок P38036 (второй белок) имеет доменную архитектуру:
Cas3_HD - DEAD - Cas3-like_C_2
(11-235) - (309-491) - (567-671)

При помощи программы BlastP я построил карты локального сходства при пороге E-value=0.05 и E-value=10.
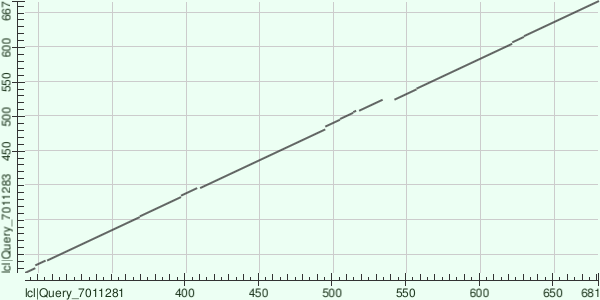
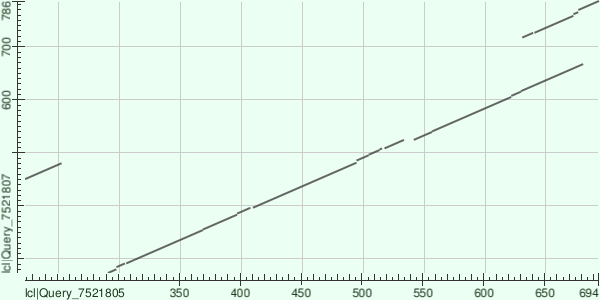
По горизонтали у нас белок P38036 (второй белок), по вертикали у нас Q5YRB2 (первый белок)
При пороге E-value=0.05 в локальном выравнивании берётся участок от 292-ой до 681-ой аминокислоты второго белка P38036 (по горизонтали) и участок от 274-ой до 667-ой аминокислоты первого белка Q5YRB2 (по вертикали)
На данном участке мы наблюдаем всего 13 инделов, то есть можно точно утвержать о как минимум суммарно 13 инсерциях или делециях, произошедших на данном участке со времени дивергенции видов.
Также интересно то, что на карте локального сходства участок 292-681(aa) второго белка P38036 (по горизонтали) соответствует доменам DEAD и Cas3-like_CC2
Аналогично участок 274-667(aa) первого белка Q5YRB2 (по вертикали) соответвует доменам DEAD и Cas3-like_C_2
При этом общий для них домен Cas3_HD, отввечающий за нуклеазную активность (по одношению к одноцепочечным РНК и ДНК) белка не попал на карту локального сходства, то есть этот домен у данных белков имеет низких процент схожести. Возможно в процессе эволюции этот домен так сильно преобразовался у этих двух видов бактерий (граммотрицательные и граммположительные бактерии эволюционно очень далеко находятся друг от друга), что несмотря на функциональную схожесть и схожесть 3D структур, аминокислотных состав, за исключением нескольких консервативных аминокислот активного центра, стал очень разным.
Если же увеличить порог E-value до 10, то кроме предыдущего участка, на карте локального сходства будут ещё участки:
–от 632-ой до 694-ой аминокислоты второго белка P38036 (по горизонтали) и участок от 718-ой до 786-ой аминокислоты первого белка Q5YRB2 (по вертикали)
–от 224-ой до 253-ой аминокислоты второго белка P38036 (по горизонтали) и участок от 451-ой до 480-ой аминокислоты первого белка Q5YRB2 (по вертикали)
Между участками белков наблюдается небольшая схожесть, однако из-за большого значению E-value сложно говорить о достоверности этих находок. Существует лишь небольшая вероятность того, что произошла траслокации (в случае первой находки) или дупликация (в случае второй находки)