Выравнивание белков
Задание 1. Программа подсчёта инделей
Задание 2. Глобальное парное выравнивание гомологичных белков
| Protein Name | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels |
|---|---|---|---|---|---|---|---|
| DNA repair protein RadA | RADA_ECOLI | RADA_BACSU | 1067.5 | 46.8% | 66.0% | 18/468 | 10 |
| Large ribosomal subunit protein bL9 | RL9_ECOLI | RL9_BACSU | 195.0 | 33.1% | 54.3% | 4/151 | 4 |
| Adenine deaminase | ADEC_ECOLI | ADEC_BACSU | 853.5 | 34.7% | 52.8% | 39/602 | 14 |
Задание 3 и 4
| Protein Name | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels | Coverage 1 | Coverage 2 |
|---|---|---|---|---|---|---|---|---|---|
| DNA repair protein RadA | RADA_ECOLI | RADA_BACSU | 1075.0 | 47.4% | 67.0% | 14/460 | 8 | 98.70% | 98.69% |
| Large ribosomal subunit protein bL9 | RL9_ECOLI | RL9_BACSU | 195.0 | 33.3% | 54.7% | 3/150 | 3 | 100.00% | 99.32% |
| Adenine deaminase | ADEC_ECOLI | ADEC_BACSU | 858.5 | 36.3% | 54.9% | 25/570 | 12 | 94.39% | 96.71% |
Белки гомологичны почти по всей длине, так как покрытие в локальном выравнивании приближается к 100 процентам.
Сравнение глобального выравнивания с локальным может быть информативным, вот некоторые факты, которые я смог узнать:
- ⯀ Небольшие участки концов белков (несколько аминокислот) починки ДНК RadA негомологичны и отсутствуют в локальном выравнивании
- ⯀ Локальное выравнивание рибосомальных белков говорит нам о том, что белки гомологичны почти по всей длине, за исключением одной аминокислоты в конце.
- ⯀ Локальное выравнивание по аденин деаминазе более интересное: так как начальные участки белков не гомологичны, что говорит о том, что они малоконсервативны и их изменения оказывает незначительное влияние на функции белков. Также есть пара негомологичных аминокислот в конце.
Задание 5
| Тип выравнивания | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels | Coverage 1 | Coverage 2 |
|---|---|---|---|---|---|---|---|---|---|
| Глобальное | MURI_ECOLI | CPXY_BACSU | 22.5 | 10.6% | 20.7% | 283/489 | 16 | - | - |
| Локальное | MURI_ECOLI | CPXY_BACSU | 49.5 | 18.0% | 30.3% | 126/267 | 11 | 78.60% | 56.83% |
По результатам выравнивания случайных белков: белка Cytochrome P450 из кишечной палочки и Glutamate racemase из сенной палочки - я понял, что белки не являются гомологичными.
На то есть несколько причин: во-первых, очень низкий вес выравнивания, во-вторых, низкий процент идентичности (меньше 20 процентов даже при локальном выравнивании) и схожести, огромное количество гэпов (занимающих около половины всего выравнивания). К тому же максимальное число подряд идущих одинаковых аминокислот в выравнивании равно 4, что крайне мало.
Задание 6
Для множественного выравнивания я выбрал мнемонику белков - RADA; рекомендованное полное имя - "DNA repair protein RadA". При поиске в UniProtKB белков с мнемоникой RADA нашлось 92 результата, из них 6 белков было удалено, то есть всего 86 белков из Swiss-Prot при поиске через UniProtKB. При поиске белков из Swiss-Prot при помощи EMBOSS было найдено 84 белка с такой мнемоникой (получается, что за то время, пока Swiss-Prot на kodomo не обновлялся успели добавить ещё 2 белка с мнемоникой RADA). Кроме белков сенной и кишечной палочки, я случайно выбрал ещё 5 белков из следующих организмов:
Helicobacter pylori (RADA_HELPY), Sulfolobus acidocaldarius (RADA_SULAC), Thermoplasma volcanium (RADA_THEVO), Picrophilus torridus (RADA_PICTO), Rickettsia typhi (RADA_RICTY)
Для выравнивания я по инструкции: сначала создал текстовый файл из 7 строчек, каждая из которых содержала: "sw:[ID белка]"
Затем перевёл его в fasta формат при помощи команды seqret, а в конце при помощи команды muscle провёл множественное выравнивание и импортировал файл в Jalview.
Ccылка на файл Jalview
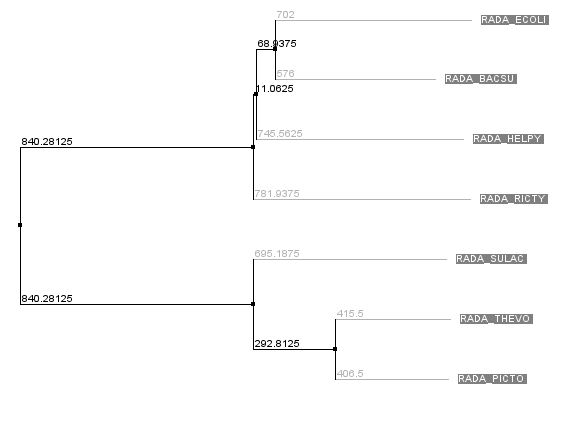
По результатам множественного выравнивания я могу сказать, что белки выравнялись хорошо и все они с большой долей вероятностью являются гомологичными, однако среди 7 белков можно выделить 2 группы наиболее схожих и родственно близких белков:
Первая группа состоит из 3 белков Sulfolobus acidocaldarius (RADA_SULAC) - 321 aa, Thermoplasma volcanium (RADA_THEVO) - 323 aa, Picrophilus torridus (RADA_PICTO) - 323 aa
Вторая группа состоит из 4 белков Escherichia coli (RADA_ECOLI) - 460 aa, Bacillus subtilis (RADA_BACSU) - 458 aa, Helicobacter pylori (RADA_HELPY) - 448 aa, Rickettsia typhi (RADA_RICTY) - 445 aa
Совпадающие позиции в выравнивании у всех 7 белков: 1, 102-105, 116, 119, 121-122, 152, 214-215, 245, 328, 338
Также существуют позиции в выравнивании, на которых у одного или двух белков случилась мутация: на 93 позиции у RADA_HELPY треонин заменился на серин, на 406 позиции у RADA_PICTO глицин заменился на серин. По расчётам наиболее консервативные участки это: 101-106, 121-126, 147, 149, 151-152, 211-217, 230-232 - в них или у всех белков одинаковая аминокислота, либо они разные, но обладают одинаковыми свойствами.
Также в программе Jalview можно построить парные выравнивания между интересующей парой белков. Используя эту функцию я выяснил, что RADA_THEVO и RADA_PICTO неимоверно схожи (процент идентичности = 81.73%).
Первая группа белков на сотню нуклеотидов меньше второй из-за чего во множественном выравнивании наблюдается огромное число гэпов у белков первой группы на позициях: 266-324, 344-392, 412-459, 461-474 - возможно при дивергенции этих бактерий произошло несколько инсерций или делеций. Эти участки также можно считать наименее консервативными при сравнении групп белков.