Практикум 8. Нуклеотидный бласт
Задание 1. δ-субъединица АТФ-синтазы Penicillium digitatum
При помощи средств поиска bash я нашёл преполагаемую дельта цепь митохондриальной АТФ-синтазы: ATP synthase delta chain, mitochondrial
Также была получена последовательность FASTA этого белка
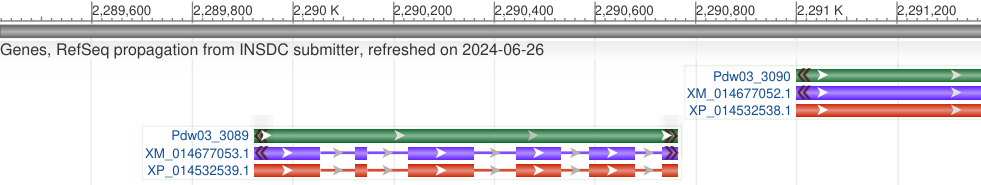
Идентификатор нуклеотидной транскрипта: XM_014677052
Идентификатор нуклеотидной записи хромосомы на которой располагается ген: NC_089384.1
Идентификатор белка: XP_014532539.1
Координаты кодирующей части гена: 2 289 923-2 290 766 нуклеотиды на Chromosome 1
Нуклеотидная последовательность ДНК гена delta цепи АТФ-синтазы с небольшой окрестностью до и после в FASTA-формате
Задание 2. Применение различных вариантов BLAST для последовательности ДНК.
Все 4 семейства: Пауки, Пчёлы, Кошачьи, Собачьи - генетически далеки от Penicillium digitatum, а потому подходят для выполнения задания. Я выбрал семейство Собачьих - Canidae (можно было выбрать любое из 4 семейств)
Нам необходимо сравнить результаты поиска гомологов по нуклеотидной последовательности и по белковой последовательности (транслированной геномной или последовательности белка) среди геномов семества Canidae, где в качестве запроса выступает CDS гена delta chain ATP synthase Penicillium digitatum
База данных RefSeq Genome Database (refseq genomes) включает 7 геномов собак
Поиск при помощи megablast ничего не дал, так как длина слова-якоря слишком большая и так как это алгоритм подходит только для последовательностей с высокой схожестью.
Поэтому в этой работе я использовал алгоритмы tblastx и blastn, так как оба алгоритма используют ген в качестве запроса, но tblastx транслирует его по всем рамкам считывания и ищет по транслированной нуклеотидной базе данных, а blastn ничего не транслирует и ищет по нуклеотидной базе данных. Это позволит нам произвести более точное сравнение двух алгоритмов и показать, что гомология прослеживается лучше в случае с белковой последовательностью, так как мутированный кодон может продолжать кодировать ту же аминокислоту, что и до мутации.
Для алгоритма tblastx: была взята последовательность CDS гена с окрестностями из предыдущего задания в качестве запроса, установлена длина слова 2 нуклеотида и оставлены стандартные значения остальных параметров.
Для алгоритма blastn: была также взята нуклеотидная последовательность из предыдущего задания, установлена длина слова 7 нуклеотидов, порог E-value 1 и оставлены стандартные значения остальных параметров.


Задание 3. Нахождение генов рРНК по далекому гомологу
makeblastdb -in GCF_016767815.1_ASM1676781v1_genomic.fna -dbtype nucl -out database.blast
Командой выше я создал базу данных для blast из последовательности генома Penicillium digitatum
Затем при помощи приведённых ниже двух команд нашел участки сходства для 16S RNA и 23S RNA на последовательности генома Penicillium digitatum:
blastn -task blastn -query 16S_rRNA_ecoli.fasta -db database.blast -out 16Sblastfmt.out -word_size 4 -outfmt 7
blastn -task blastn -query 23S_rRNA_ecoli.fasta -db database.blast -out 23Sblastfmt.out -word_size 4 -outfmt 7
В результате получил 2 таблицы:
Результат выдачи blastn для 16S RNA
Результат выдачи blastn для 23S RNA
Однако по текстовым файлам неудобно выявлять гомологи, поэтому привожу ссылки на google-таблицы, где координаты начала выравнивания геномной последовательности Penicillium digitatum располагаются по возрастанию.
Таблица для удобного нахождения гомологов
Для 16S rRNA E.coli обнаружилось 33 находки в геноме Penicillium digitatum из них 15 находок имеют E-value меньше 0.5; 15 находкам с маленьким E-value соответствует только 3 гомолога (которые являются копиями друг друга) на минус-цепи (в гугл-таблицах гомологи, содержащие множество находок, я разукрасил разными цветами).
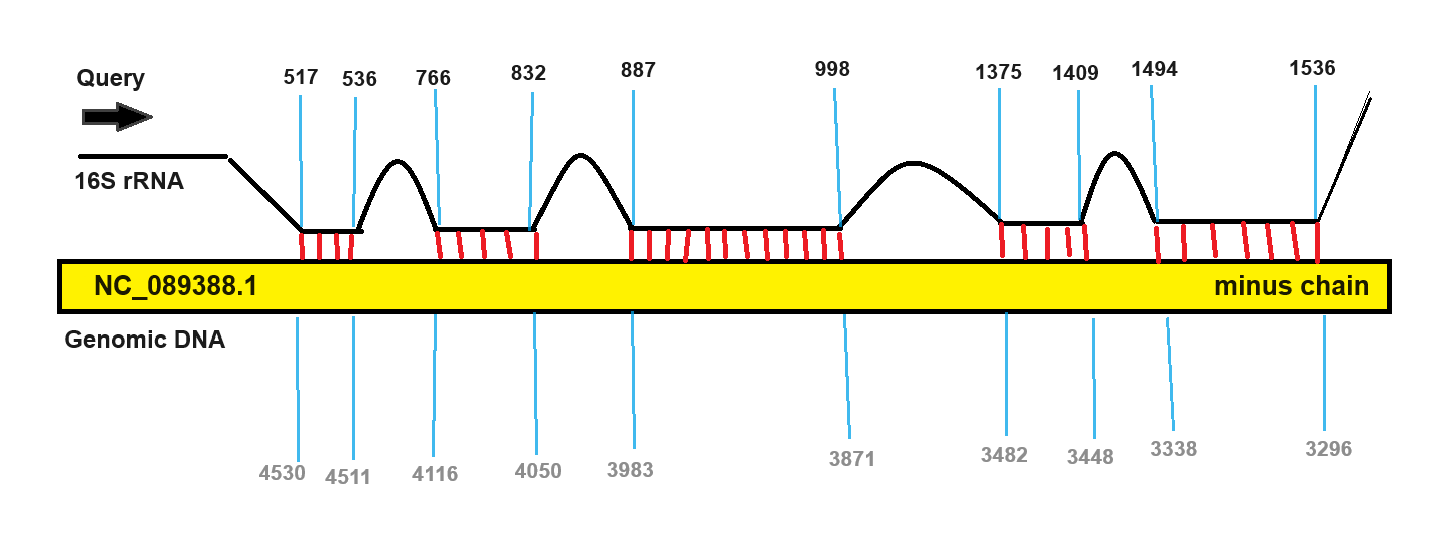
Для запроса 23S rRNA E.coli было найдено 32 находки в геномной последовательности Penicillium digitatum. Имеются 3 гомологичные последовательности, которые содержат большое количество находок (6, 7 и 7 находок) + 3 находки с крайне низким E-value (1.17e-14, 3.82e-08 и 3.82e-08).
Задание 4. Построение карт локального сходства для двух геномов
Для этого задания я решил выбрать для построение карт локального сходства - архей из рода Thermus.
Я выбрал Thermus brockianus (про него был написан мой миниобзор, поэтому я и решил использовать архей из рода Thermus для построение карт локального сходства) и Thermus thermophilus HB8. Для построения карт я выбрал их хромосомы
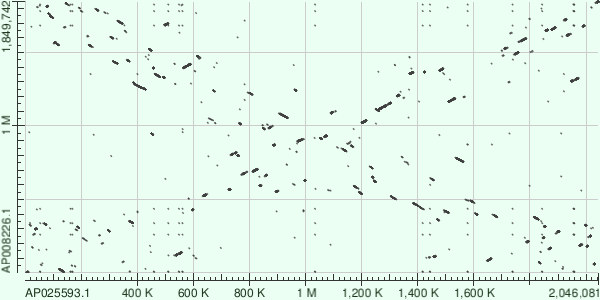
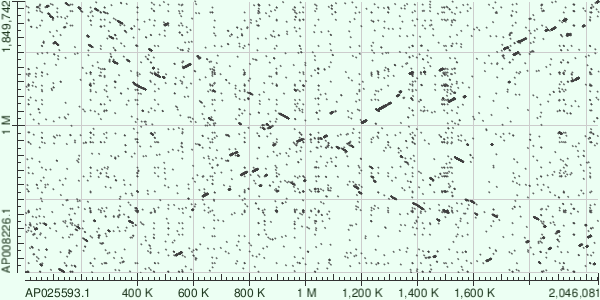
tblastx выдал ошибку при нахождении гомологических последовательностей между хромосомами. Возмножно tblastx не поддерживает построение карт локального сходства между настолько большими последовательностями.
На картах локального сходства наблюдается интересная ситуация. Гомологичные последовательности образуют крест. Возможно к расхождению видов привела крупная мутация, которая заключалась в том, что геном дублировался, а затем, перевернувшись, одна из копий вставилась в другую. Так как хромосома Thermus brockianus больше, возможно именно в его ДНК произошла похожая мутация.