Трансмембранные белки
Задание 1. База данных OPM
Белок с альфа-спиралями в трансмембранном пространстве - Blue-light absorbing proteorhodopsin (4knf)
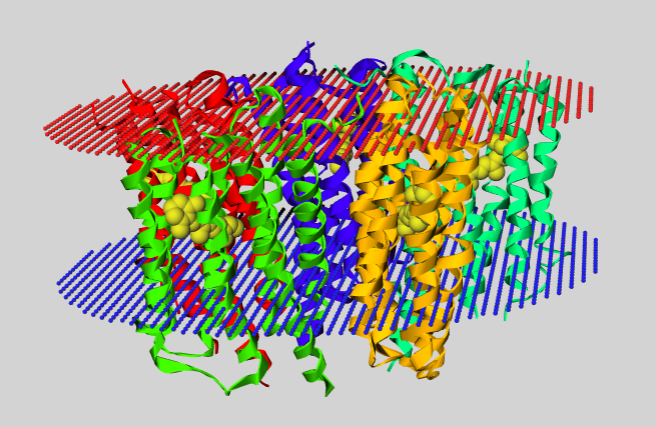
| Толщина гидрофобной части белка в мембране | 28.4 ±- 1.5 A |
| Координаты трансмембранных спиралей |
Subunits: 5 |
| Среднее количество остатков в одной трансмембранной спирали | 20.6 |
| Положение | Внутренняя мембрана Грам-отрицательной бактерии |
Белок с бета-листами в трансмембранном пространстве - Outer membrane protein A (OMPA) (1bxw)
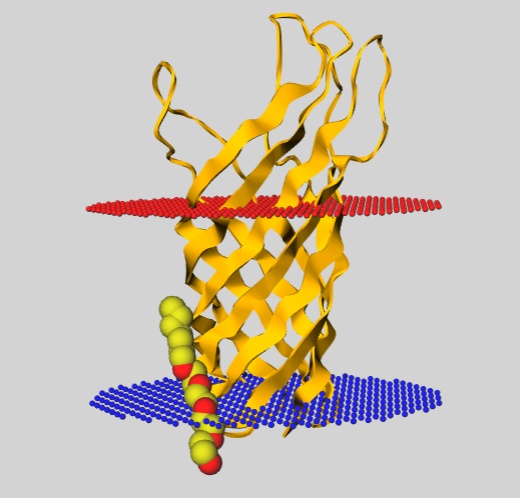
| Толщина гидрофобной части белка в мембране | 25.4 ± 1.9 A |
| Координаты трансмембранных спиралей |
Subunits: 1 |
| Среднее количество остатков в одной трансмембранной спирали | 9.5 |
| Положение | Внешняя мембрана Грам-отрицательной бактерии |
Задание 2. Анализ предсказания трансмембранных спиралей
Последовательность белка Blue-light absorbing proteorhodopsin (4knf) в fasta-формате была подана на вход сервису TMHMM. Выдача оказалась следующей:
# sp|Q9AFF7|PRRB_PRB02 Length: 251 # sp|Q9AFF7|PRRB_PRB02 Number of predicted TMHs: 7 # sp|Q9AFF7|PRRB_PRB02 Exp number of AAs in TMHs: 153.43582 # sp|Q9AFF7|PRRB_PRB02 Exp number, first 60 AAs: 36.87104 # sp|Q9AFF7|PRRB_PRB02 Total prob of N-in: 0.86117 # sp|Q9AFF7|PRRB_PRB02 POSSIBLE N-term signal sequence sp|Q9AFF7|PRRB_PRB02 TMHMM2.0 inside 1 4 sp|Q9AFF7|PRRB_PRB02 TMHMM2.0 TMhelix 5 22 sp|Q9AFF7|PRRB_PRB02 TMHMM2.0 outside 23 31 sp|Q9AFF7|PRRB_PRB02 TMHMM2.0 TMhelix 32 50 sp|Q9AFF7|PRRB_PRB02 TMHMM2.0 inside 51 62 sp|Q9AFF7|PRRB_PRB02 TMHMM2.0 TMhelix 63 85 sp|Q9AFF7|PRRB_PRB02 TMHMM2.0 outside 86 99 sp|Q9AFF7|PRRB_PRB02 TMHMM2.0 TMhelix 100 122 sp|Q9AFF7|PRRB_PRB02 TMHMM2.0 inside 123 141 sp|Q9AFF7|PRRB_PRB02 TMHMM2.0 TMhelix 142 164 sp|Q9AFF7|PRRB_PRB02 TMHMM2.0 outside 165 188 sp|Q9AFF7|PRRB_PRB02 TMHMM2.0 TMhelix 189 211 sp|Q9AFF7|PRRB_PRB02 TMHMM2.0 inside 212 222 sp|Q9AFF7|PRRB_PRB02 TMHMM2.0 TMhelix 223 245 sp|Q9AFF7|PRRB_PRB02 TMHMM2.0 outside 246 251
Сервис предсказал 7 трансмембранных спиралей (Number of predicted TMHs). В целом предсказанные границы близки к настоящим, почти совпадают границы 1 и 5 сабюнитов: 28-50 и 148-166 в ОРМ - 32-50 и 142-164 в TMHMM. Предсказан сабюнит перед первым (в ОРМ) с границами 5-22, пропущен 4 сабюнит 123-143.
Ниже приведена графическая выдача TMHMM. По оси Х отложена кордината аминокислоты в аминокислотной последовательности от N к C концу, по Y - вероятность этой аминокислоты оказаться в трансмембранной части (красная линия), красными блоками нарисовано предположительное положение трасмембранных участков в последовательности, синей линией изображена вероятность участка последовательности находиться во внутренней стороне мембраны, розовым цветом - во внешней.
Посмотрим на выдачу сервиса Phobius. Он предсказал 6 трансмембранных спиралей. Phobius не предсказал сабюнит до первого, в отличие от TMHMM. Третий и четвертый сабюниты базы ОРМ 3( 91- 109), 4( 123- 143) в выдаче Phobius превратились в один сабюнит 99-125. Границы остальных спиралей близки к настоящим.
ID sp|Q9AFF7|PRRB_PRB02 FT SIGNAL 1 20 FT REGION 1 3 N-REGION. FT REGION 4 14 H-REGION. FT REGION 15 20 C-REGION. FT TOPO_DOM 21 29 NON CYTOPLASMIC. FT TRANSMEM 30 50 FT TOPO_DOM 51 61 CYTOPLASMIC. FT TRANSMEM 62 79 FT TOPO_DOM 80 98 NON CYTOPLASMIC. FT TRANSMEM 99 125 FT TOPO_DOM 126 145 CYTOPLASMIC. FT TRANSMEM 146 165 FT TOPO_DOM 166 184 NON CYTOPLASMIC. FT TRANSMEM 185 211 FT TOPO_DOM 212 222 CYTOPLASMIC. FT TRANSMEM 223 244 FT TOPO_DOM 245 251 NON CYTOPLASMIC.
Посмотрим на графическое изображение Phobius. По оси Х отложена координата аминокислоты, по оси Y - вероятность для данного участка оказаться сигнальным пептидом (красная линия), цитоплазматическим участком (зеленой линией), нецитоплазматическим участком (синим цветом) и трансмембранным участком (серым цветом).
Работа Phobius показалась более правдоподобной. Оба сервиса ошиблись в районе 5-6 сабюнитов, но TMHMM к тому же предсказал несуществующий сабюнит перед первым.
Задание 3. База данных TCDB
Для белка с альфа-спиралями по ID Q9AFF7 найдена запись 3.E.1.6.12. Цифра 3 в начале значит, что это основной активный транспортер, использующий первичные источники энергии: химические, электрические и солнечные. 3.E значит, что он используют световую энергию для управления транспортированием растворенного вещества. 3.Е.1 - Семейство ион-транслоцирующих микробных родопсинов (MR), далее цифра 6 обозначает подсемейство, а последняя цифра 12 указывает на конкретный белок.
Для белка с бета-листами найдена запись 1.B.6.1.1. Цифра 1 означает, что белок относится к канальным фасилитаторам, то есть катализирует облегченную диффузию (с помощью энергонезависимого процесса) путем прохождения через трансмембранную водную пору или канал без подтверждения механизма, опосредованного носителем. Далее 1.B - класс бета-листовых поринов, 1.B.6 - семейство OmpA-OmpF поринов, дальше цифра 1 - подсемейство, и последняя цифра указывает на данный белок.