

Обзор сводной информации о геноме некоторого организма является удобной формой представления некоторых аспектов организации генома в краткой форме. В данной работе приведен обзор генома Methylomusa anaerophila – сравнительно недавно описанной бактерии, имеющей практическое значение для создания микробных топливных элементов. Данные, вошедшие в обзор, были обработаны при помощи методов электронных таблиц.
Methylomusa – род бактерий из типа Firmicutes, описанный вместе с типовым видом Methylomusa anaerophila, в работе Амано с соавторами (Amano et al., 2018). Представители M. anaerophila были выделены из сообществ, образующихся на аноде в микробных топливных элементах (MFCs – Microbial Fuel Cells) (Cao et al., 2019). Они представляют собой немного изогнутые палочки, несущие один жгутик; анаэробны, не образуют клеточных агрегатов (Amano et al., 2018). Наличие M. anaerophila в подобных установках обусловлено тем, что эти бактерии используют метанол, являющийся загрязнителем в сточных водах, как источник углерода и энергии, преобразуя в конечном счете его в ацетат, который затем используются другими участниками сообщества (Amano et al., 2018). Подобные топливные элементы, в которых для получения электричества используется загрязненная вода, были описаны ещё в 2004 году в работе Лю с соавторами (Liu et al., 2004).
Геном M. anaerophila состоит из 4781198 п.н. Недавно в их геноме были обнаружены гены альтернативных нитрогеназ (Addo & Dos Santos, 2020).
Для обработки и визуализации данных нами была использована программа Excel из пакета MS Office 2016. Для составления сводных таблиц по конкретным темам на основе общей таблице по геному мы использовали ряд методов электронных таблиц, а именно функции: СЦЕПИТЬ, ДЛСТР, СЧЁТЕСЛИ, МАКС, МИН, ВПР; а также другие методы: транспонирование строк в столбцы и обратно, специальная вставка, распространение формул, вставка и форматирование гистограммы, сортировка и форматирование ячеек.
Также для подсчёта нуклеотидов в геноме и подсчёта частоты каждого из нуклеотидов нами был написан соответствующий скрипт на Python версии 3.7.2.
Для подсчета числа G-квадруплексов, частоты кодонов, кодирующих одну и ту же аминокислоту, и числа k-меров были использовали программы bash (команды 1, 2 и 3 соответственно). Программа для подсчёта паттернов в последовательности (в нашем случае – G-квадруплексов) относится к пакету EMBOSS. В качестве паттерна потенциального G-квадруплекса мы использовали такую последовательность: “GGGN(1,7)GGGN(1,7)GGGN(1,7)GGG”, где N – любой нуклеотид, цифры в скобках – диапазон длины петли между триадами гуанина.
Все исходные материалы (последовательности генома и отдельно белок-кодирующих генов в формате fasta и таблицу, содержащую информацию о генах, которая доступна в сопроводительных материалах – лист genes) мы скачали с базы NCBI (доступ по ссылке).
Общая длина генома Methylomusa anaerophila составляет 4781198 пар оснований.
В геноме обнаруживаются 4301 ген. Из них ровно 4100 генов кодируют белки, 87 кодируют различные типы некодирующих молекул РНК и 114 являются псевдогенами. Из 87 генов, в которых записаны последовательности некодирующих РНК, 70 кодируют транспортные РНК, 11 – рибосомальные, 2 – РНК, содержащиеся в рибонуклеазе P, являющейся рибозимом, 1 – РНК SRP-частицы, 1 – транспортно-матричную РНК и 2 несут информацию о прочих некодирующих РНК (таблица доступна на листе genes_per_types в файле сопроводительных материалов).
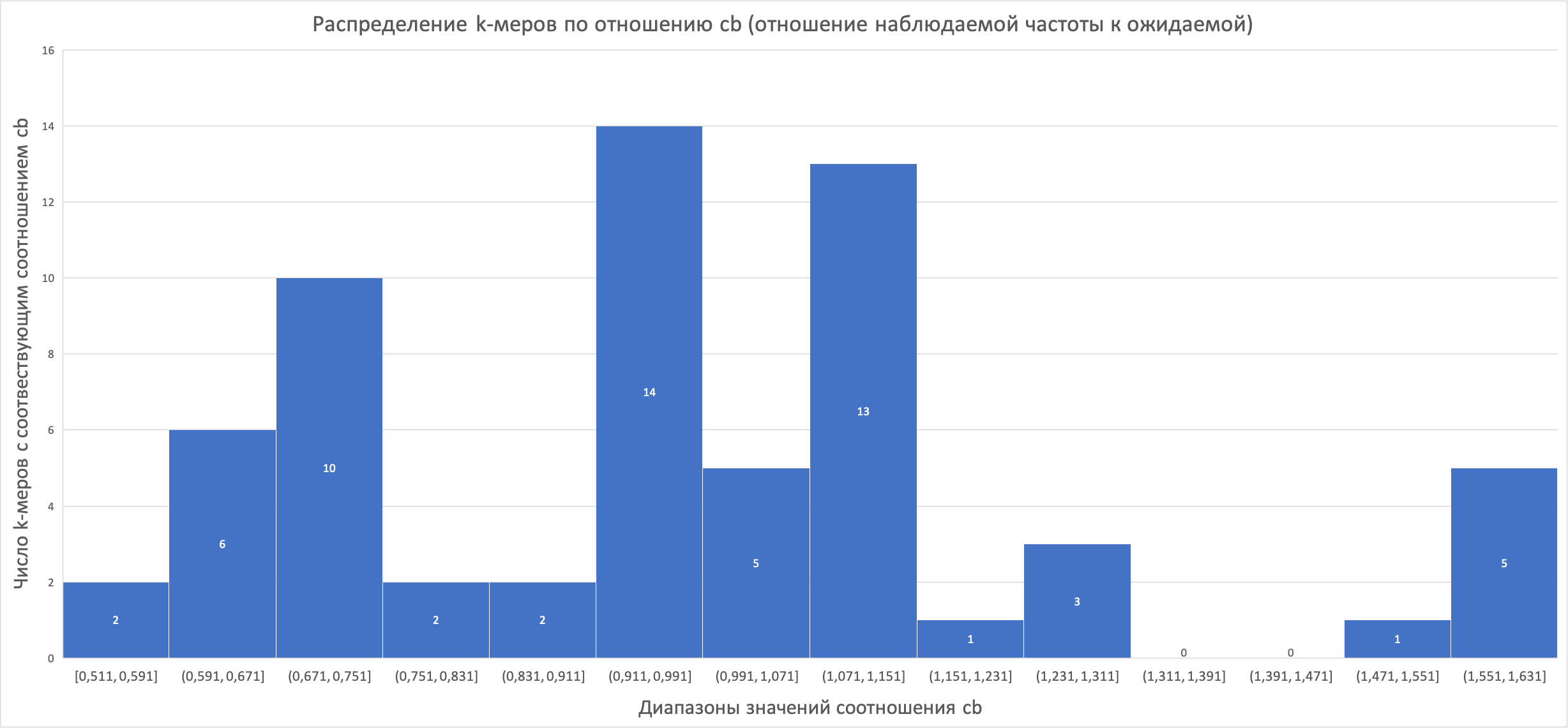
По результатам подсчета k-меров длины в 3 нуклеотида в геноме M. anaerophila мы обнаружили все 64 возможных трехбуквенных слова. После подсчета мы составили таблицу частоты каждого из k-меров (доступна на листе kmer таблицы в сопроводительных материалах neverov_supple.xlsx), по которой посчитали отношение наблюдаемой частоты к ожидаемой (cb). Гистограмма распределения числа k-меров с соответствующим значением cb приведена ниже (рис. 1). Также мы разбили k-меры на три категории по значению cb. Недопредставленными оказались 20 k-меров (cb < 0,8), 10 – перепредставленным (cb > 1,2). Остальные 34 мы отнесли к представленным примерно в соответствии с ожидаемой частотой.
Частота k-меров в геноме организма может служить одним из способов сравнения его с другими организмами, а сравнение количества G-C пар с другими организмами может быть в некоторых случаях косвенным свидетельством того, при каких температурах обитает данный организм.
Итог подсчёта частоты различных кодонов, кодирующих одну и ту же аминокислоту, мы представили в виде таблице, находящейся в полном варианте на листе cusp таблицы в сопроводительных материалах, а её сокращённый вариант – на листе third_nucleotide.
Не для всех аминокислот, представленных в геноме более чем одним кодоном, максимальная и минимальная частоты различаются в одинаковой степени. Так, для аланина, глутамата, фенилаланина, глицина, гистидина, изолейцина, лизина, серина, треонина, валина и тирозина частота кодона, наиболее представленного в геноме, и частота наименее представленного кодона различаются примерно в два раза. В случае же лейцина, пролина и аргинина эти величины различаются примерно в 4 раза, а для цистеина наоборот – практически не различаются.
Сейчас известно, что третья позиция в кодоне (а именно стоящим в 3 позиции нуклеотидом различаются синонимичные кодоны всех аминокислот, кроме серина, в случае которого существует вариативность и первого нуклеотида) является наиболее быстро эволюционирующей из трех (Bofkin & Goldman, 2007). Однонуклеотидная замена в третьей позиции кодона во многих случаях не приводит к изменению аминокислотного состава белка из-за вырожденности генетического кода, поэтому вполне логично, что наблюдается наибольшая вариабельность именно в этой позиции. «Предпочтение» какого-то из кодонов может служить, например, для регуляции синтеза белков, содержащих «редкие» кодоны (представленные в заметно меньшей степени). Из-за более долгого нахождения рибосомы на «редком» кодоне в связи с небольшим количеством тРНК с соответствующим антикодоном, может осуществляться регуляция транскрипции по принципу аттенюации, либо замедляться трансляция каких-то белков, богатых «редкими» кодонами.Всего в геноме нами были обнаружены 118 G-квадруплексов, из которых 56 на прямой (лист quadruplex+ файла сопроводительных материалов) и 62 на обратной (лист quadruplex- файла сопроводительных материалов) цепях ДНК. Гистограммы распределения числа G-квадруплексов по геному для прямой и для обратной цепи приведены ниже (рис. 2, 3).
На прямой цепи G-квадруплексы распределены сравнительно более равномерно, чем на обратной (рис. 2). В карман длиной 50000 нуклеотидов максимум попали 7 G-квадруплексов. В ещё один карман попали 5, а в четыре других – по 4 G-квадруплекса. В целом, заметно больше G-квадруплексов собраны во второй половине цепи.
На обратной цепи ситуация иная (рис. 3). Большая часть G-квадруплексов сконцентрирована в первой трети цепи, при этом наблюдается пик – в один из карманов попало 22 G-квадруплекса. В следующий по высоте столбец, расположенный поблизости к пику, попали 8 G-квадруплексов. Остальные столбцы имеют высоту 3 и меньше. В целом, на обратной цепи G-квадруплексы более сконцентрированы на отдельном участке. Возможно, это связано с тем, что этот участок несёт отдельную регуляторную функцию (т.к. G-квадруплексы могут участвовать в регуляции транскрипции). Либо эти участки защищены от каких-то повреждающих факторов, т.к. сворачивание нити в несколько G-квадруплексов может помочь экранировать какой-либо участок от воздействия белков цитоплазмы или наоборот создать структурный участок с определёнными функциями.
Таким образом, нами был составлен обзор генома бактерии Methylomusa anaerophila, в который вошли широкие темы, такие как размер генома и его состав по продуктам транскрипции генов, а также более узкие темы, связанные с наличием и распределением в геноме k-меров длиной в 3 нуклеотида, G-квадруплексов, а также включающие рассмотрение частот различных синонимичных кодонов.
Файл Excel с сопроводительными сводными таблицами доступен к скачиванию с Google Диска по ссылке.
Мы благодарим преподавателей биоинформатики ФББ МГУ, которыми был составлен курс информатики, в рамках которого был написан данный обзор. И отдельно хотелось бы поблагодарить Андрея Владимировича Алексеевского за помощь и руководство в написании этого обзора.