

Для определения таксономического положения организма, из которого был взят образец из практикума 6 по секвенированию, я использовал blastn с параметрами по умолчанию, кроме размера слова (я брал 7). База данных – nucleotide collection (nt) – базы GenBank, EMBL, DDBJ, PDB, и RefSeq, исключая EST, STS, GSS, WGS и TSA.
При сортировке выдачи по проценту идентичности две верхние находки с параметрами: вес в битах 1195 и 1191, покрытие 91%, процент идентичности равный 100% и столь малым e-value, что оно обозначается 0.0, принадлежат полихете Polycirrus medusa Grube, 1850. Параметры находок позволяют считать, что наш образец, видимо, принадлежит этому виду полихет. Другие (но определённые) виды этого рода также присутствуют в выдаче, но с меньшими значениями параметров (например, процент идентичности 83% и менее, а вес в битах меньше практически в два раза). Фрагменты выдачи с первыми двумя находками (принадлежат Polycirrus medusa) и первой в выдаче находкой другого известного вида этого рода приведены ниже:
Scientific Common Max Total Query E Per. Acc. Description Name Name Taxid Score Score cover Value Ident Len Accession Polycirrus medusa voucher BIOUG(CAN):WS0178 cytochrome oxidase... Polycirrus m... NA 1037304 1195 1195 91% 0.0 100.00 662 GU672524.1 Polycirrus medusa voucher WS0223 cytochrome oxidase subunit 1... Polycirrus m... NA 1037304 1191 1191 91% 0.0 100.00 660 GU672483.1 <...> Polycirrus carolinensis voucher SIO:BIC:A1101 cytochrome oxida... Polycirrus c... NA 1311521 689 689 90% 0.0 83.43 657 JX423769.1
В выдаче так же присутствуют образцы этого вида с чуть меньшим процентом идентичности (98% и 97%), что вызвано 10 и 18 заменами на 658 нуклеотидном участке. Однако эта разница заметно меньше, чем с другими видами этого рода. От точечных замен никто не застрахован, поэтому принадлежность организма мы можем определить с точностью до вида (ссылка на запись в базе Taxonomy).
Судя по нуклеотидному бласту, эта последовательность принадлежит последовательности гена первой субъединицы цитохромоксидазы, 658-нуклеотидный фрагмент которого используется для молекулярной систематики животных и баркодинга.
Для большей уверенности я дополнительно использовал blastx для поиска гомологов в белковой базе SwissProt (параметры по умолчанию, кроме размера слова – я взял 3). При сортировке по проценту идентичности первым в выдаче стоит полипептид 1 цитохром-C-оксидазы дождевого червя (Lumbricus terrestris Linnaeus, 1758), что подтверждает вывод о том, что наша последовательность относится к гену цитохромоксидазы (первая субъединица). Этот белок является частью митохондриальной цепи транспорта электронов, а его ген из-за своей консервативности часто используется для молекулярной филогении, что также добавляет уверенности в определении таксономической принадлежности образца.
Scientific Common Max Total Query E Per. Acc. Description Name Name Taxid Score Score cover Value Ident Len Accession RecName: Full=Cytochrome c oxidase subunit 1; AltName:... Lumbricus te... common earth... 6398 307 307 97% 1e-101 81.28 513 Q34941.1
В качестве неаннотированной последовательности я взял контиг CAJGIK010000069.1 из сборки генома сумчатого организма квокки Setonix brachyurus (Quoy and Gaimard, 1830) с AC: GCA_904810765.1. Для поиска генов белков в этой последовательности я использовал blastx, так как поиск по аминокислотным последовательностям позволяет находить более дальние гомологии из-за более высокой консервативности последовательности белка по сравнению с последовательностью его гена. Я взял базу nr, но ограничил поиск позвоночными (Vertebrata) для его ускорения и исключил таксон Setonix brachyurus. Параметры поиска по умолчанию (если бы не нашлось, можно было бы сделать более мягкий порог на e-value или уменьшить слово, но не понадобилось).
По результатам поиска можно с уверенностью сказать, что участок последовательности между 2 и 6484 нуклеотидами кодирует белок adenomatous polyposis coli. Этот белок является супрессором опухолей. Мутации в его гене приводят обычно к укорачиванию белкового продукта и являются необходимым вводным при раннем развитии аденом и карцином в толстом кишечнике (см. запись в UniProt). Скорее всего мы видим неполный ген, так как последовательность этого белка, например, человека имеет длину 2843 аминокислотных остатка, а первая в выдаче бласта последовательность этого белка, принадлежащая лисовидному поссуму (Trichosurus vulpecula (Kerr, 1792)), имеет длину 2844 остатка, причем около 700 аминоксилотных остатков белка из квокки не хватает с N-конца.
Чтобы найти недостающие куски я взял первые 770 аминокислот этого белка из лисовидного поссума и с помощью tblastn попытался найти гомологичные участки в базе нуклеотидных последовательностей WGS из квокки. К сожалению, удалось восстановить только ещё 118 аминокислотных остатков с N-конца уже имеющегося фрагмента по другому контигу этой же сборки. Видимо, остальная часть оказалась не прочитана при секвенировании. Чтобы это подтвердить, я хотел запустить tblastn против базы SRA квокки, но риды проекта, в рамках которого была сделана эта сборка, я не нашёл, а те, что нашлись, не содержали нужной информации.
Итоговая неполная последовательность гена
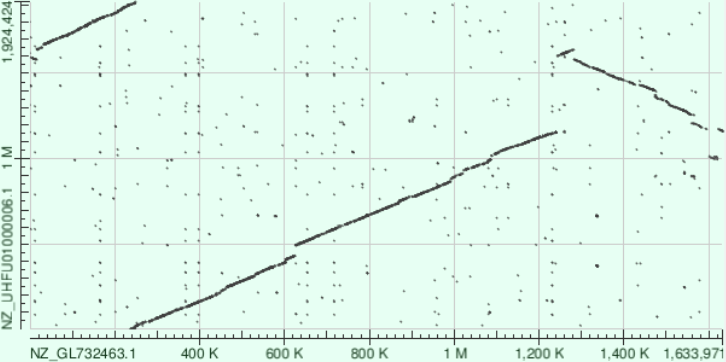
Для построения карты локального сходства я использовал инструмент blast2seq с алгоритмом blastn. Я сравнивал полные геномы бактерий Streptococcus peroris (ось X) и S. infantis (ось Y). На получившемся графике мы видим две глобальные перестройки: делеция в геноме S. peroris на уровне примерно 200 килобаз, а также инверсия участка между 1300 и 1600 килобазами. Также есть небольшая делеция в геноме S. peroris на уровне примерно 630 килобаз.