
Были скачаны протеомы бактерий из практикума 1-2 (кроме LACLM и STAES - они были заменены на LACLA и STAEQ, соответственно). Из них была образована база данных протеинов следующей коммандой:
makeblastdb -in db_input -dbtype prot
Затем я произвел поиск по этой базе данных схожих белков с CLPX_ECOLI при помощи программы blastp:
blastp -db db_input -evalue 0.0001 -query CLPX_ECOLI.fasta -out result
И получил следующий список лучших находок:
Query= sp|P0A6H1|CLPX_ECOLI ATP-dependent Clp protease ATP-binding subunit
ClpX OS=Escherichia coli (strain K12) OX=83333 GN=clpX PE=1 SV=2
Length=424
Score E
Sequences producing significant alignments: (Bits) Value
CLPX_GEOKA Q5KWJ9 ATP-dependent Clp protease ATP-binding subuni... 520 0.0
CLPX_BACSU P50866 ATP-dependent Clp protease ATP-binding subuni... 516 0.0
CLPX_BACAN Q81LB9 ATP-dependent Clp protease ATP-binding subuni... 513 2e-180
CLPX_LISMO Q8Y7K9 ATP-dependent Clp protease ATP-binding subuni... 511 2e-179
CLPX_STAEQ Q5HNM9 ATP-dependent Clp protease ATP-binding subuni... 499 5e-175
CLPX_ENTFA Q833M7 ATP-dependent Clp protease ATP-binding subuni... 498 2e-174
CLPX_LACLA Q9CGE6 ATP-dependent Clp protease ATP-binding subuni... 490 1e-171
Q1GAP8_LACDA Q1GAP8 ATP-dependent Clp protease ATP-binding subu... 475 2e-165
Было реконструировано дерево найденных гомологов (выравнивание последовательностей было произведено программой muscle) в программе MEGAX методом анализа филогении Neighbour-Joining. Параметры запуска не были изменены (стандартные).
Формула Newick:
((((((((CLPX_BACSU:0.06775970,CLPX_BACAN:0.08123326):0.00488624,CLPX_GEOKA:0.07775241):0.03411883,CLPX_LISMO:0.10874450):0.06206534,CLPX_STAEQ:0.18868833):0.02683257,(CLPX_LACLA:0.16866127,CLPX_ENTFA:0.17660868):0.00714998):0.02475819,Q1GAP8_LACDA:0.22539936):0.27599454,((HSLU_LACDA:0.23369464,HSLU_ENTFA:0.23890978):0.04330005,(HSLU_STAEQ:0.25817582,(HSLU_LISMO:0.21560178,(HSLU_GEOKA:0.12531746,(HSLU_BACAN:0.15066389,CLPY_BACSU:0.13199124):0.01002432):0.06943619):0.02447140):0.03741240):0.39945847):0.15288106,Q1GBN8_LACDA:1.01760915,(CLPE_BACSU:0.17150964,(Q1GB74_LACDA:0.28326429,Q8Y8B1_LISMO:0.26530166):0.06838814):0.68766269);

Как можно увидеть из рисунка 1, примерами ортологичных пар белков являются Q1GAP8_LACDA и CLPX_BACSU, HSLU_BACAN и CLPY_BACSU, Q1GB74_LACDA и Q8Y8B1_LISMO.
Также можно заметить, что белками паралогами являются пары CLPX_BACSU и CLPY_BACSU, CLPX_BACAN и HSLU_BACAN, CLPX_GEOKA и HSLU_GEOKA.
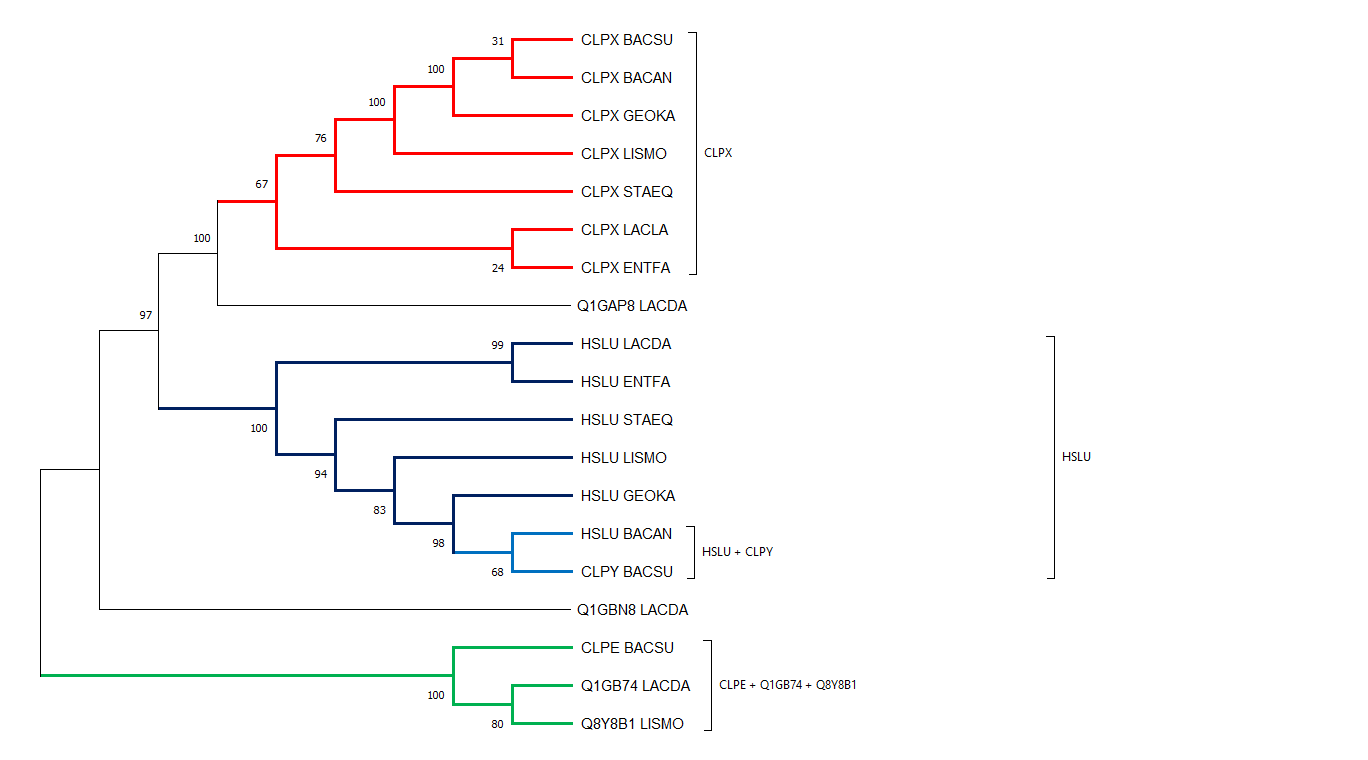
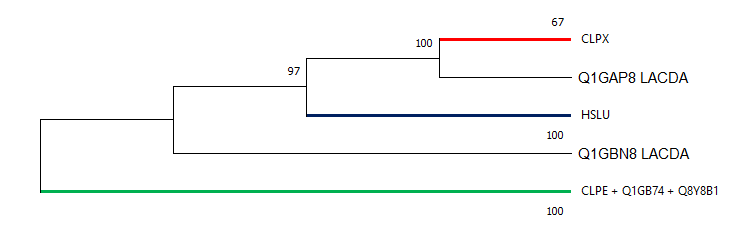
Красная и синяя группы полностью соответствуют филогении бактерий. В зеленой же ветви указано большее родство QBY8B1_LISMO к Q1GB74_LACDA, чем к CLPE_BACSU.
Вернуться на страницу семестра