

Выбранная архитектура включает в себя 2 домена: PF00180 (Iso_dh) - Isocitrate/isopropylmalate dehydrogenase, PF18324 (TT1725) - гипотетический положительнозаряженный домен
| ID домена | AC домена | Название домена | Число последовательностей, содержащих домен (по Uniprot) |
|---|---|---|---|
| Iso_dh | PF00180 | Isocitrate/isopropylmalate dehydrogenase | 70 036 |
| TT1725 | PF18324 | TT1725 hypothetical protein | 1 349 |
Ссылка на скачивание таблицы с результатом поиска записей Uniprot, имеющих выбранную доменную архитектуру. Всего было найдено 1289 таких последовательностей.
Сначала была построена гистограмма для распределения длин белков из таблицы предыдущего задания. Большая часть белков заключена в промежутке между 470 и 500 - будем считать этот интервал длин нормальным для заданной архитектуры.
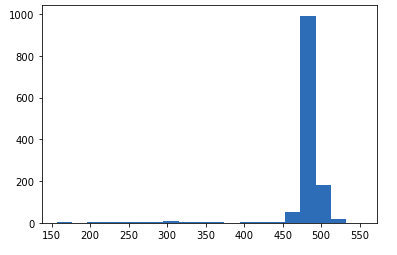
Затем были выбраны 50 белков характерной длины с доменной архитектурой из задания 2.
Ссылка на скачивание файла selected.xlsx с выбранными белками (выбранные белки обозначены значением 'True' в колонке 'selected')
Ссылка на скачивание файла selected.xlsx с только выбранными белками
Запрос Uniprot:
U5C8H3 OR V6I1I3 OR U5BZA9 OR U5WP34 OR M6U6L9 OR M9WNK2 OR Q2GEM9 OR A8GMS0 OR A8F127 OR M9X8B4 OR M6FSE4 OR A3UKF0 OR M9WSH2 OR K6YAD9 OR X7ZCS4 OR S6G8H6 OR B6Y8S9 OR I0AGX4 OR F8KVK0 OR G4KMJ2 OR K8Y1B0 OR I0AFK3 OR G0IWN1 OR I7IAA0 OR M6LJR2 OR I8UC02 OR S7VXZ0 OR J9HAR4 OR I0JWT3 OR M7NTI9 OR H8KCS9 OR F5L777 OR F7SAS2 OR N1UNR6 OR H2CGS7 OR M6CP77 OR K4IHG8 OR T0F2X0 OR M5V0S1 OR X5MLB3 OR M3F173 OR A9DIJ5 OR D7BEE6 OR D7CYB3 OR C0R3B0 OR M7Y1V7 OR H6PSY7 OR M7XHY4 OR H7FUJ6 OR M6Z387
Следующим шагом я выровнял выбранные последовательности с помощью программы muscle. Ссылка на выравнивание.
Перед постройкой HMM-профиля я произвел ревизию выравнивания. В исходном проекте Jalview я удалил позиции 0-36. Также я удалил последовательность X7ZCS4 (предсказанный белок). С C-концом операций не производилось, т. к. мне он показался в достаточной степени консервативным.

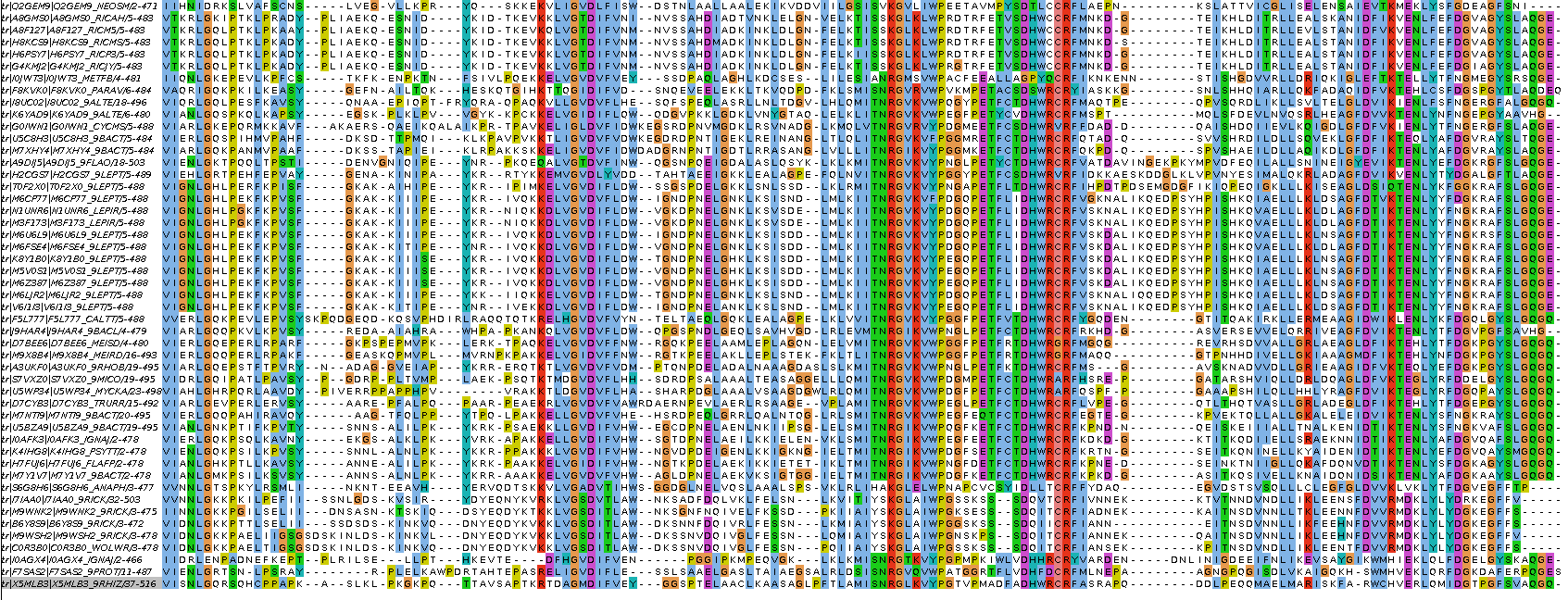
Выравнивание передал на вход следующей команде (построение профиля по выравниванию):
hmm2build -g profile sel_seqs_align.fasta
hmm2build stdout:
hmmbuild - build a hidden Markov model from an alignment HMMER 2.3.2 (Oct 2003) Copyright (C) 1992-2003 HHMI/Washington University School of Medicine Freely distributed under the GNU General Public License (GPL) - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - Alignment file: sel_seqs_align.fasta File format: a2m Search algorithm configuration: Global alignment (hmms) Model construction strategy: MAP (gapmax hint: 0.50) Null model used: (default) Prior used: (default) Sequence weighting method: G/S/C tree weights New HMM file: profile - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - Alignment: #1 Number of sequences: 50 Number of columns: 540 Determining effective sequence number ... done. [9] Weighting sequences heuristically ... done. Constructing model architecture ... done. Converting counts to probabilities ... done. Setting model name, etc. ... done. [sel_seqs_align] Constructed a profile HMM (length 482) Average score: 1224.66 bits Minimum score: 920.06 bits Maximum score: 1366.45 bits Std. deviation: 112.81 bits Finalizing model configuration ... done. Saving model to file ... done. //
Для пересчета веса профиля была вызвана следующая команда:
hmm2calibrate --num 50000 profile
hmm2calibrate stdout:
hmmcalibrate -- calibrate HMM search statistics HMMER 2.3.2 (Oct 2003) Copyright (C) 1992-2003 HHMI/Washington University School of Medicine Freely distributed under the GNU General Public License (GPL) - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - HMM file: profile Length distribution mean: 325 Length distribution s.d.: 200 Number of samples: 50000 random seed: 1586809995 histogram(s) saved to: [not saved] POSIX threads: 8 - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - HMM : sel_seqs_align mu : -485.792480 lambda : 0.118019 max : -430.169006 //
В качестве "опорного" домена был выбран TT1725 (по запросу taxonomy:"Bacteria [2]" database:(type:pfam pf18324)), так как количество белков с этим доменом составляет 1349 (в отличие от его соседа - 70036 результатов). По этим последовательностям был осуществлен поиск по нашему профилю:
hmm2search profile domain.fasta > search_result
Далее средствами языка Python был обработан выходной файл. Ссылка на .ipynb
Для того, чтобы узнать какой вес дает наиболее точное предсказание (все веса больше критического - положительно предсказываемые записи с выбранной архитектурой, не обязательно имеющие ее в реальности) был произведен перебор всех возможных весов (т.е. за критический вес в каждом тесте считалось значение каждого последующего веса из search_result) и были подсчитаны чувствительность и мощность (FDR, 1 - специфичность) тестов. Фактически, была построена ROC-кривая.
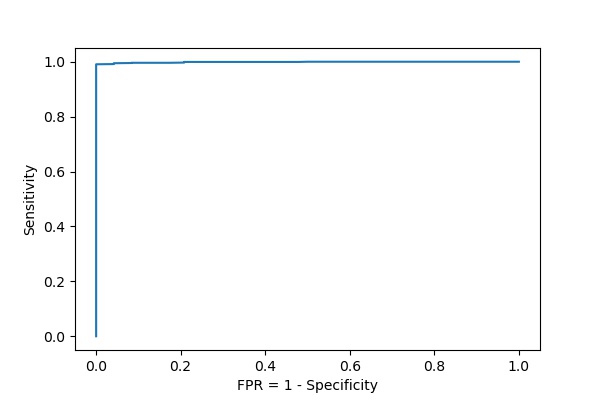
На ее основании был сделан выбор порогового веса, который равняется 37,3. А также была построена так называемая матрица ошибок:
| Positive | Negative | |
|---|---|---|
| Positive | 1272 | 0 |
| Negative | 15 | 24 |
Выбор такого веса был обусловлен тем, что для нашего теста нежелательно производить ложноположительные результаты.
Такую странную ROS-кривую и такой низкий критический вес можно объяснить, как я думаю, тем, что домен TT1725 чаще всего встречается только в составе белков с выбранной мной доменной архитектурой (возможно, функция домена TT1725 раскрывается только в сочетании с доменом Iso_dh).
Вернуться на страницу семестра