Описание сигнала
Для выполнения задания данного практикума было решено выбрать следующий сигнал: точка начала репликации у бактерий (OriC). Многие бактерии имеют одну кольцевую молекулу ДНК и один такой oriC. Это довольно вариабельная область на бактериальной ДНК, которая является множественным сигналом,
у разных видов бактерий ее длина, конкретные мотивы и сайты варьируют (длина в пределах 250 - 2000 п. о.). Тем не менее есть общие закономерности. Сигналы в этой области адресованы различным белкам, ответственным за инициацию репликации. Именно с этой области начинается репликация ДНК у бактерий. Здесь подробно рассмотрим данную область для E. coli, как для известного модельного организма и представителя с наиболее изученным oriC (конкретно у этой бактерии его длина составляет примерно 250 п.о.).
В общем в данной области можно выделить 3 функциональных участка:
- Участки связывания белка DnaA (DnaA-боксы) - сигнальные участки, которые нужны для связывания ключевого инициаторного белка DnaA (белок из семейства AAA+ ATPаз, который умеет образовывать олигомерные структуры и гилролизовать ATP для переключения своего состояния, соответственно ATP-связная форма активна, а ADP-связная неактивна). Представлены основными 2 типами последовательностей: 5 повторов размером
примерно по 9 п.о., это так называемые R-сайты, которые высокоафинны в принципе к DnaA, как к активной, так и к неактивной форме, консенсусная последовательность имеет следующий вид TTATCCACA, а также 3 менее афинных участка, так называемые I-сайты, с которыми связывается хорошо только ATP-связная форма DnaA. Таким образом данные сигналы адресуют сигнал активным формам белка DnaA, которые связываются с ДНК в R- и I-сайтах, образуя олигомерный комплекс, который закручивает спираль ДНК вокруг себя, инициируя репликацию.
- ДНК-расплетающий элемент (DUE) - это A=T богатый участок, это 3 тандемных 13-мерных повтора с консенсусом GATCTNTTNTTTT, разделенных коротким вставками, не богатыми тимином и аденином. Данный участок термодинамически нестабилен (в силу множества слабых двойных водородных связей) и очень восприимчив к сверхспиральному напряжению, которое возникает при образовании олигомерного комплекса DnaA, тем самым при инициации репликации он денатурирует после посадки DnaA, что подает сигнал для посадки других белков, которые уже вместе образуют реплисому (осуществуляющую этап элонгации репликации). На Рис. 1 ниже приведена схема устройства oriC.
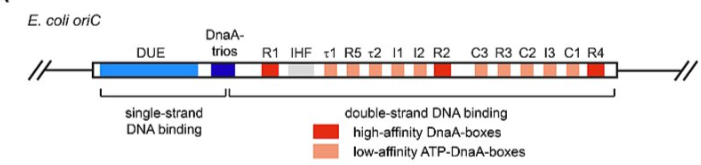 Рис. 1. Схема устройства oriC у E. coli, красным указаны DnaA-боксы, а синим участок DUE.
Рис. 1. Схема устройства oriC у E. coli, красным указаны DnaA-боксы, а синим участок DUE.
- Участки связывания регуляторных белков - помимо DnaA в инициации репликации участвуют многие другие белки и на них в oriC тоже могут быть свои сайты для связывания, которые соответственно адресуют сигналы этим вспомогательным белкам. У E. coli, например, белок IHF (integration host factor) при связывании с одноименном сайтом в oriC облегчает связывание DnaA с I-сайтами, а белок FIS (factor for inversion stimulation), связываясь со своим сайтом в oriC, ингибирует преждевременное соединение ДНК с DnaA. Тем самым данные сигналы регулируют процесс начал репликации. Более подробную схему устройства oriC у E. coli можно посмотреть тут, а более полный список регуляторных белков и их сайтов связывания с oriC - здесь.
Из описанного выше можно заключить, что данный множественный сигнал устроен очень сложно и имеет очень важное значения для бактерий, так как благодаря нему осуществляется один из важнейших процессов в жизнидеятельности организмов - репликация. Из-за такой важности и сложности организации можно предположить, что данный сигнал обладает высокой силой, это также подтверждает тот факт, что несмотря на свою вариабельность среди разных бактерий, область сохраняет какие-то общие черты и паттерны, свойственные большинству бактерий.
Согласно литературным данным (ссылка на источник) конкретно для штамма K12 MG1655 координаты oriC следующие: 3,923,767–3,923,998. Последовательность всего генома этого штамма можно увидеть здесь по данным NCBI. С помощью этого файла
и следующего специализированного скрипта на языке Python была вырезана область oriC у этого штамма, результат можно увидеть здесь (конкретный пример данного множественного сигнала, в начале очень четко видна DUE).
Сервис для поиска выбранного сигнала
В качестве сервиса, который способен искать выбранный сигнал, а именно oriC, был выбран сервис Ori-Finder 2022. Это удобный веб-сервис для поиска oriC в бактериальных геномах с высокой точностью и надежностью, с помощью этой программы были установлены ориджины репликации в более чем сотни недавно отсеквенированных геномах различных бактерий в различных исследованиях.
Данный сервис способен прогнозировать месторасположение oriC в геноме бактерии, красиво визуализирует результаты, а также предоставляет различные инструменты для анализа найденного oriC и удобный интерфейс для взаимодействия. На Рис. 2 изображены схема работы данного сервиса:
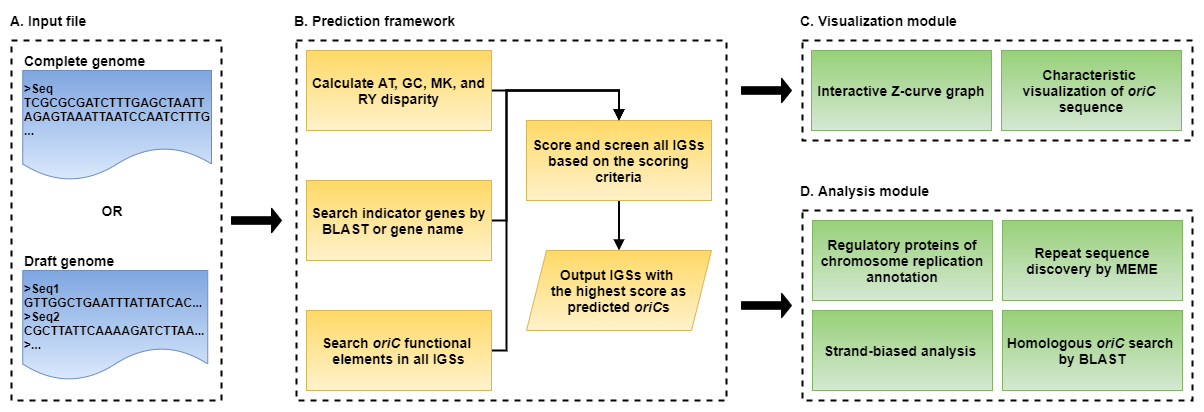 Рис. 2. Схема работы сервиса Ori-Finder 2022 для поиска oriC и его сигнальных сайтов в бактериальном геноме.
Рис. 2. Схема работы сервиса Ori-Finder 2022 для поиска oriC и его сигнальных сайтов в бактериальном геноме.
На вход сервису нужно подать полный или черновой геном в формате fasta или GBK. Для прогнозирования месторасположения oriC сервис оринтируется на несколько факторов:
- Вычисляет GC, AT, MK, RY составы для отдельных участков генома, по ним строит специальную Z-кривую, отражающую последовательность генома (в данном методе минимумы GC-состава отражают возможное расположение oriC).
- Ориентируется на гены-индикаторы oriC (может находить их с помощью встроенного алгоритма BLAST), например, гены DnaA (обычно ориджины расположены рядом с ними). Эти гены можно настроить самому, указав про них дополнительную информацию (тип хромосомы, организм, его систематику), тем самым сделав поиск более эффективным.
- Ищет в последовательности функциональные элементы ориджинов такие как DnaA-боксы, их количество и расположение, сайты метилирования GATC и т.д.
Для проверки работоспособности данного сервиса на вход ему была подана последовательность генома упомянутого в прошлом разделе штамма K12 MG1655 кишечной палочки (координаты oriC которой представлены так же выше). В качестве параметров дополнительно было указано название организма, тип сборки (Complete), в качестве основного гена-индикатора был выбран ген DnaA, в качестве мотива связывания DnaA с oriC был указан также упомянутый в прошлом разделе консенсус TTATCCACA. Сервис отработал достаточно быстро (время ожидания порядка парочки минут). Основной результат выдачи можно увидеть ниже на Рис. 3:
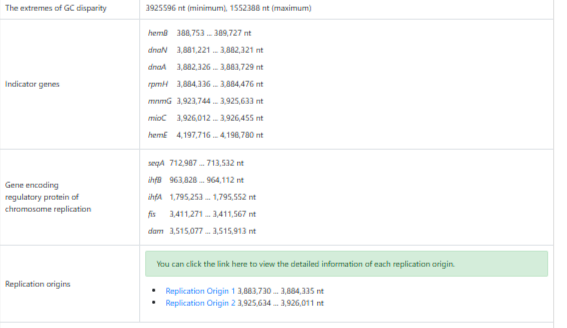 Рис. 3. Результаты работы сервиса для выбранного примера.
Рис. 3. Результаты работы сервиса для выбранного примера.
Видно, что сервис спрогнозировал два участка oriC, причем второй предсказанный участок 3,925,634 ... 3,926,011 оказался достаточно близок к истинному (ошибка на порядка 1500-2000 п.о., истинные координаты 3,923,767–3,923,998). Помимо этого в выдаче можно видеть экстремальные участки GC-состава, а также гены-индикаторы. Помимо этого сервис построил Z-кривую для последовательности генома, которую можно видеть на Рис. 4, минимумы GC-состава на ней (зеленая кривая) соответствуют как раз позиции спрогнозированных oriC.
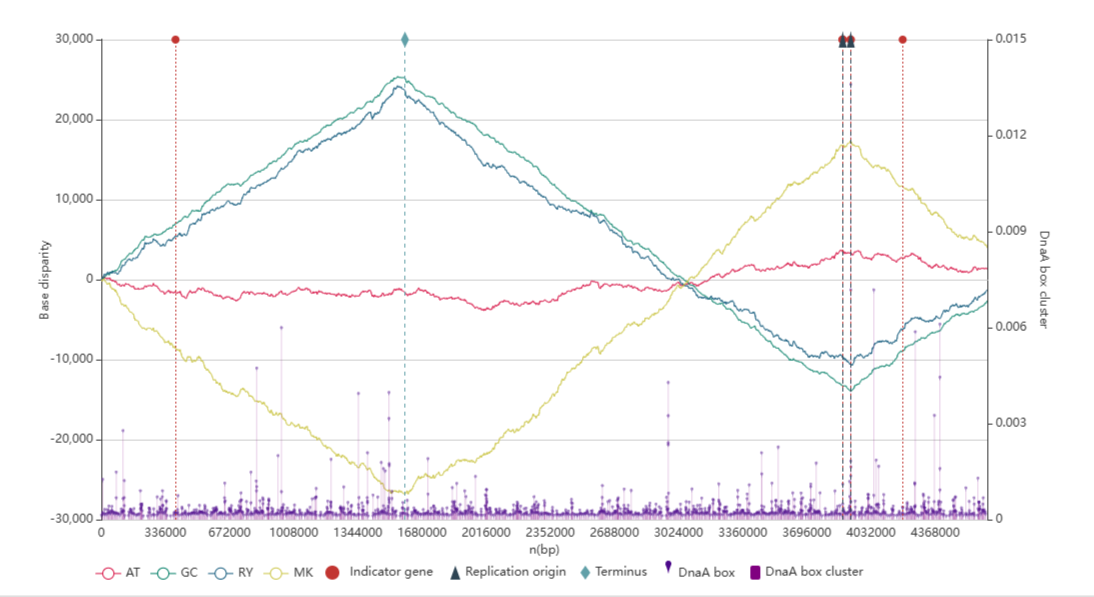 Рис. 4. Z-кривая для генома E. coli K12 MG1655, хорошо видны экстремальные значения GC-состава (зеленая кривая).
Рис. 4. Z-кривая для генома E. coli K12 MG1655, хорошо видны экстремальные значения GC-состава (зеленая кривая).
Также данный сервис выдает саму последовательность спрогнозированного oriC и расположение предсказанных в нем функциональных участков. На Рис. 5 можно видеть это для второго наиболее близкого к истине результата:
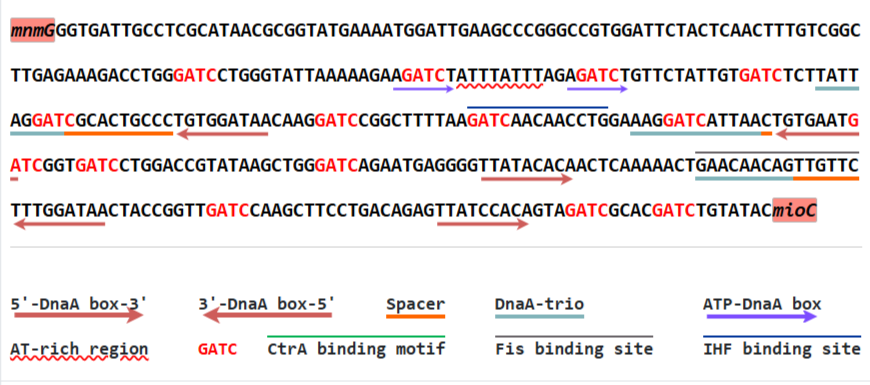 Рис. 5. Последовательность второго спрогнозированного oriC сервисом с отмеченными функциональными участками.
Рис. 5. Последовательность второго спрогнозированного oriC сервисом с отмеченными функциональными участками.
Видно, что сервис сумел предсказать наличие 5 R-сайтов, 3 I-сайта, DUE, IHF, Fis-сайты. Подводя итог, можно сказать, что учитывая размеры геномов, а также саму сложность задачи поиска oriC, данный сервис достаточно хорош, удобен и прост в использованни, выдает много полезной информации, находит oriC достаточно точно.