Практикум 10
Выбранные геномы
Я искала геномы в базе данных RefSeq Nuccore на сайте NCBI по запросу «apicoplast[Title] AND complete[Title] AND genome[Title]».
Я выбрала геномы апикопластов двух паразитических представителей Apicomplexa, Plasmodium malariae (AC: NC_031401.1) и Toxoplasma gondii (AC: NC_001799.1). Апикопласт — это частично редуцированная пластида, утратившая функцию фотосинтеза. Апикопласт — результат вторичного эндосимбиоза с красной водорослью. Так как он (как любая пластида) имеет бактериальное происхождение, он является мишенью антиапикомплексных лекарств (McFadden, 2010).
Геном апикопласта (как и геном других пластид) кольцевой. Некоторые из белков апикопласта закодированы в его геноме, некоторые — в геноме ядерном. В геноме апикопласта закодирована только одна из двух типичных для пластид РНК-полимераз (Nisbet & McKenzie, 2016).
Результат BLAST
Я запускала BLAST на сайте NCBI, указывая как Query ID NC_031401.1, как Subject ID NC_001799.1 и используя параметры megablast и blastn по умолчанию (размеры слов 28 и 11 нуклеотидов соответственно).
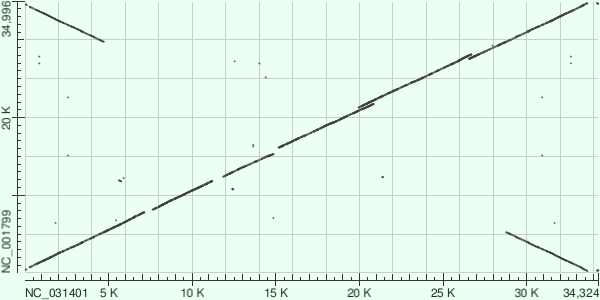
Dot Plot для этих запусков BLAST можно видеть на рисунках 1 и 2.
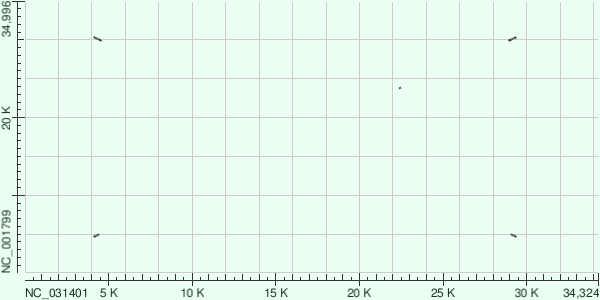
Гены, выровнявшиеся megablast'ом
На рис. 1. можно видеть, что алгоритм megablast построил 5 выравниваний геномов. Два участка генома апикопласта P. malariae выровнялись на два участка генома апикопласта T. gondii. Это свидетельствует о том, что этот участок дуплицировался в геноме когда-то у общего предка этих организмов. Эти дуплицированные участки — участки генов рРНК, что логично: если их нашел алгоритм megablast, они должны быть консервативны.
Еще один участок генома, выровнявшийся megablast'ом, но уже только один раз, кодирует β-цепь РНК-полимеразы у P. malariae и β'-цепь РНК-полимеразы у T. gondii.
Перестройки генома, выявленные blastn'ом
Дупликация в геноме в 29000—5000
На рис. 2. видно, что большие участки геномов (приблизительно 29000..5000 для обоих геномов) выровняны на другой геном дважды, т.е. произошла дупликация участка генома, причем дуплицированные участки находятся в противоположной ориентации.
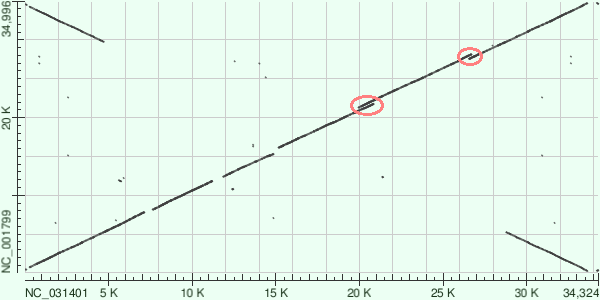
Low compexity regions в ~21000 и ~27000
На рис. 3. можно видеть два региона (обведены красным) неясного происхождения. С одной стороны, это не может быть дупликация (т.к. перекрывания есть в проекциях на обе оси). С другой стороны, повторы должны выглядеть по-другому (должен быть прямоугольник с параллельными линиями).
После я решила посмотреть внимательнее на эти регионы, уменьшив длину слова и отключив фильтр на low complexity regions. Результаты можно видеть на рисунках 4 и 6. Действительно можно видеть, что в этом месте последовательности выравниваются друг на друга много раз, причем в обоих направлениях.

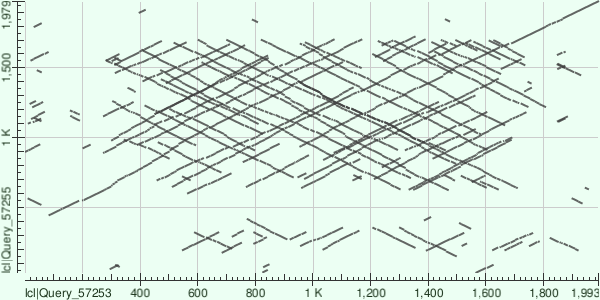
Очень странно, но эти регионы малой сложности в геноме апикопласта P. malariae находятся в белок-кодирующих генах. Регион в ~21000 находится внутри гена rpoD, фактора инициации транскрипции, а регион в ~27000 находится в гене rpoB, кодирующем β-цепь РНК-полимеразы. Я решила выровнять последовательность гена rpoD саму на себя, результат можно видеть на рис. 6. И в этом гене, и в нескольких других генах этого апикопласта (не привожу в отчете) нашлось много выравниваний помимо основного. Когда я запустила тот же алгоритм с теми же параметрами для случайного места генома E. coli, нашлось только одно выравнивание.

Может показаться, что в геномах апикопластов P. malariae и T. gondii просто почему-то очень много повторов, причем иногда палиндромных.
Но, кажется, это не совсем так. Дело в том, что GC-состав апиклпласта P. malariae — 14%, апикопласта T. gondii — 21%. GC-состав гена rpoD апикопласта P. malariae, выровненного на себя на рис. 6., — 8%. Понятно, что при таком большом количестве AT-пар вероятность совпадения двух случайных букв больше, чем при равномерном буквенном составе, и e-value всех находок будет заниженно.
Чтобы проверить эту идею, я перемешала ген rpoD апикопласта P. malariae при помощи инструмента shuffleseq пакета emboss (использовала веб-интерфейс) и выровняла получившуюся последовательность саму на себя; результат можно видеть на рис. 7. Видно, что получившаяся последовательность выравнивается на себя примерно так же, как и нормальный ген, т.е. такое большое количество «побочных» выравниваний возникает из-за смещенного GC-состава.

Иными словами, обсуждаемые аномалии — следствие сниженного GC-состава. Видимо, в апикопласте используется измененный генетический код: иначе внутри аннотированных рамок считывания возникает много стоп-кодонов.
BLAST белкового продукта гена rpoD апикопласта P. malariae
Мне захотелось понять, может ли вообще ген с таким низким GC-составом кодировать работающий белковый продукт. Для этого я взяла из RefSeq транслированную последовательность гена rpoD апикопласта P. malariae и нашла его гомологи про помощи алгоритма blastp с параметрами по умолчанию, исключив из поиска белки Apicomplexa.
В основном нашлись продукты генов пластид красных и зеленых водорослей, но не генов факторов инициации транскрипции, а генов β'-цепей РНК-полимераз.
После я решила выровнять гены rpoD апикопласта P. malariae и rpoD E. coli. Алгоритм blastp не нашел значимых (критическое значение — 0,05) находок; алогритм needle показал, что similarity этих двух последовательностей — 1,1%; алгоритм water нашел участки длиной около 130 а/к с similarity около 40%. Я полагаю, что это неродственные белки, а найденный алгоритмом water участок либо найден по случайности, либо является каким-нибудь ДНК-связывающим доменом или другим доменом, общим для РНК-полимеразы и фактора транскрипции.
Кажется, либо этот ген аннотирован ошибочно, либо почему-то транскрипционным фактором rpoD у E. coli и у P. malariae называются совсем разные вещи.
Список литературы
- McFadden GI. The apicoplast. Protoplasma. 2011 Oct;248(4):641-50. doi: 10.1007/s00709-010-0250-5. Epub 2010 Dec 17. PMID: 21165662.
- Nisbet RE, McKenzie JL. Transcription of the apicoplast genome. Mol Biochem Parasitol. 2016 Nov-Dec;210(1-2):5-9. doi: 10.1016/j.molbiopara.2016.07.004. Epub 2016 Jul 30. PMID: 27485555; PMCID: PMC5404108.