Практикум 15
Подготовка чтений и геномная сборка
Мне достались чтеня с AC проекта SRR4240358.
Я конкатенировала все последовательности адаптеров в один файл, отрезала адаптеры от чтений, триммировала чтения, удалив с конца нуклеотиды с качеством менее 20, и отфильтровала, оставив только чтения не менее 32 нуклеотидов. После я проиндексировала все k-меры длиной 31 и собрала на их основе геном. Код привожу ниже.
cat ../../adapters/* >> ./all_ad.fa TrimmomaticSE -phred33 SRR4240358.fastq.gz without_ad.fq.gz ILLUMINACLIP:all_ad.fa:2:7:7 TrimmomaticSE -phred33 without_ad.fq.gz fin_trim.fq.gz TRAILING:20 MINLEN:32 velveth velv_dir 31 -fmtAuto -short fin_trim.fq.gz velvetg velv_dir/
На первом этапе очистки было оставлено 10 368 884 из 10 543 839 ридов (174 955 (1.66%) удалено).
На втором этапе очистки было оставлено 8 016 437 из 10 368 884 ридов (2 352 447 (22.69%) удалено).
До обработки файл весил 493 Мб, после первой очистки — 485 Мб, после второй — 257 Мб.
Результат сборки
Файлы, полученные в результате сборки, можно скачать по ссылкам: файл с контигами и файл со статистикой.
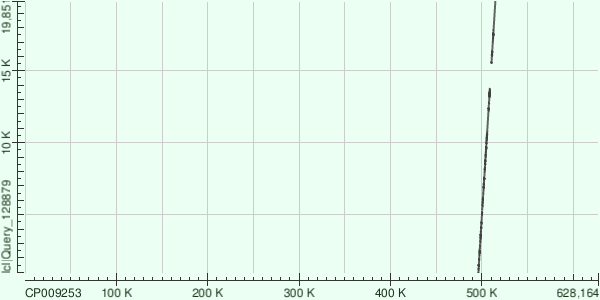
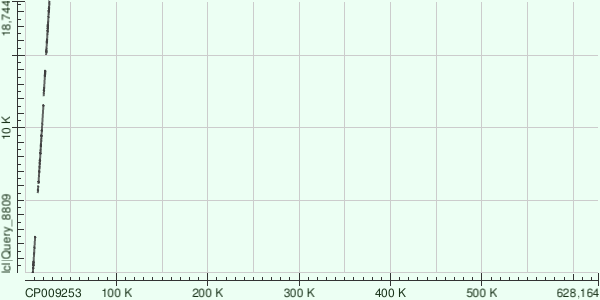
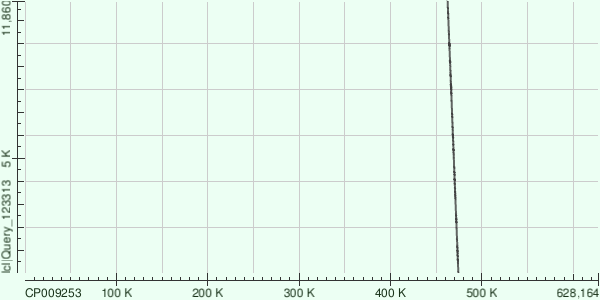
N50, выданный velvetg, равен 8600 (в k-мерах, видимо, а в нуклеотидах 8630). Длины трех самых длинных контигов — 19821, 18714 и 16436 (в нуклеотидах 19851, 18744 и 16466). Их покрытия — 29,5, 29,9 и 30,8.
Среди контигов длиной более 1 k-мера были контиги с покрытием от 1 до 553. Не все контиги из файла со статистикой есть в файле с контигами (по информации из мануала, в этот файл входят только контиги длиной более 2k).
Плюс-минус длинные контиги имели покрытие около 30. Я выбрала три контига длиной более 1 k-мера с наибольшим покрытием, чья последовательность была в файле с контигами. Это были контиги с ID 18, 97 и 129 (их длины — 60, 53 и 129 k-меров, покрытия — 412, 405 и 333). Я выровняла алгоритмом blastn последовательности этих контигов на хромосому Buchnera aphidicola. Контиг с ID 18 выровнялся 1 раз, два остальных — более одного раза.
Три самых длинных контига имели ID 56, 34 и 40, их длины — 19821, 18714 и 16436 k-меров. Результаты выравнивания при помощи megablast привожу в таблице 1.
| ID | Длина контига в нуклеотидах | Количество выравниваний megablast | Длины выравниваний | Число различий нуклеотидов | Число гэпов | Координаты участка хромосомы |
|---|---|---|---|---|---|---|
| 56 | 19851 | 3 | 8614, 4396 и 4325 | 1756, 733 и 912 | 345, 83 и 156 | 500370 — 508806, 510438 — 514772 и 496111 — 500325 |
| 34 | 18744 | 6 | 2220, 3781, 3228, 2530, 1850 и 478 | 294, 702, 683, 488, 293 и 77 | 30, 144, 92, 60, 49 и 9 | 17962 — 20171, 23067 — 26764, 14727 — 17919, 8599 — 11103, 20358 — 22183 и 13994 — 14465 |
| 40 | 16466 | 2 | 6962 и 5019 | 1412 и 991 | 206 и 164 | 467412 — 474242 и 462496 — 467424 |
На рис. 1, 2 и 3 можно видеть dot plot'ы результатов выравнивания контигов с ID 56, 34 и 40 на геном Buchnera aphidicola.
Вообще довольно странно, что контиги настолько непохожи на геном: и ложатся они с промежутками, и идентичность внутри одного выравнивания около 75%, хотя принадлежат они тому же виду.