Практикум 4
Стебли тРНК
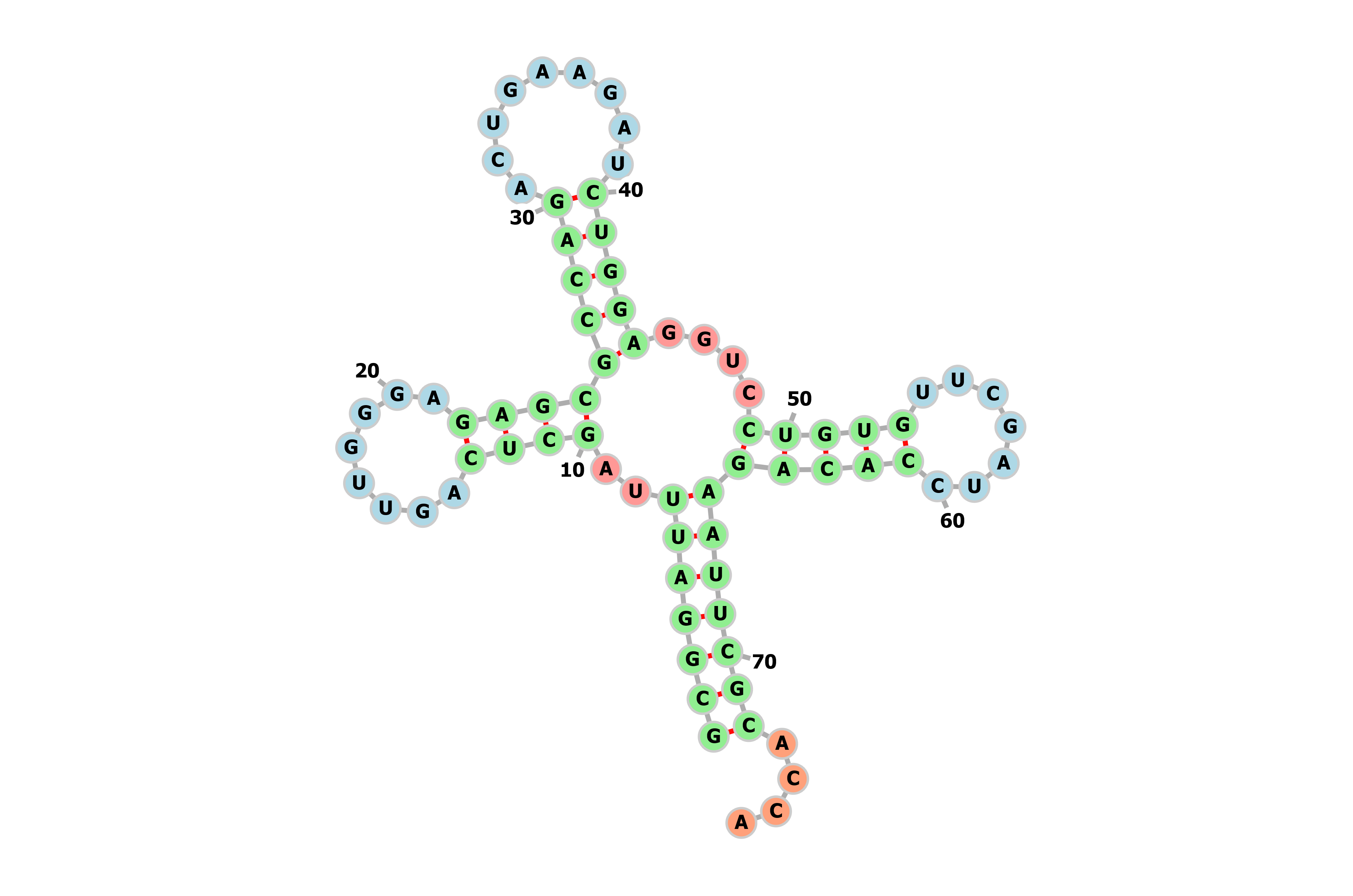
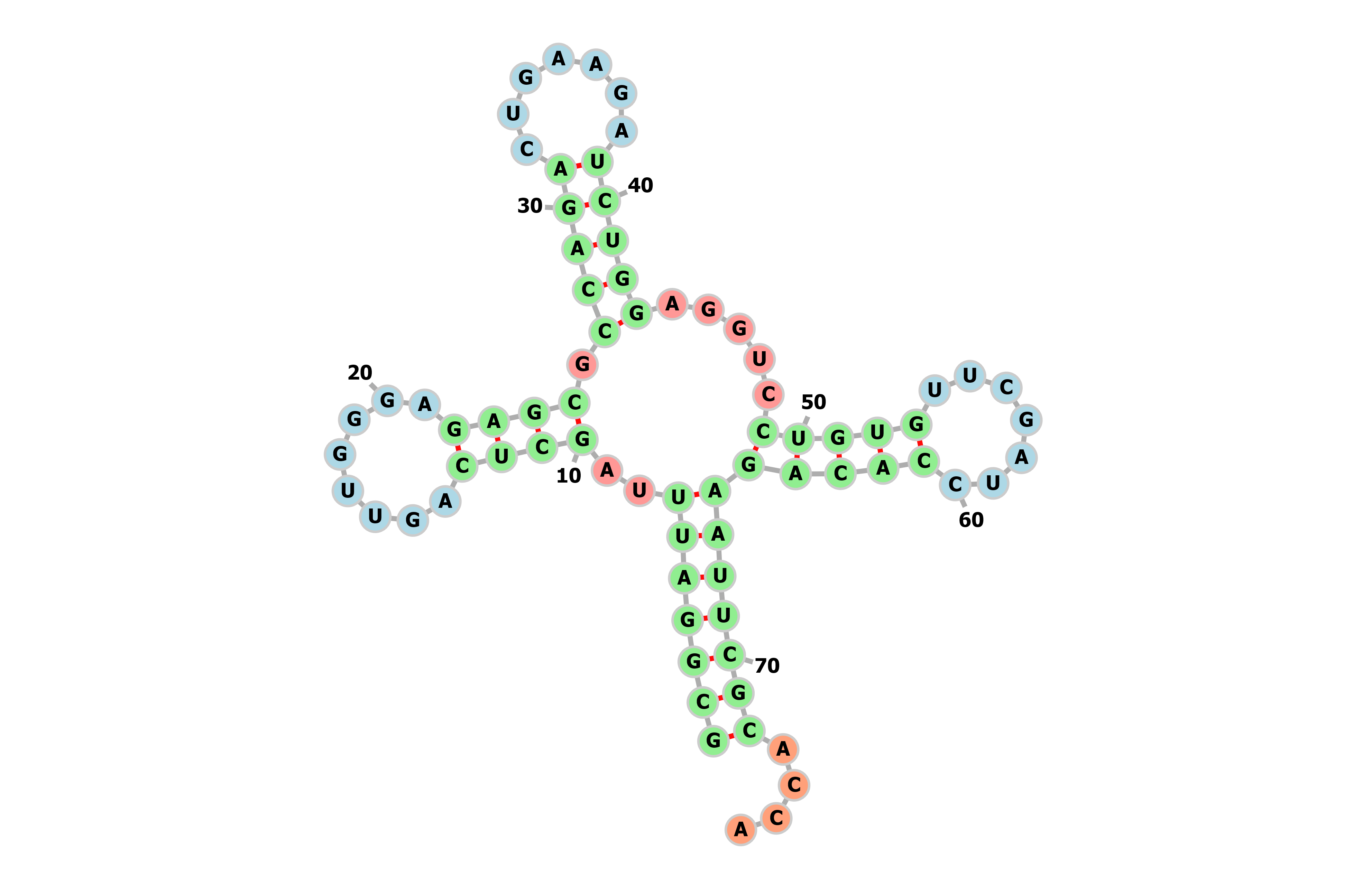
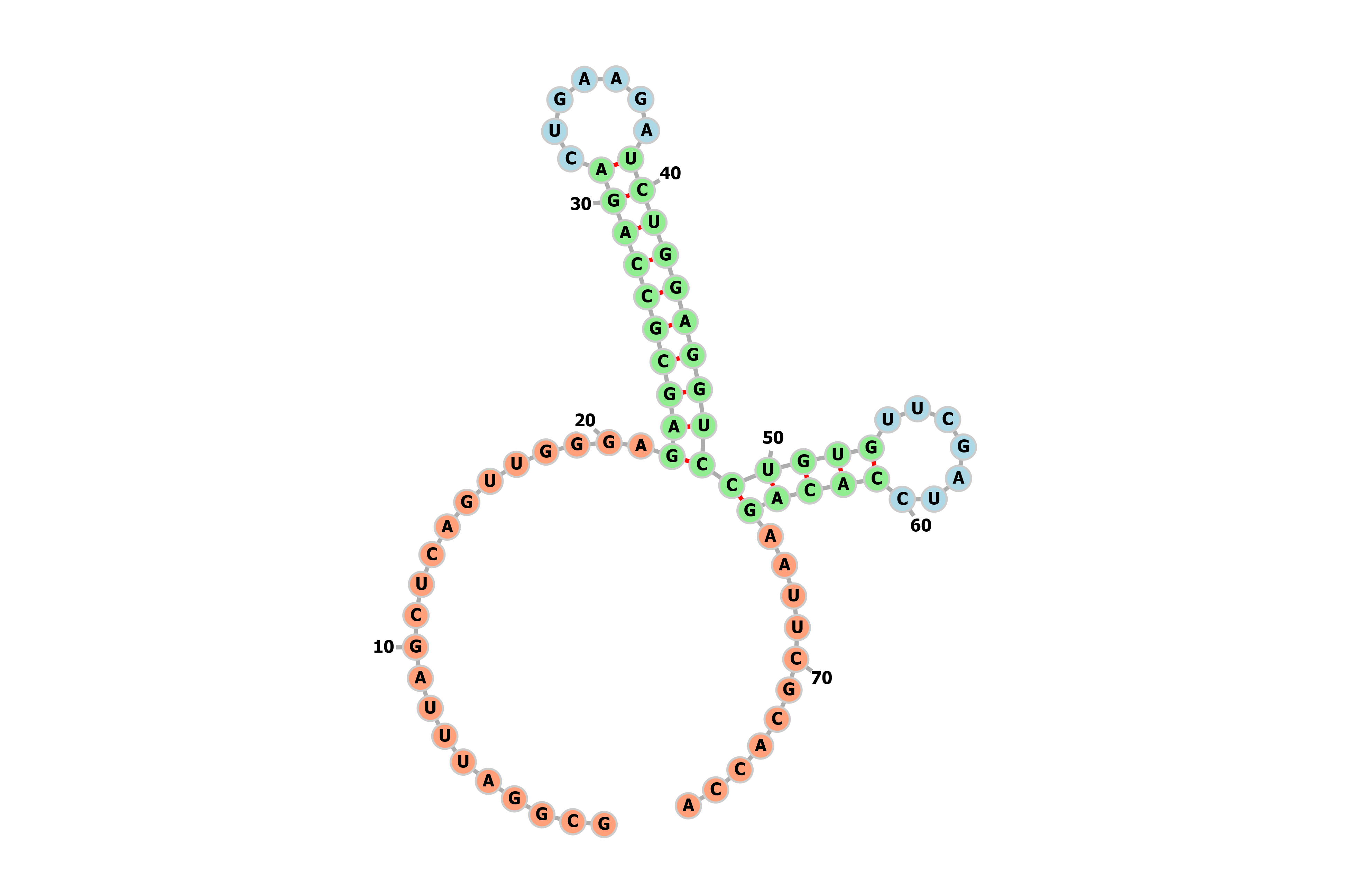
В таблице 1 указаны стебли тРНК с PDB id 1i9v, найденные при помощи ViennaRNA и einverted и найденные в 3D-структуре этой тРНК.
| Стебель | Из 3D-структуры | Из ViennaRNA | Из einverted |
|---|---|---|---|
| Акцепторный | 1 — 7 и 72 — 66 | 1 — 7 и 72 — 66 | — |
| T | 10 — 13 и 25 — 22 | 10 — 13 и 25 — 22 | — |
| Антикодоновый | 40 — 44 и 30 — 26 | 39 — 43 и 31 — 27 | 39 — 48 и 31 — 22 |
| D | 49 — 53 и 65 — 61 | 49 — 53 и 65 — 61 | 49 — 53 и 65 — 61 |
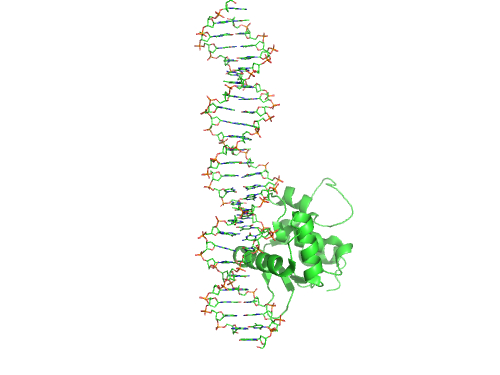
Я визуализировала полученные структуры при помощи веб-интерфейса ViennaRNA. Результаты можно видеть на рис. 1, 2 и 3. Видно, что алгоритм ViennaRNA предсказывает структуру лучше, чем einverted.
Упражнение 1
Поскольку в PyMol нельзя писать скрипты, показывающие изображения с паузами, я написала скрипт, который сохраняет к пользователю пять нужных картинок. Скрипт можно скачать по ссылке. Картинки привожу на рис. 4, 5, 6, 7 и 8.




Упражнение 2
В таблице 2 можно видеть, количество разных «контактов» белковой цепи B с ДНК. Полярным контактом считались атомы азота или кислорода, расположенные на расстоянии не более 3,5 ангстрем, неполярным — атомы углерода, фосфора и серы на расстоянии не более 4,5 ангстрем.
Во-первых, можно видеть, что неполярных контактов просто больше. Это логично, поскольку атомов углерода (считающихся неполярными) просто больше всего.
Во-вторых, контактов со стороны большой бороздки больше, чем со стороны малой. Это тоже логично: белок «сидит» в большой бороздке, если посмотреть на структуру.
| С остатками 2'-дезоксирибозы | С остатками фосфорной кислоты | С остатками азотистых оснований со стороны большой бороздки | С остатками азотистых оснований со стороны малой бороздки | |
|---|---|---|---|---|
| Полярные | 3 | 11 | 5 | 4 |
| Неполярные | 16 | 18 | 24 | 1 |
Упражнение 3
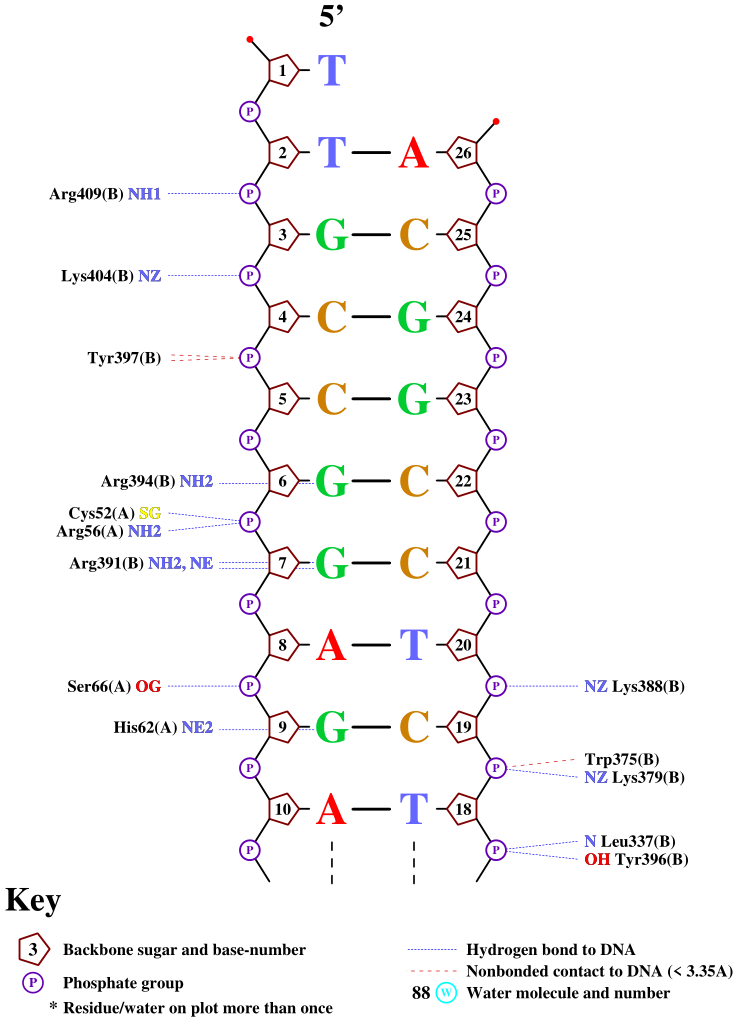
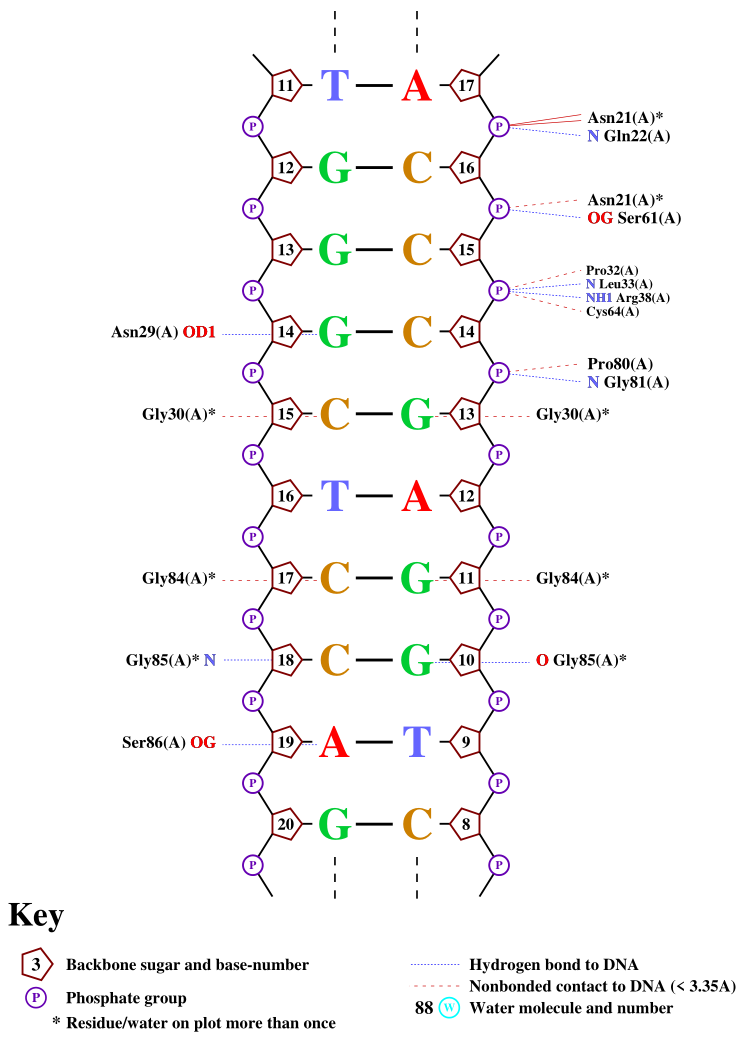
На рис. 9, 10 и 11 можно видеть визуализированные при помощи nucplot контакты ДНК с белками. Nucplot был запущен с парметрами по умолчанию, pdb-файл не содержал информацию о молекулах воды.
Обратите внимание, что тут отмечены контакты и с A-, и с B-цепью белка, тогда как ранее я работала только с B-цепью.
Упражнение 4
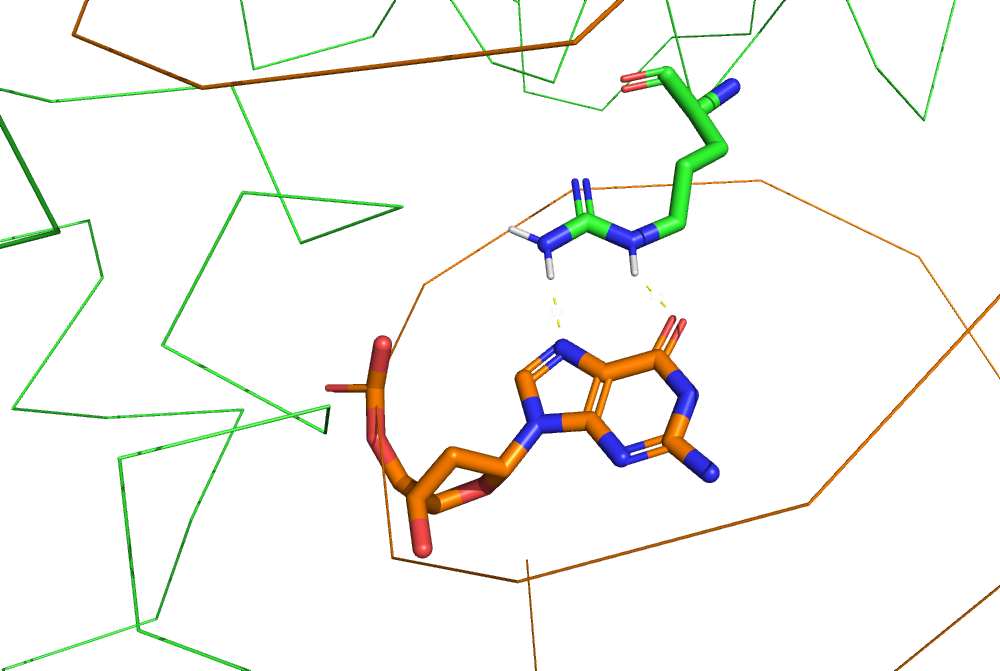
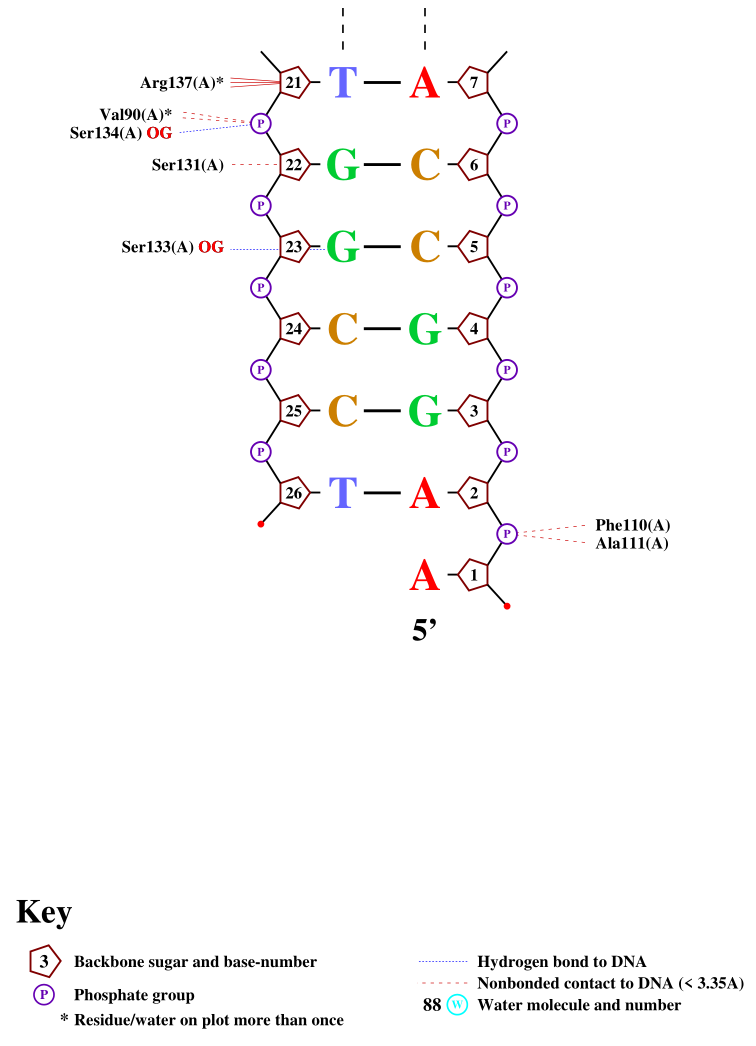
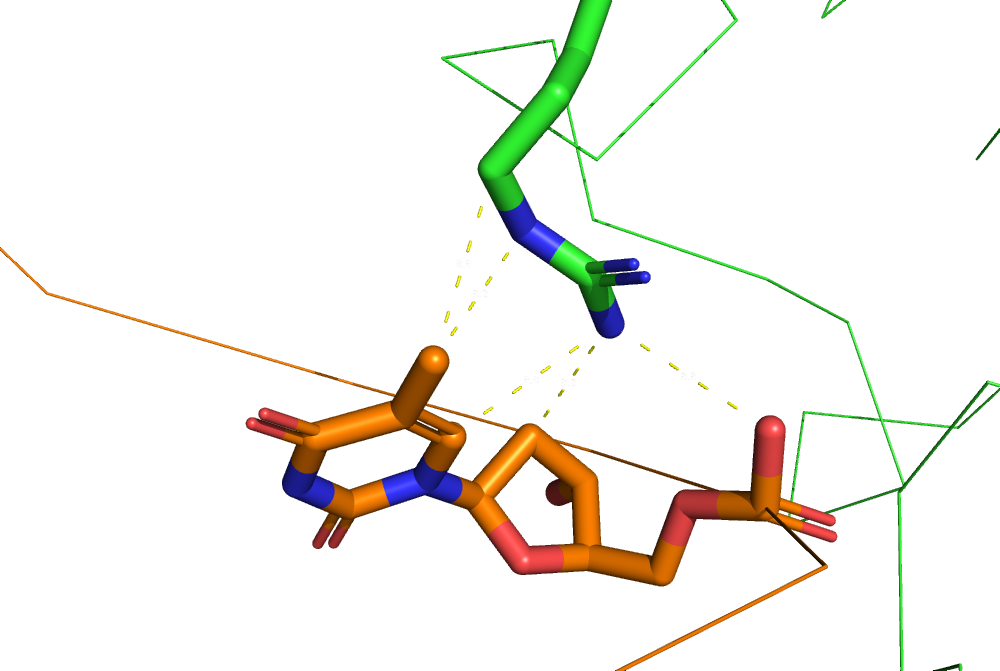
Больше всего контактов с ДНК образуют остатки аргинина-137 и аспарагина-21 из белковой цепи A. Их контакты с ДНК изображены на рис. 12 и 13.
Почему-то PyMol (с той же границей — 3,35 ангстрем) нашел не три контакта аргинина-137 с ДНК (как nucplot), а пять. Возможно, nucplot считает не количество контактов (т. е. количество пар атомов), а количество атомов аминикислоты, вступающих в контакт.
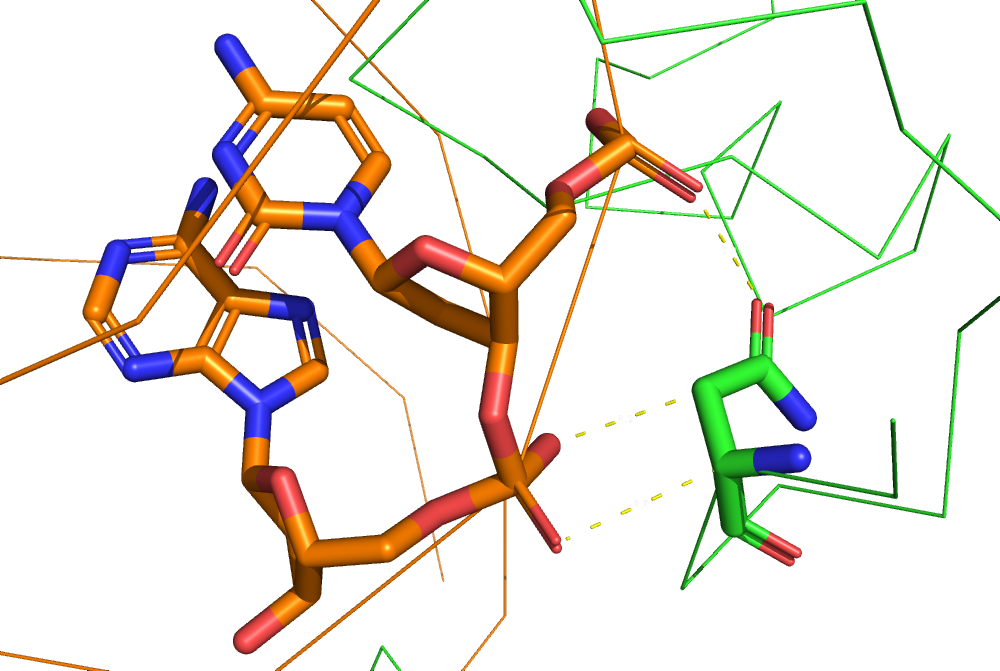
Чтобы найти «наиболее важный» для распознавания ДНК аминокислотный остаток, я выбрала остатки, образующие более одной водородной связи с ДНК (водородные связи более специфичны, чем гидрофобные, наверное). Таких оказалось два: аргинин-391 в белковой цепи B и глицин-85 в белковой цепи A. Я выбрала аргинин, посчитав более «важным» остаток, образующий водородные связи при помощи радикала. Связи аргинина-391 белковой цепи B с ДНК можно видеть на рис. 14.