Практикум 10
Поиск мотивов в домене PF13864
Для анализа я выбрала домен PF13864, Enkurin. Это кальмодулин-связывающий домен, своим C-концом связывающийся с кальциевыми каналами, а своим N-концом — с белками с доменом SH3[1].
Я скачала его seed выравнивание и убрала последовательности, идентичные на 65% и выше. Количество последовательностей уменьшилось со 100 до 36.
Я искала в выравниваении паттерн с N-конца, имеющее паттерн G[RK][IVLA]P.Y[IVLM]. Все находки (20 штук) оказались на N-конце (см. рис. 1).

Чтобы найти этот мотив в других белках, я осуществила поиск по SwissProt при помощи инструмента ScanProsite. Нашлось 203 находки в 202 последовательностях. Выровненные при помощи MAFFT эти последовательности можно скачать по ссылке. Я оставила послдедовательности с идентичностью в 98% и ниже, их осталось 52. Я нашла в Jalview тот же мотив в этом выравнивании. Очевидно, в каждой последовательности будет хотя бы одна находка. На рис. 2 можно видеть, что около половины из них находятся в одном месте выравнивания.
Это свидетельствует в пользу того, что во всех этих последовательностях правда есть этот домен.

Поиск мотивов в одной ветви дерева
Я взяла все выравнивание seed из прошлого задания и в Jalview построила дерево при помощи алгоритма NJ. Результаты можно видеть на рис. 3.
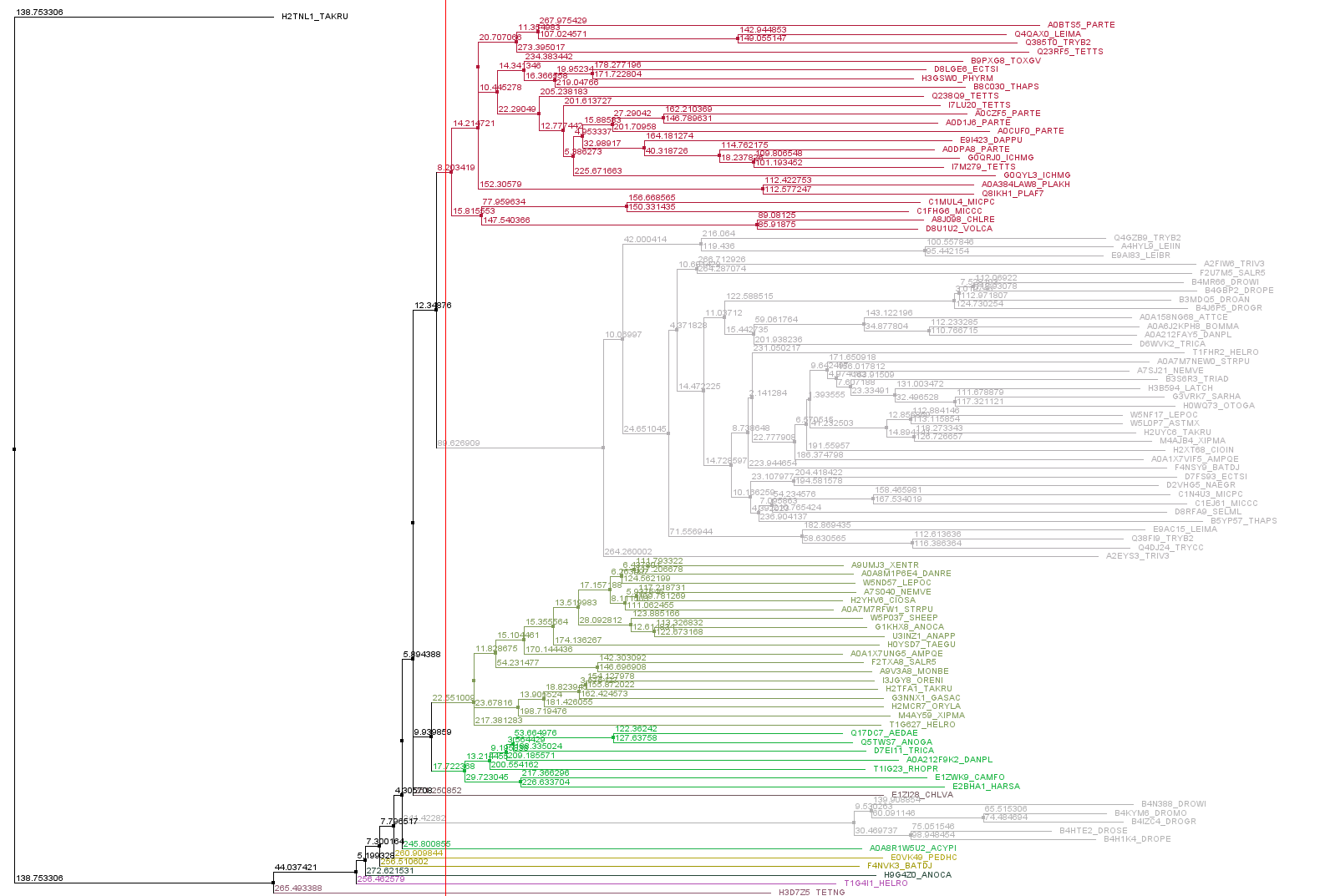
Для поиска мотива я взяла группу, отмеченную красным на рис. 3. Я выбрала мотив [RK]{2,3}E..E..L, он встречается в белках из этой ветви 14 раз, все находки находятся друг под другом (рис. 4).
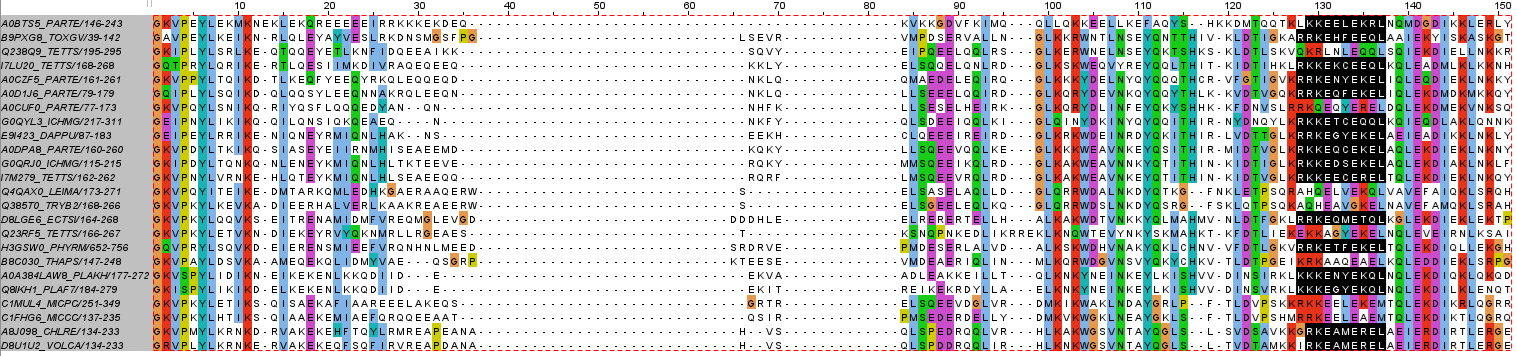
Поиск этого мотива по всему выравниванию выдал 19 находок, 3 из которых, соответственно, принадлежали не этой ветви, при этом одна из 3 была в том же месте, а еще 2 — в другом. Видимо, этот мотив не до конца специфичен для этой ветви и этого места.
PSI-BLAST
Для PSI-BLAST я выбрала белок с AC P19954. Это связывающийся с рибосомой фактор, регулирующий синтез белка в зависимости от света и темепратуры. Принадлежит этот белок шпинату, Spinacia oleracea. Я запустила PSI-BLAST для этого белка с поиском по Swiss-Prot и остальными параметрами по умолчанию. Результаты итераций можно видеть в табл. 1.
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
|---|---|---|---|---|---|
| 1 | 17 | P30334.1 | 0.004 | P19954.2 | 0.0 |
| 2 | 28 | P9WMA8.1 | 0.003 | P19954.2 | 5 * 10-154 |
| 3 | 28 | P9WMA8.1 | 7 * 10-13 | P19954.2 | 4 * 10-147 |
Можно видеть, что схождения мы добились и что, видимо, найденные белки принадлежат одному семейству. Даже с поправкой на множественные сравнения после третьей итерации все находки остаются значимыми.
Все находки, кроме последней, имеют в названии «Ribosome factor», последняя же находка называется «Dormancy associated translation inhibitor». Вообще, видимо, так как ингибиторы трансляции тоже связываются с рибосомой, это белок со схожей функцией, но если предположить, что он из другого семейства, скачаок в e-value между правильными и неправильными находками составляет более 8 порядков.
Поиск мотивов при помощи MEME и FIMO
Для того, чтобы найти мотивы MEME в этом домене, я скачала последовательности белков, в архитектуре которых есть только этот домен. Для тренировки MEME я взяла белки со свидетельством существования «Protein level» или «Transcript level» (суммарно 60 последовательностей, скачать можно по ссылке), для теста взяла белки с предсказанным существованием (3281 последовательность, скачать по ссылке).
Для поиска в MEME и FIMO я запустила на kodomo следующие команды
meme idmapping_existence_1_and_2_2024_05_21.fasta -o results -mod anr -minw 4 -maxw 8 -nmotifs 4 fimo results/meme.txt idmapping_existence_4_2024_05_22.fasta
Таблицу с результатами FIMO можно скачать по ссылке.
Результаты MEME и FIMO можно видеть в таблице 2.
| Мотив | MEME-1 | MEME-2 | MEME-3 | MEME-4 |
|---|---|---|---|---|
| Изображение | 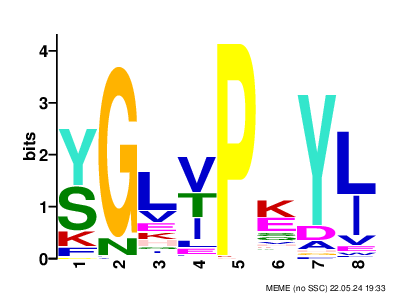 |
 |
 |
 |
| E-value | 1.6 * 10-116 | 5.1 * 10-67 | 3.6 * 10-40 | 1.7 * 10-36 |
| Количество находок q-value < 0,05 |
4225 | 3968 | 2683 | 2287 |
Ожидаемое число случайных находок с q-value < 0,05 — 0, поэтому, видимо, все эти мотивы действительно характерны для этого домена.
Список литературы
- Sutton KA, Jungnickel MK, Wang Y, Cullen K, Lambert S, Florman HM. Enkurin is a novel calmodulin and TRPC channel binding protein in sperm. Dev Biol. 2004 Oct 15;274(2):426-35. doi: 10.1016/j.ydbio.2004.07.031. PMID: 15385169.