Практикум 11
Подготовка данных
Для анализа я выбрала домен PF00104, Ligand-binding domain of nuclear hormone receptor. Это домен ядерных рецепторов с большим количеством альфа-спиралей, связывающийся с молекулой лиганда.
Я взяла белки с доменной архитектурой PF02166 - PF00105 - PF00104, Androgen receptor - Zinc finger, C4 type (two domains) - Ligand-binding domain of nuclear hormone receptor. Белков с такой архитектурой 251, их послдедовательности можно скачать по ссылке.
Я выровняла эти белки при помощи программы MAFFT, вырезала три домена по координатам, указанным в PFAM (6 — 860 для белка P15207). Вырезанные домены я заново выровняла при помощи MAFFT, затем отредактировала выравнивание: удалила последовательности с «X» и последовательности с большими делециями в доменах, а также высокосходные (98% и более) последовательности. Полученное отредактированное выравнивание можно скачать по ссылке.
Для поиска профилей я создала два файла с последовательностями: файл с 93 последовательностями с той же струкутрой, не вошедших в материал для построения профиля, и файл со 118 последовательностями со структурой PF14046 - PF14046 - PF14046 - PF14046 - PF00104 для отрицательного контроля.
Построение и поиск профилей HMM
Я запустила на kodomo следующие комады:
hmm2build hmm_build_output pr11_new_alignment_3_domens_edited.fasta hmm2calibrate --cpu=1 hmm_build_output hmm2search --cpu=1 hmm_build_output pr11_unused_for_test.txt > hmm_search_output hmm2search --cpu=1 hmm_build_output pr11_neg_control.fasta > hmm_search_output_neg
Полученные файлы для положительного и для отрицательного контролей можно скачать по соответствующим ссылкам.
В 93 из 93 последовательностях положительного контроля (последовательности белков в той же структурой) были найдены находки паттерна, в каждой по одной. У всех их них, кроме пяти, e-value был равен машинному нулю, а самый большой e-value был равен 2,1 * 10-163. В 8 из 118 последовательностях отрицательного контроля (последовательности белков с другой архитектурой) тоже были находки (по одной в каждой последовательности). Их e-value находились в пределах от 1,9 до 9,8.
На гистограмме на рис. 1. можно видеть распределение весов находок для положительного и отрицательного контролей. Здесь и далее использовала скрипт, лежащий в Google colab и принимающий на вход вручную преобразованные в csv результаты hmm2search.
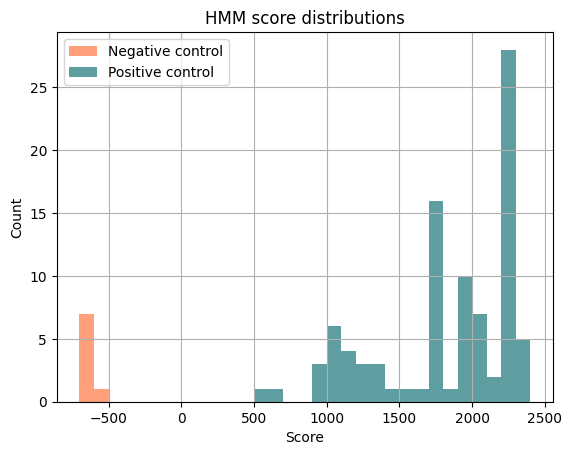
Видно, что распределения весов находок вообще не пересекаются. Логично взять за прог веса 0, тогда и чувствительность, и специфичность будут равны 1 (в нашем тесте)
На рис. 2 и 3 можно видеть кривую F1 и кривую ROC. Кривая ROC не очень информативна, поскольку распределения весов не пересекаются, и хоть какая-то вероятность ложноположительного результата возможна только при отсутствии вероятности ложноотрицательного результата. На кривой F1 можно видеть оптимальные пороги веса — где-то с -500 до 500.
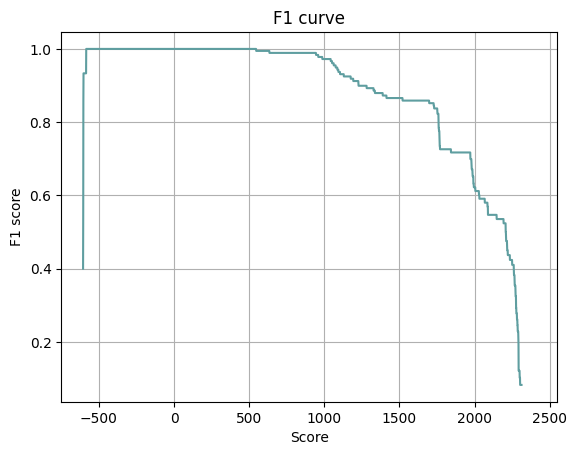
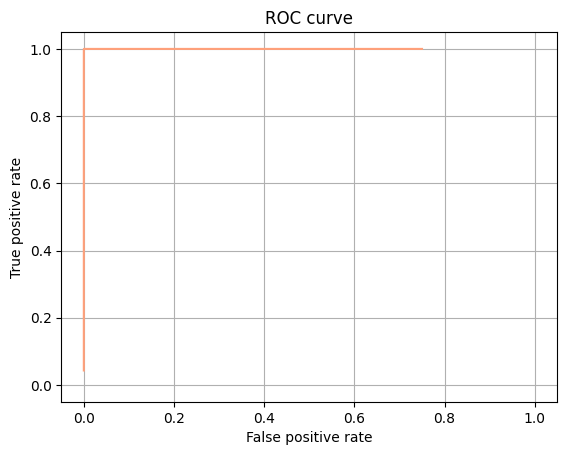
Скорее всего, такие хорошие результаты получаются, поскольку находок в последовательностях отрицательного контроля мало. Впрочем, это тоже хорошо: значит, у hmm2search большая специфичность.