Практикум 4
Запуск blastp
Я проиндексировала геномы выбраных бактерий, сделав из них базы для blastp. После я искала в них гомологи при помощи blastp с длиной слова 2 и порогом на e-value 0,001. Пример кода для одной бактерии привожу ниже, результаты — в таблице 1.
makeblastdb -in AGRFC.fasta -dbtype prot -out AGRFC_db
blastp -db AGRFC_db -query CLPX_ECOLI -out results_BRUSU -evalue 0.001 -word_size 2
| Организм | ID | Score (bits) | E-value |
|---|---|---|---|
| Agrobacterium fabrum | CLPX_AGRFC | 596 | 0.0 |
| Agrobacterium fabrum | HSLU_AGRFC | 92 | 4e-21 |
| Agrobacterium fabrum | Q7CT50_AGRFC | 85.5 | 6e-19 |
| Agrobacterium fabrum | Q7CT50_AGRFC | 45.4 | 7e-06 |
| Agrobacterium fabrum | A9CKX5_AGRFC | 39.7 | 3e-04 |
| Agrobacterium fabrum | RUVB_AGRFC | 39.3 | 4e-04 |
| Agrobacterium fabrum | A9CJ23_AGRFC | 38.5 | 9e-04 |
| Brucella suis | CLPX_BRUSU | 586 | 0.0 |
| Brucella suis | HSLU_BRUSU | 90.9 | 5e-21 |
| Brucella suis | A0A0H3GCZ6_BRUSU | 45.4 | 4e-06 |
| Brucella suis | RUVB_BRUSU | 39.3 | 2e-04 |
| Brucella suis | A0A0H3G530_BRUSU | 39.3 | 3e-04 |
| Escherichia coli | CLPX_ECOLI | 860 | 0.0 |
| Escherichia coli | HSLU_ECOLI | 93.6 | 1e-21 |
| Escherichia coli | FTSH_ECOLI | 46.2 | 3e-06 |
| Escherichia coli | CLPA_ECOLI | 43.1 | 3e-05 |
| Paracoccus denitrificans | CLPX_PARDP | 580 | 0.0 |
| Paracoccus denitrificans | HSLU_PARDP | 103 | 5e-25 |
| Paracoccus denitrificans | A1B8N4_PARDP | 50.1 | 2e-07 |
| Paracoccus denitrificans | A1AZV8_PARDP | 44.3 | 1e-05 |
| Paracoccus denitrificans | RUVB_PARDP | 43.1 | 3e-05 |
| Paracoccus denitrificans | A1BBJ2_PARDP | 41.6 | 1e-04 |
| Paracoccus denitrificans | A1AY35_PARDP | 41.6 | 1e-04 |
| Polynucleobacter asymbioticus | CLPX_POLAQ | 613 | 0.0 |
| Polynucleobacter asymbioticus | A4SXL5_POLAQ | 43.5 | 1e-05 |
| Polynucleobacter asymbioticus | A4SZP7_POLAQ | 39.7 | 2e-04 |
| Polynucleobacter asymbioticus | A4T0L3_POLAQ | 38.1 | 3e-04 |
| Proteus mirabilis | CLPX_PROMH | 769 | 0.0 |
| Proteus mirabilis | HSLU_PROMH | 96.7 | 1e-22 |
| Proteus mirabilis | B4EV83_PROMH | 50.1 | 2e-07 |
| Proteus mirabilis | B4F2B3_PROMH | 46.6 | 2e-06 |
| Yersinia pestis | CLPX_YERPE | 805 | 0.0 |
| Yersinia pestis | HSLU_YERPE | 95.1 | 3e-22 |
| Yersinia pestis | A0A5P8YGZ0_YERPE | 51.2 | 7e-08 |
| Yersinia pestis | A0A5P8YB42_YERPE | 46.2 | 3e-06 |
| Yersinia pestis | A0A5P8YCE6_YERPE | 45.8 | 3e-06 |
Дерево гомологов
Я построила филогенетическое дерево этих белков при помощи сервиса NGPhylogeny, используя параметры из задания (MAFFT → FastME, "Gamma distributed rates across sites" — No, "Starting tree" — BIONJ, "No refinement", 100 бутстреп реплик). Скобочная форма дерева доступна для скачивания по ссылке.
Далее я визуализировала полученное дерево при помощи сервиса iTOL (рис. 1 и 2), укоренив его в мидпоинт.
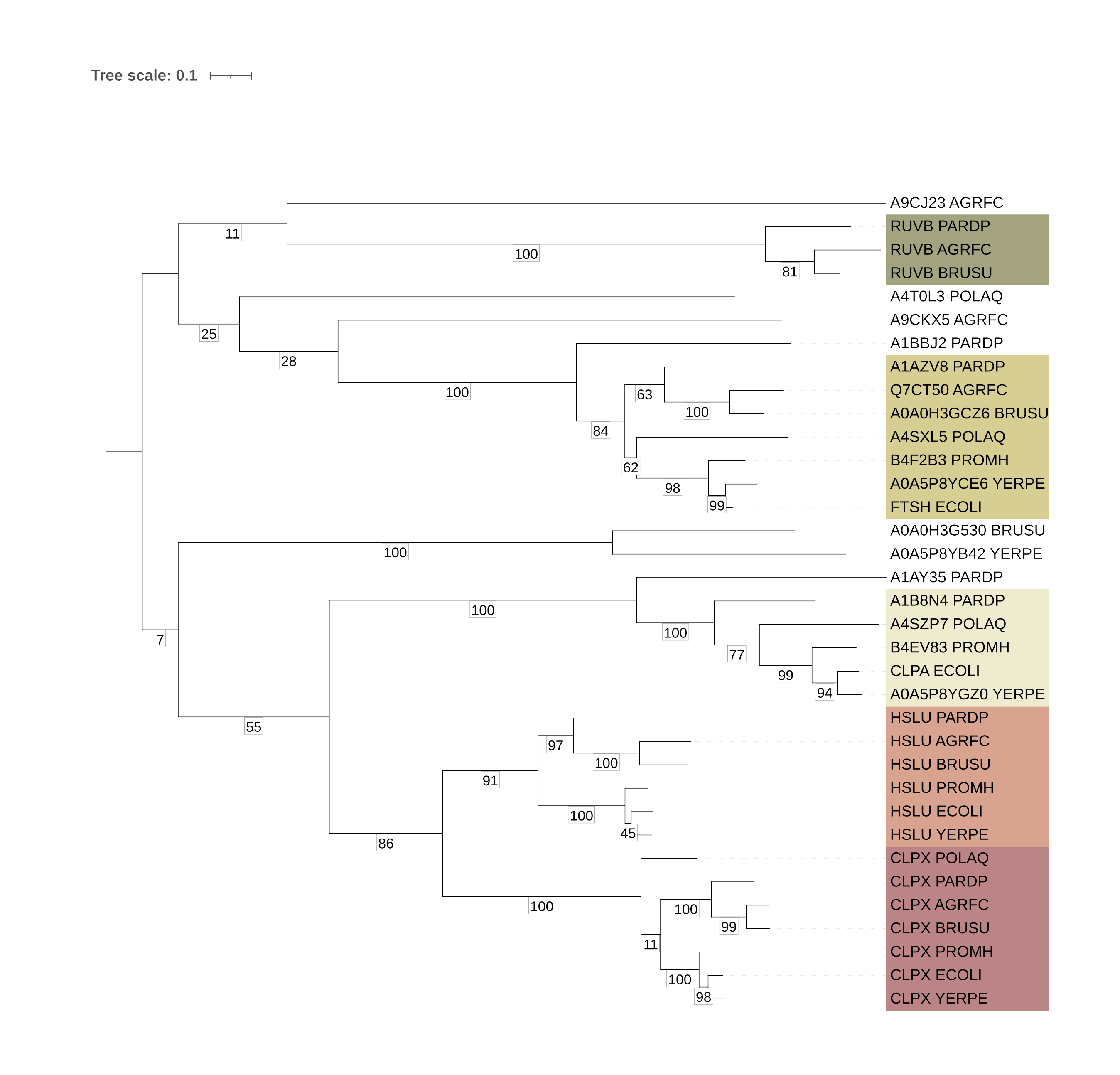
На этом дереве можно видеть множество ортологов и паралогов. Я считала группу белков ортологами, если они составляли одну кладу и в них не было белков из одной бактерии. Примеры пар ортологов — пары HSLU_BRUSU и HSLU_YERPE, CLPX_POLAQ и CLPX_AGRFC, RUVB_PARDP и RUVB_AGRFC. Поскольку мы считаем находки BLAST гомологами, любые два белка из одной бактерии будут паралогами. Примеры пар паралогов — CLPX_AGRFC и HSLU_AGRFC, CLPX_BRUSU и HSLU_BRUSU, CLPX_ECOLI и HSLU_ECOLI.
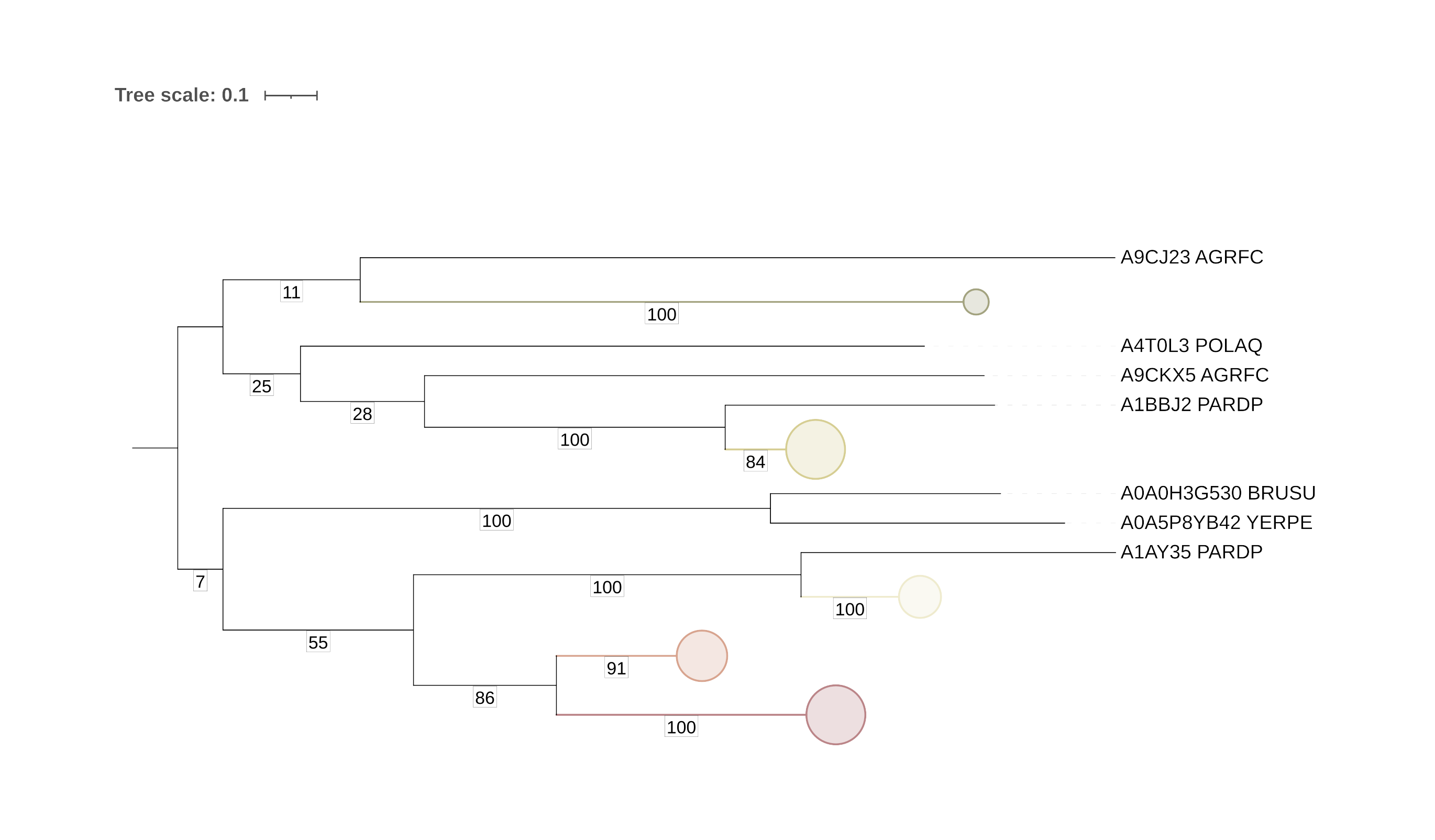
Схлопнутые группы сверху вниз:
1) Белок RuvB, субъединица комплкса, осуществляющего миграцию ветвей в структуре Холлидея;
2) Белок FtsH, АТФ-зависимая цинковая металлотрансфераза;
3) Белок ClpA, АТФ-связывающая субъединица АТФ-зависимой протеазы Clp;
4) Белок HslU, АТФазная субъедница АТФ-зависимой протеазы;
5) Сам белок ClpX, АТФ-связывающая субъединица АТФ-зависимой протеазы Clp.
Во всех группах ортологов, кроме группы белков CLPX, филогения белков соответствовала филогении бактерий. Группа CLPX оказалась как бы неправильно укореннена, белок CLPX_POLAQ оказался самым базальным, тогда как бактерия POLAQ на самом деле объединена в одну кладу с {PROMH, YERPE, ECOLI}. Но у этой неправильной ветви низкая поддержка bootstrap (11).