Практикум 6
Белок с бета-листами
Я выбрала белок с идентификатором UniProt VDAC1_HUMAN и идентификатором PDB 2JK4. UniProt по-английски описывает его как «Voltage-dependent anion-selective channel protein». Это потенциал-зависимый канал, находящийся во внешней мембраны митохондрий и в плазмалемме клеток человека. Он пропускает малые гидрофильные молекулы, а также катионы и анионы. Участвует в регуляции апоптоза и митофагии.
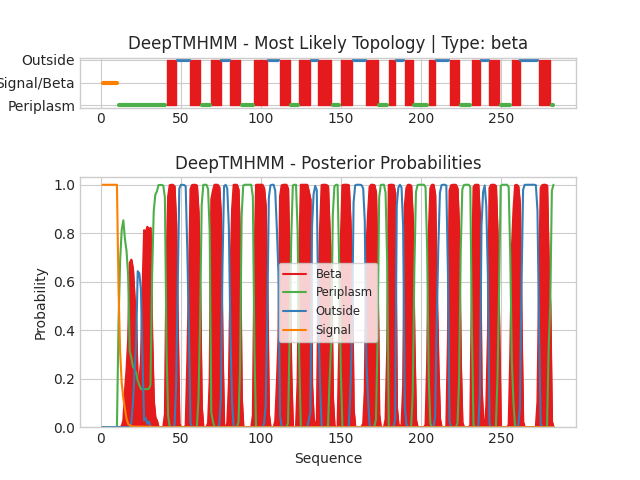
В базе данных OPM приведены координаты трансмембранных участков, дублирую их в табл. 1. Я запустила алгоритм DeepTMHMM с первичной последовательностью этого белка, файл с результататми можно скачать здесь, также результаты приведены на рис. 1. Этот алгоритм предсказал координаты 18 бета-листов, привожу их в табл. 1, «на глаз» установив соответствие.
Можно видеть, что предсказанное и реальное положение белка в мембране отличаются. Во-первых, DeepTMHMM вообще «не заметил» первый тяж бета-листа (29 — 36). На рис. 1 можно видеть, что в этом месте есть некоторые повышения вероятности бета-листа, но шумные. Возможно, это связано с тем, что алгорим не смог «понять», что этот тяж контактирует с самым концом этого белка, т. е. что этот белок заворачивается в кольцо. Во-вторых, видно, что координаты предсказанных бета-листов все время немного отличаются от коориднат реальных трансмембранных участков. Предсказанные участки все время начинаются на 1 — 2 аминокислоты раньше и заканчиваются на 3 — 4 аминокислоты раньше. Видимо, они заканчиваются раньше, потому что гидрофобные бета-листы лежат именно в гидрофобной жирнокислотной части мембраны, а в части мембраны с головками фосфолипидов уже более полярная среда, и нужды в бета-листах нет. А почему бета-листы и начинаются немного раньше, я понять не могу.
| Координата | OPM, трансмембранные участки | DeepTMHMM, бета-листы |
|---|---|---|
| 1 | 29 — 36 | — |
| 2 | 42 — 51 | 41 — 47 |
| 3 | 58 — 65 | 56 — 62 |
| 4 | 72 — 79 | 69 — 75 |
| 5 | 84 — 90 | 81 — 87 |
| 6 | 98 — 105 | 96 — 104 |
| 7 | 115 — 122 | 112 — 118 |
| 8 | 126 — 134 | 124 — 131 |
| 9 | 141 — 148 | 136 — 144 |
| 10 | 152 — 160 | 150 — 157 |
| 11 | 170 — 176 | 166 — 173 |
| 12 | 182 — 187 | 180 — 184 |
| 13 | 193 — 199 | 190 — 195 |
| 14 | 206 — 213 | 205 — 209 |
| 15 | 222 — 228 | 218 — 224 |
| 16 | 235 — 241 | 232 — 237 |
| 17 | 246 — 252 | 243 — 249 |
| 18 | 258 — 266 | 257 — 262 |
| 19 | 277 — 285 | 274 — 281 |
Белок с неизвестной структурой с альфа-спиралями
Для анализа мне достался белок идентификатором UniProt LUTP_BACSU (L-lactate permease) сенной палочки(Bacillus subtilis). Это пермеаза, транспортирующая L-лактат в клетку.
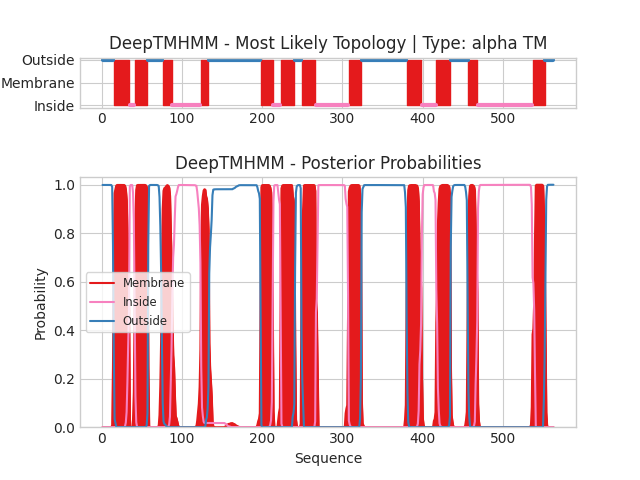
Я запустила DeepTMHMM с аминокислотной последовательностью этого белка. Результаты можно видеть на рис. 2, в табл. 2, а также скачать по ссылке
После я скачала стркутуру, предсказанную при помощи AlphaFold, и запустила с ней алгоритм PPM со следующими параметрами: Type of membrane — Gram-positive bacteria inner membrane (B. subtilis грамположительная), Allow curvature — no, Topology (N-ter) — out (поскольку DeepTMHMM предсказал положение N-конца снаружи). Результаты можно видеть в табл. 2, получившийся на выходе pdb файл можно скачать по ссылке
В табл. 2 можно видеть, что предсказания трансмембранных альфа-спиралей похожи, но не полностью совпадают. Например, различается количество предсказанных спиралей: PPM предсказал 15, DeepTMHMM — 12. Как и в прошлый раз, DeepTMHMM выдавал в среднем более короткие участки, но на этот раз они обычно и начинались позже, и заканчивались раньше.
| Координата | PPM, структура из AlphaFold, трансмембранные альфа-спирали | DeepTMHMM, трансмембранные альфа-спирали |
|---|---|---|
| 1 | 15 — 35 | 15 — 34 |
| 2 | 39 — 58 | 41 — 57 |
| 3 | 62 — 91 | 76 — 87 |
| 4 | — | 124 — 133 |
| 5 | 161 — 170 | — |
| 6 | 175 — 184 | — |
| 7 | 188 — 210 | 198 — 213 |
| 8 | 223 — 246 | 223 — 240 |
| 9 | 247 — 262 | 249 — 266 |
| 10 | 308 — 328 | 308 — 323 |
| 11 | 380 — 398 | 380 — 398 |
| 12 | 416 — 438 | 417 — 434 |
| 13 | 453 — 470 | 457 — 468 |
| 14 | 471 — 487 | — |
| 15 | 494 — 515 | — |
| 16 | 536 — 555 | 537 — 552 |