Практикум 8
Описание сигнала ориджина репликации oriC
OriC — это небольшой (до пары тысяч п.н.) участок в геномах бактерий, с которого начинается репликация их хромосомы. Он состоит из двух основных участков: duplex-unwinding element (DUE) и DnaA-oligomerization region (DOR), содержащий т.н. DnaA boxes[1, 2]. DnaA boxes обычно состоят из непалиндромных 9 п.н.[3] DUE — AT-богатый регион; в E. coli это 13-мер последовательности GATCTnTTnTTTT[4].
Рядом с oriC зачастую находятся ген dnaA и некоторые другие маркерные гены[3].
Ориждин репликации может состоять как из одной части, так и из двух. Если он состоит из двух частей, они обычно разделены геном dnaA[2].
В oriC есть DnaA boxes с высоким и низким сродством к DnaA. Боксы с высоким сродством связаны с DnaA на протяжении всего клеточного цикла, тогда как боксы с низким сродством связываются с DnaA во время инициации репликации, обеспечивая его олигомеризацию. Олигомер DnaA расплетает DUE и привлекает другие белки, необходимые для инициации репликации, в том числе хеликазы DnaB и DnaC[4].
Существует онлайн-сервис для поиска oriC в бактериальных геномах, Ori-finder 2022. Для предсказания ориджина он использует GC-skew, AT-skew, RY-skew (пурино-пиримидиновый) и MK-skew (амино-кето), а также расположение маркерных генов и паттерны DnaA boxes.
Поиск oriC в геноме Serpentinimonas maccroryi
Для поиска ориджина репликации я взяла геном бактерии, который я анализировала в первом семестре первого курса. Это была бактерия Serpentinimonas maccroryi.
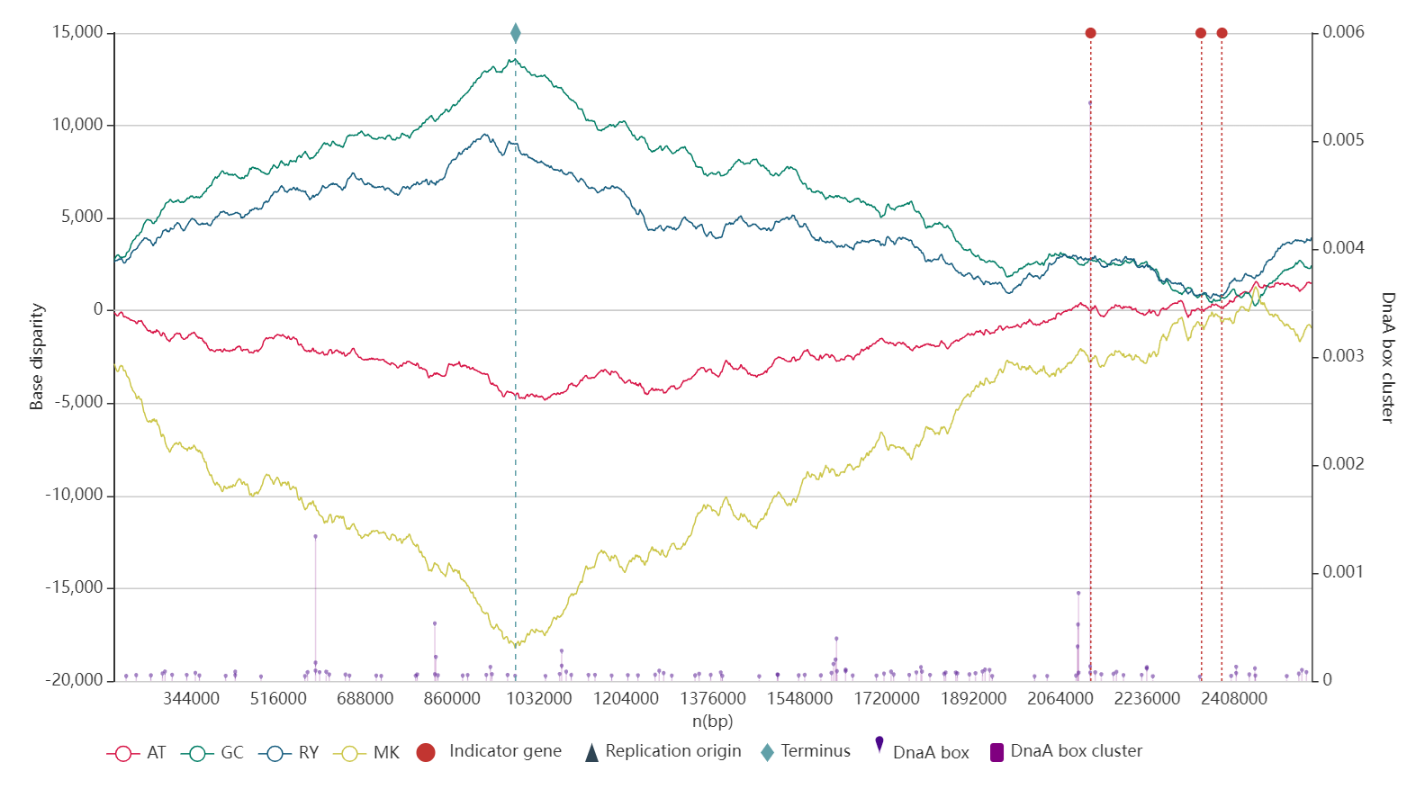
Я использовала сервис Ori-finder 2022 с параметрами Topology — Circular, Lineage — bacteria, Assembly level — Complete и остальными параметрами по умлочанию. Визуализацию результатов можно видеть на рис. 1.
Ori-finder нашел два участка oriC: 1 375 — 1 524 п. н. и 2 606 378 — 2 606 868 п. н. Эти участки разделены геном dnaA, где-то с его середины и начинается отсчет координат в хромосоме, поэтому эти участки на самом деле находятся рядом. Также сразу за первым участком находится ген DnaN, но вот другие характерные гены находятся далеко.
Последовательность Шайна — Дальгарно в геноме S. maccroryi
Код на python, написанный для выполнения этого задания, можно найти в колабе по ссылке.
Я попыталась найти сигнал последовательности Шайна-Далгарно в геноме той же бактерии, S. maccroryi. Я брала последовательности по 23 нуклеотида, 3 последних из которых — старт-кодон. Я разделила все эти последовательности на 2 равные группы: тренировочную и тестовую. Для контроля я взяла столько же (как в тестовой группе) последовательностей перед кодонами ATG, не являющимися инициаторными. Я построила pwm на данных тестовой группы последовательностей, затем посчитала веса для всех последовательностей всех групп. Результаты можно видеть на рис. 2.
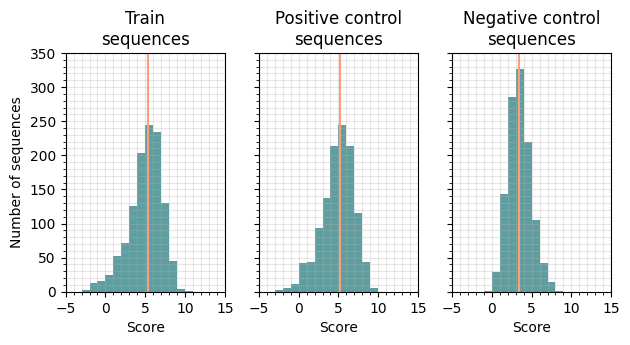
Медианный вес последовательностей в тренировочном сете — 5,28, в тестовом сете — 5,15, в отрицательном контроле — 3,39. Веса в отрицательном контроле положительны, видимо, поскольку я брала полследовательности вместе с кодоном ATG в конце: он один давал вес в 4,4 (а это больше медианы для группы отрицательного контроля!).
Медианы тестовой группы и группы негативного контроля значимо отличаются (односторонний тест Манна — Уитни, p = 5.6 * 10-87). Также значимо отличаются мединаы тестовой и тренировочной группы (односторонний тест Манна — Уитни, p = 0.045). Видимо, это значит, что для тренировочного сета немного не хватило размера выборки.
На гистограммах видно, к сожалению, что любая граница, которую мы проведем, будет условной. Я взяла границу в 4,7, чтобы вероятность ошибки первого рода была не больше 20%. Количество последовательностей в каждой группе выше и ниже границы можно видеть в табл. 1.
| Тренировочный сет | Тестовый сет | Сет отрицательного контроля | |
|---|---|---|---|
| Выше границы | 726 (62%) | 690 (59%) | 232 (20%) |
| Ниже границы | 440 (38%) | 477 (41%) | 934 (80%) |
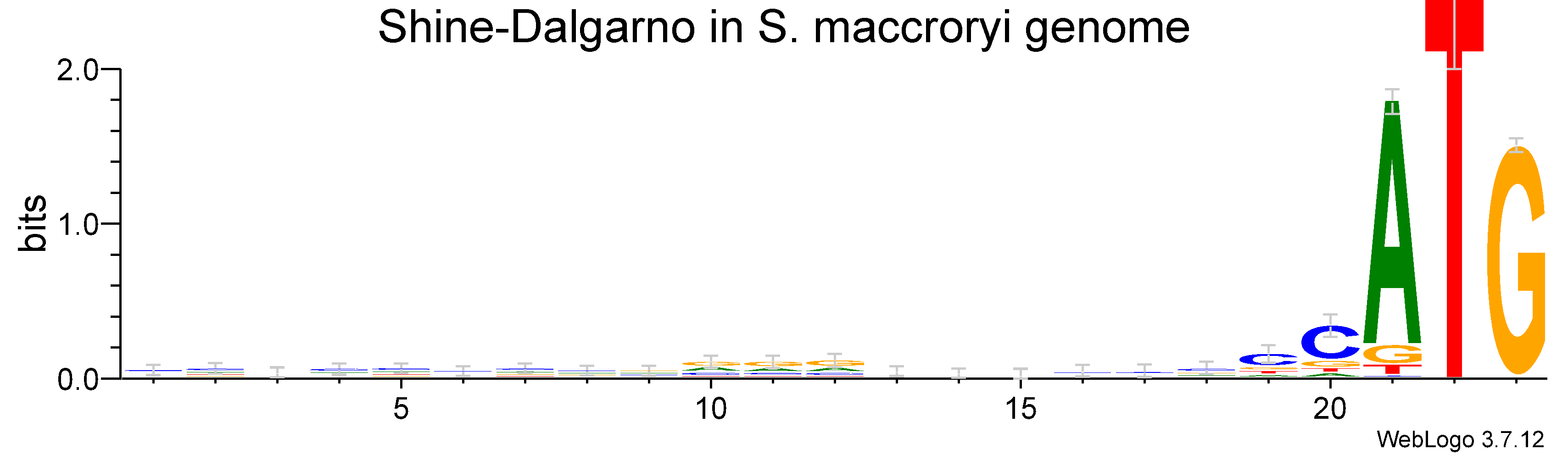
Посмотрим теперь на визуализацию энтропии букв в этой последовательности. Я построила визуализацию на рис. 3 при помощи сервиса WebLOGO 3, подав как входные данные тренировочный сет последовательностей и изменив в параметрах GC-состав на 66% — GC-состав S. maccroryi.
Можно видеть, что наиболее консервативная часть этих последовательностей — старт-кодон. Помимо старт-кодона, консервативен регион совсем перед ним и приблизительно на -10 нуклеотидов.
Список литературы
- Katayama T. Initiation of DNA Replication at the Chromosomal Origin of E. coli, oriC. Adv Exp Med Biol. 2017;1042:79-98. doi: 10.1007/978-981-10-6955-0_4. PMID: 29357054.
- Wolański M, Donczew R, Zawilak-Pawlik A, Zakrzewska-Czerwińska J. oriC-encoded instructions for the initiation of bacterial chromosome replication. Front Microbiol. 2015 Jan 6;5:735. doi: 10.3389/fmicb.2014.00735. PMID: 25610430; PMCID: PMC4285127.
- Dong MJ, Luo H, Gao F. Ori-Finder 2022: A Comprehensive Web Server for Prediction and Analysis of Bacterial Replication Origins. Genomics Proteomics Bioinformatics. 2022 Dec;20(6):1207-1213. doi: 10.1016/j.gpb.2022.10.002. Epub 2022 Oct 17. PMID: 36257484; PMCID: PMC10225481.
- Leonard AC, Grimwade JE. The orisome: structure and function. Front Microbiol. 2015 Jun 2;6:545. doi: 10.3389/fmicb.2015.00545. PMID: 26082765; PMCID: PMC4451416.