
С помощью алгоритма pblast (то есть blast для белковых последовательностей) был проведен поиск возможных гомологов белка YPJD_BACSU по базе RefSeq. RefSeq - это крупная база данных, где представлены полные геномы. Для получения нужных результатов были изменены некоторые параметры, представленные в таблице 1. Гомологами считались белки с E-value меньше, чем 10-5. Для поиска его значение было задано равным 1.
Таблица 1. Таблица с заданными для поиска параметрами BLAST.
| Поиск | Алгоритм BLAST | Название базы данных | Ограничения по таксонам | Порог e-value | Максимальное количество хитов (количество результатов) |
| По прокариотам | Blastp (protein-protein BLAST) | RefSeq | Без филума Firmicutes | 1 | 1000 (613) |
| По эукариотам | Blastp (protein-protein BLAST) | RefSeq | Только Eukaryota | 1 | 100 (39) |
Гомологи белка были найдены у бактерий, архей и даже вирусов (5 белков, принадлежащих двум представителям). Мною были выбраны по 1-2 гомологам разных представителей каждого филума (филумы - самые крупные единицы дробления у прокариот, сравнимые с делением на царства у эукариот), кроме филума Firmicutes (к которому принадлежит рассматриваемый белок, но он также указан в таблице). Гомологи белка YPJD_BACSU встречаются также и у эукариот. Не нашлось ни одного белка, для которого было бы указано неэукариотическое происхождение (то есть белок кодировался бы в геноме митохондрии или хлоропласта), так что можно предположить, что все гомологи относятся к ядерному геному. В таблице 2 приведены большинство таксонов, в которых встречаются гомологи YPJD_BACSU, а также названия представителей, чьи последовательности были сохранены мной для создания множественного выравнивания.
Таблица 2. Таблица встречаемости белков в различных таксонах. Серым выделен организм, которому принадлежит белок YPJD_BACSU.
| Домен | Филум/Царство | Название организма | Количество белков |
| Archaea | Crenarchaeotes | Pyrococcus horikoshii | 45 |
| Thermococcus kodakarensis | |||
| Euryarchaeotes | Desulfurococcus fermentans DSM 16532 | 2 | |
| Desulfurococcus kamchatkensis 1221n | |||
| Bacteria | Actinobacteria | Candidatus Microthrix parvicella | 3 |
| Cellulomonas flavigena DSM 20109 | |||
| CFB group bacteria | Cytophaga hutchinsonii | 215 | |
| Marinilabilia sp. AK2 | |||
| Deinococcales | Deinococcus geothermalis | 7 | |
| Firmicutes | Bacillus subtilis strain 168 | ||
| Planctomycetes | Blastopirellula marina | 10 | |
| Rhodopirellula baltica | |||
| Spirochetes | Leptospira interrogans | 31 | |
| Leptospira wolbachii | |||
| Thermales | Marinithermus hydrothermalis DSM 14884 | 9 | |
| Thermus aquaticus | |||
| Eukaryotes | Animals | Ailuropoda melanoleuca | 17 |
| Bos taurus | |||
| Canis lupus familiaris | |||
| Cavia porcellus | |||
| Dasypus novemcinctus | |||
| Felis catus | |||
| Metaseiulus occidentalis | |||
| Ochotona princeps | |||
| Odobenus rosmarus divergens | |||
| Oryctolagus cuniculus | |||
| Otolemur garnettii | |||
| Ovis aries | |||
| Sarcophilus harrisii | |||
| Sorex araneus | |||
| Sus scrofa | |||
| Green plants | Cucumis sativus | 21 | |
| Fragaria vesca subsp. Vesca | |||
| Glycine max | |||
| Physcomitrella patens subsp. patens | |||
| Populus trichocarpa | |||
| Ricinus communis | |||
| Solanum lycopersicum | |||
| Vitis vinifera | |||
| Zea mays | |||
| Viruses | Mycobacterium phage SWU1 | 5 | |
| Streptococcus phage 2972 |
Ниже приведены изображения "деревьев" таксономических групп организмов, к которым принадлежат найденные гомологи.
 |
 |
| Рисунок 1. Прокариотические гомологи. | Рисунок 2. Эукариотические гомологи. |
Выравнивание было построено с помощью программы Muscle на сайте Европейского Биоинформатического Института. Результат выравнивания и эукариотических, и прокариотических гомологов белка YPJD_BACSU (и его включительно) можно увидеть на рисунке 3.
Для окраски использую созданную мной схему, где цвета соответствуют следующим аминокислотам: светло-зеленый – алифатические; зеленый – иминокислота пролин; желтый – серосодержащие; синий – положительно заряженные; красный – отрицательно заряженные; розовый - нейтральные.
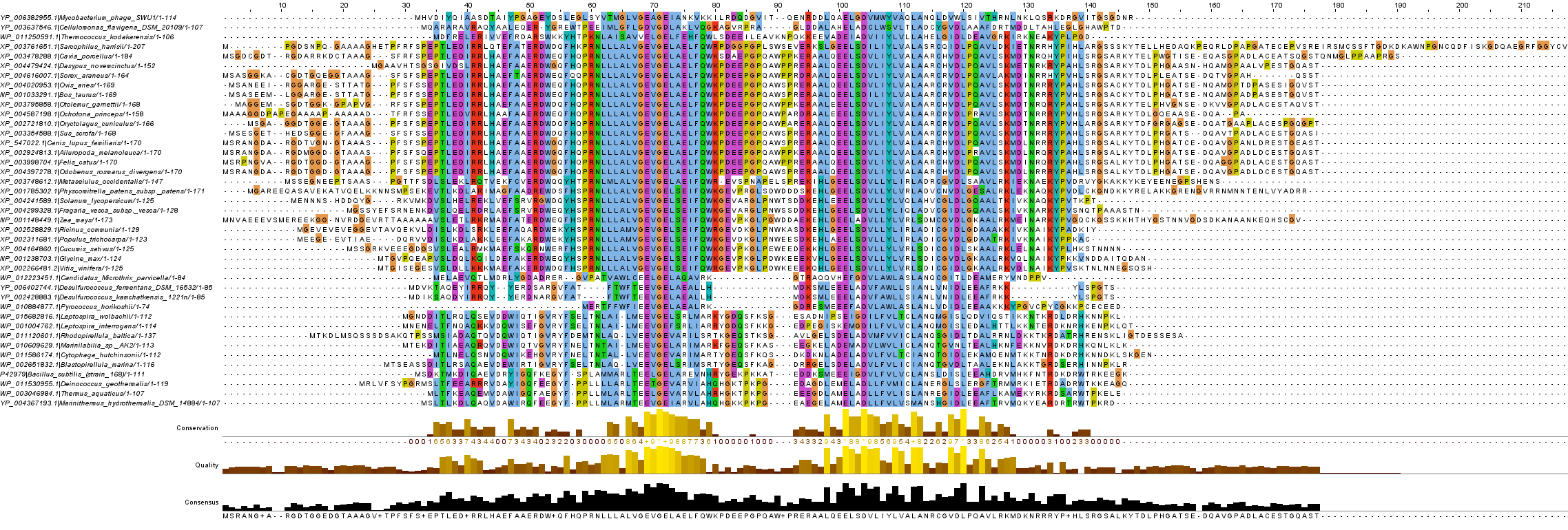 Рисунок 3. Множественное выравнивание белка YPJD_BACSU и его гомологов с помощью программы Muscle.
Ссылка на выравнивание в формате jar
Рисунок 3. Множественное выравнивание белка YPJD_BACSU и его гомологов с помощью программы Muscle.
Ссылка на выравнивание в формате jar
Выравнивание можно считать хорошим, так как большинство участков консеративности приходится на какие-либо элементы вторичной структуры.
Самое плохое выравнивание (или почти отсутствующее) - выравнивание на концах и между блоками. Разброс на этих участках закономерен и объясняется тем, что на этих участках обычно располагаются петли (они не образуют вторичной структуры и могут сильно видоизменяться в ходе эволюции).
На участки вторичной структуры приходится меньше колонок-гэпов, чем на остальное выравнивание, однако существуют белки, в которых на участках, соответствующих вторичной структуре, возникают вставки. В результате прямо на середине спирали могут образовываться колонки гэпов.
Выбранные мной блоки в значительной мере совпадают с элементами вторичной структуры, выбор был основан на большой толщине и в какой-то степени консервативности выравнивая на каждом участке.
На рисунках 4 и 5 представлены иллюстрации консервативности остатков, связывающих лиганд в белке YPJD_BACSU. В белке 4 цепи и каждая связывает по одному лиганду - иону натрия. Цепи идентичны, на примере цепи А иожно увидеть, что лиганд связывают 3 остатка глутаминовой кислоты (Glu37, Glu40 и Glu66). Консервативность этих участков очень и очень высока: 60-ый не заменяется, 37-ый и 40-ой остатки иногда заменяются на аспарагиновую кислоту. Такая замена является довольно логичной.
Нельзя определенно сказать, что все гомологи способны связывать этот лиганд, однако эта вероятность очень высока, так как эти остатки почти не заменяются и лежат напротив участков вторичных структур (спиралей).
Даже по названиям некоторых гомологов: MazG, пирофосфотазы, неохарактеризованные белки, -
можно говорить о схожести их функций. Тот факт, что большинство этих белков имеют высококонсервативные участки
в местах связывания лиганда (то есть, теоретически, они тоже могут связывать ионы натрия),
также говорит о том, что они могут выполнять похожие функции с белком YPJD_BACSU.

Рисунок 4. Расположение аминокислотных остатков, связывающих лиганд, по отношению ко всему белку.
Лиганд покрашен фиолетовым цветом, аминокислотные остатки - по разработанной мной схеме.
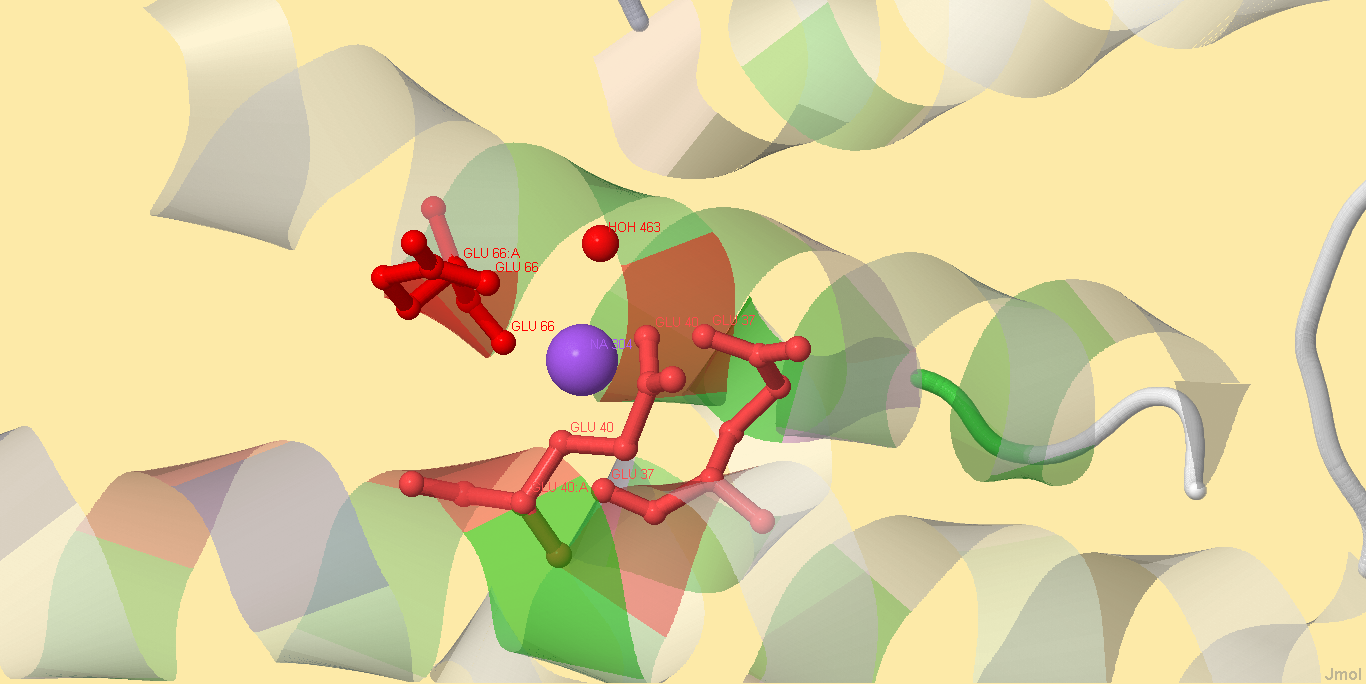
Рисунок 5. Отдельно цепь А. Расцветка такая же, как и на рисунках 3 и 4. Подписаны остатки глутаминовой кислоты, связывающие лиганд.
Источники: