
| Доменная структура белка YPJD_BACSU по данным Pfam | ||||
Cхема из Pfam:  |
||||
| MazG nucleotide pyrophosphohydrolase domain | ||||
| Pfam AC | Pfam ID | Полное название семейства доменов | Положение в последовательности белка YPJD_BACSU | Клан |
| P42979 | MazG | Нуклеотид пирофосфогидролазы (или нуклеозид-трифосфат пирофосфогидролазы) | 25 - 104 | MazG (CL0231), в который также входят DUF550, dUTPase_2, MazG-like, PRA-PH |
Домен MazG входит в 20 различных архитектур, причем существует 3126 последовательностей с такой же архитектурой, как у белка YPJD_BACSU.
Известны последовательности 7328 белков, содержащих этот домен.
Для 8 белков, содержащих домен, определена пространственная структура (всего 41 структура) Выравнивание "seed" фрагментов белков, соответствующих домену.
Существует 229 последовательность со следующей архитектурой: TP_methylase, MazG

Рассматриваются последовательности, в которых встречается один из доменов данной архитектуры.
Представленность доменов TP_methylase (PF00590) и MazG (PF03819) в организмах разных таксонов
| Таксон | Количество белков с доменом PF00590. | Количество белков с доменом PF03819. | |
| Эукариоты | Зеленые растения | 60 | 105 |
| Грибы | - | 266 | |
| Животные | 54 | 112 | |
| Остальные эукариоты | 6 | 95 | |
| Археи | 725 | 115 | |
| Бактерии | 15713 | 5051 | |
| Вирусы | - | 64 | |
Оба рассматриваемых домена являются преимущественно бактериальными, однако эти домены существуют и у других таксонов, причем MazG более распространен среди них.
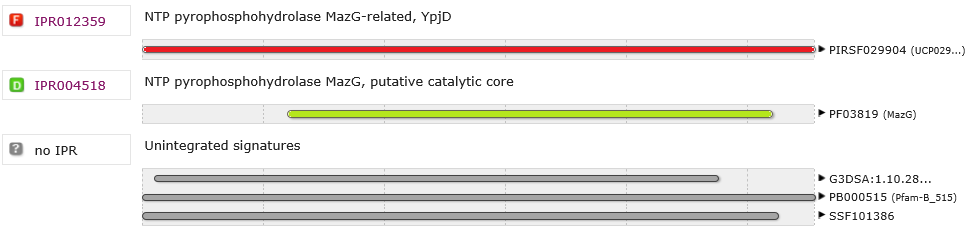
Самый короткий мотив - домен PF03819 (выделен зеленым цветом). Он описан в банке Pfam.
Cамый длинный мотив - PIRSF029904 (выделен красным). Он описан в банке PIRS.
В InterPro интегрированы следующие структурные подписи: NTP pyrophosphohydrolase MazG-related, YpjD (включает в себя весь белок) и NTP pyrophosphohydrolase MazG, putative catalytic core (домен MazG).
Границы структурных доменов не отличаются от границ доменов Pfam.
Источники: