
 |
СЕМЕСТРЫ | ПРОЕКТЫ | ОБО МНЕ | ССЫЛКИ |
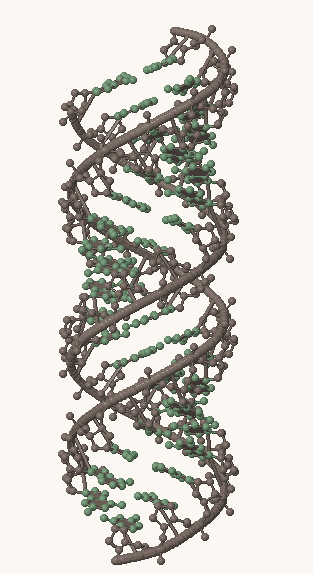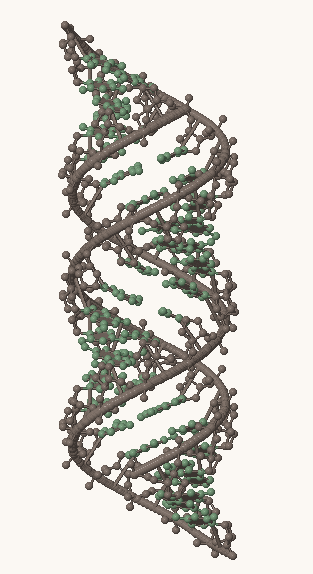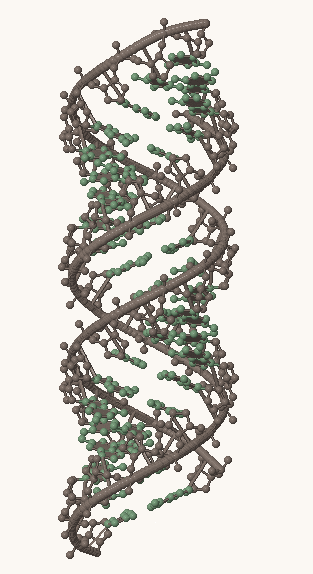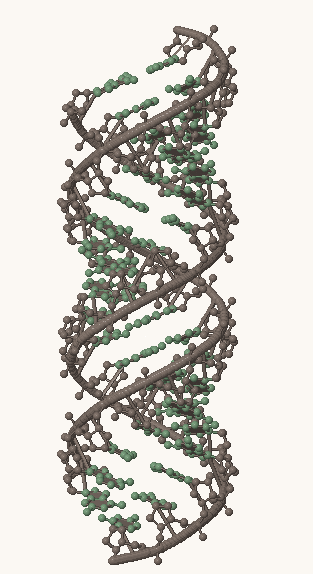
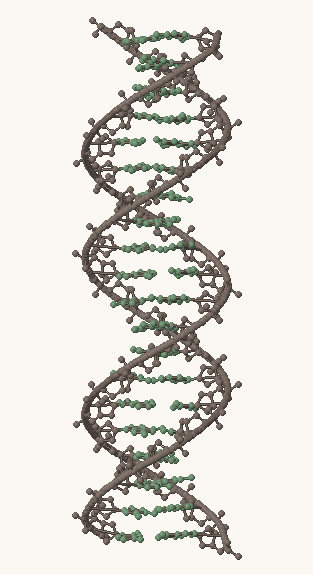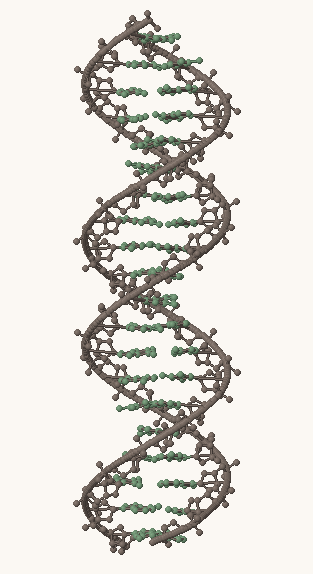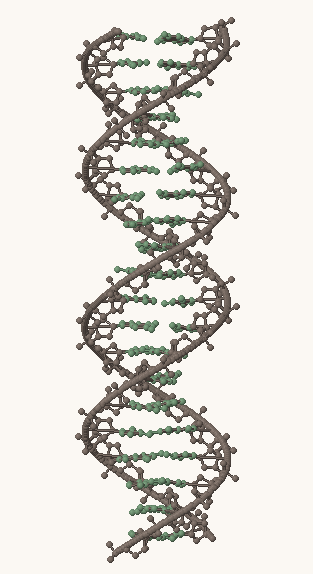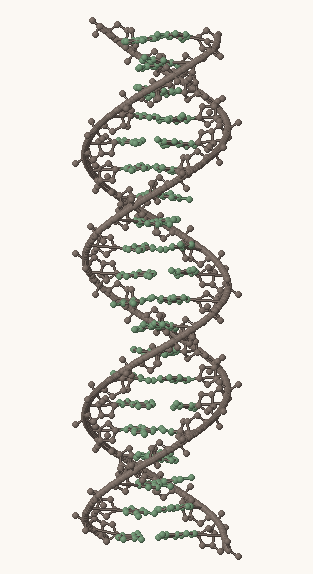
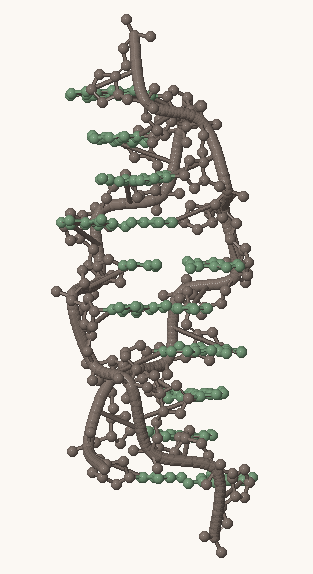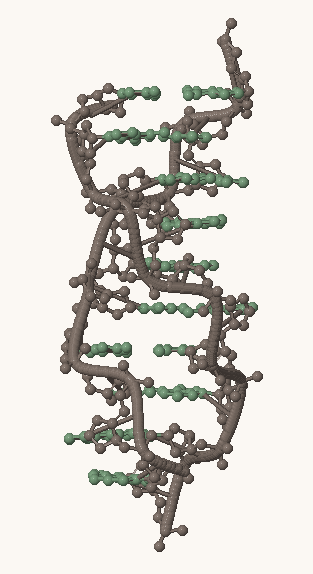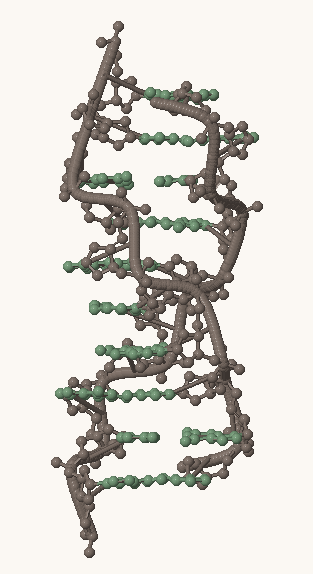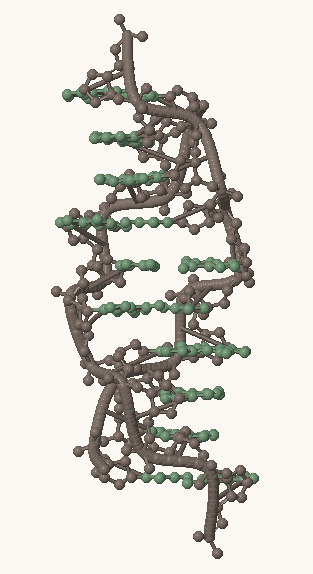
Рис. 1, 2, 3. Структуры A-, B- и Z-формы ДНК, соответственно. Серым выделен сахарофосфатный остов, остальные атомы покрашены в зеленый цвет.
 |
 |
 |
 |
| Рис. 4. | Рис. 5. | Рис. 6. | Рис. 7. |
В данных мне белках (с идентификатором 1H4S - Prolyl-tRNA синтетаза и 1MHD, кристаллическая структура связи домена SMAD MH1 с ДНК) не было обнаружено разрывов цепей нуклеиновых кислот, что и показывает приведенная ниже проволочная структура.
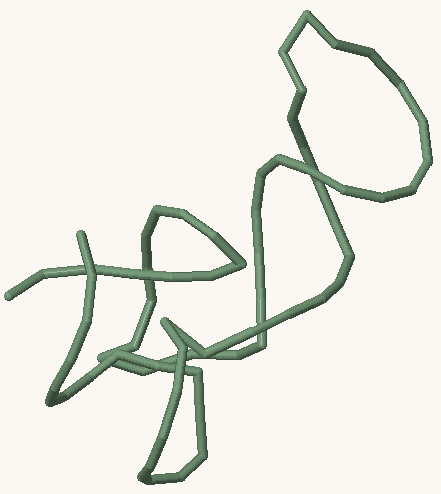
Рис. 8. РНК

Рис. 9. ДНК
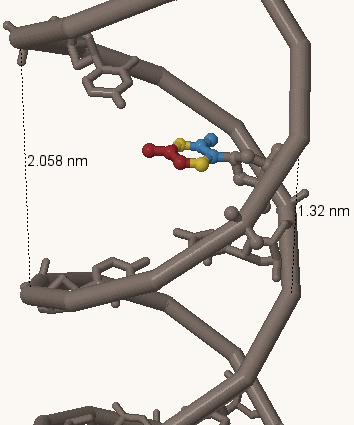
Рис. 10. Цитозин №12 на участке цепи B-формы ДНК с отмеченными большой и малой бороздками.
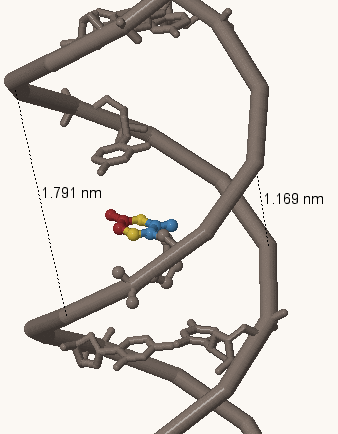
Рис. 11. Цитозин №12 на участке цепи A-формы ДНК с отмеченными большой и малой бороздками.
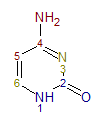
Рис. 12. Рисунок в Chemsketch для обеих структур.
| A-форма | B-форма | *Z-форма | |
| Тип спирали | правая | правая | левая |
| Шаг спирали (A) | 28,03 | 33,75 | 43,5 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 16,81 ([T]31:B.P - [G]13:A.P) | 17,91 ([T]11:A.P - [A]26:B.P) | 18,30 ([C]14:B.P - [C]4:A.P) |
| Ширина малой бороздки | 7,98 ([A]10:A.P - [G]25:B.P) | 11.69 ([T]31:B.P - [A]14:A.P) | 9.87 ([G]7:A.P - [C]18:B.P) |
В Jmol находим значения торсионных углов для разных форм.
На рисунках показаны проведенные измерения.
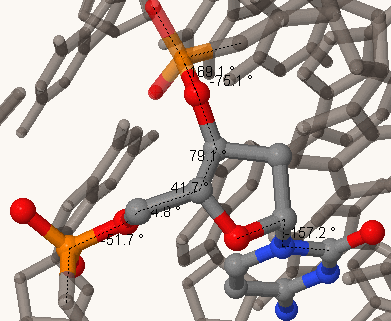
Рисунок 1. А-форма

Рисунок 1. В-форма
Форма |
a |
b |
c |
d |
e |
g |
h |
А(Вручную) |
64,1 |
174,8 |
41,7 |
79,1 |
169,1 |
-75,1 |
-157,2 |
А |
62 |
173 |
52 |
88 или 3 |
178 |
-50 |
-160 |
В(Вручную) |
85,9 |
136,3 |
31,2 |
143,3 |
105,8 |
-44,7 |
-98 |
В |
63 |
171 |
54 |
123 или 131 |
155 |
-90 |
-117 |
В файлах с названиями gatc-*_old.out находим торсионные углы и сравниваем найденные значения с полученными вручную в Jmol.
Форма/td> | a |
b |
c |
d |
e |
g |
h |
А-форма (Вручную) |
64,1 |
174,8 |
41,7 |
79,1 |
169,1 |
-75,1 |
-157,2 |
A (analyze) |
51.7 |
174.8 |
41.7 |
79.1 |
-147.8 |
-75.1 |
-157.2 |
В (Вручную) |
85,9 |
136,3 |
31,2 |
143,3 |
105,8 |
-44,7 |
-98 |
В (analyze) |
-29.9 |
136.3 |
31.2 |
143.3 |
-140.8 |
-160.5 |
-98.0 |
Z (analyze) |
-139.5 (51.9) |
-136.8 (179.0) |
50.8 (-173.8) |
137.6 (94.9) |
-96.5 (-103.6) |
82.0 (-64.8) |
-154.3 (58.7) |
Пакет 3DNA пока работает только со старым форматом PDB, поэтому сначала переводим файлы в старый формат с помощью программы remediator, установленной на kodomo.
Применив команду find_pair -t 1H4S_old.pdb stdout | analyze находим значения торсионных углов для заданной тРНК. С помощью excel находим средние значения, представленные в таблице ниже. Данная тРНК больше всего похожа на А-форму, а сильнее других значения отклоняются у гуанинов (особенно 5, 9, 17 и некоторых других).
1H4S (средние) |
-38,07 |
61,35 |
51,07 |
86,26 |
-150,73 |
-71,21 |
-130,40 |
G5 |
146,70 |
51,60 |
84,10 |
-134,30 |
-73,10 |
177,10 |
|
G9 |
140,90 |
-141,80 |
176,10 |
81,00 |
-129,10 |
-79,40 |
174,00 |
G17 |
160,40 |
-172,10 |
179,80 |
83,8 |
-142,20 |
-79,00 |
179,10 |
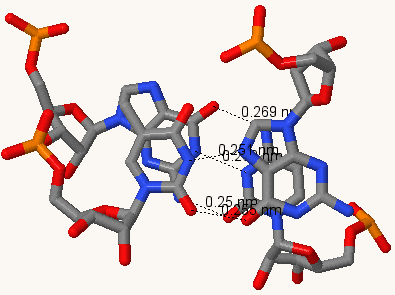
В файле 1H4S_old.out была найдена информация о вторичной структуре заданной тРНК, в таблице цветом выделены нуклеотиды, образующие стебли. Остальные пары также участвуют в стабилизации структуры РНК.
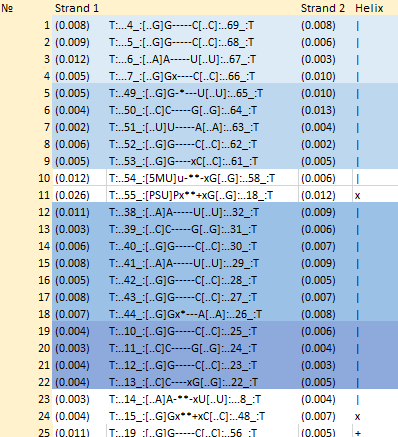
В том же файле найдем неканонические пары оснований, они выделены цветом в таблице, приведенной ниже.
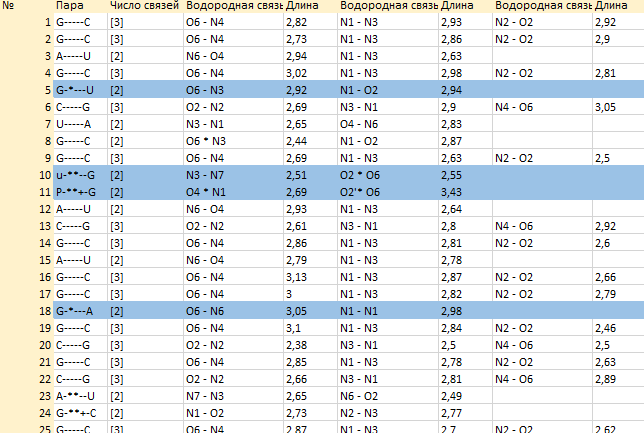
Взаимодействия расположенных друг над другом пар нуклеотидов называются стекинг-взаимодействиями и помогают в стабилизации третичной структуры РНК.
Сила взаимодействий зависит от площади перекрывания этих пар. В таблице представлены площади взаимных перекрываний.
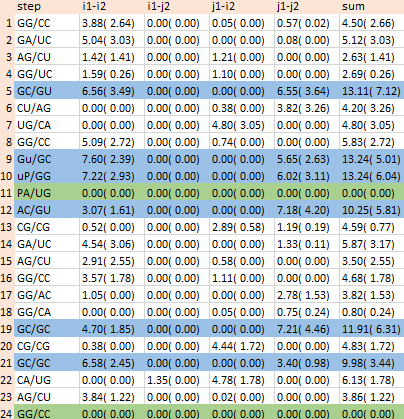
В таблице голубым цветом выделены пары с наибольшими перекрываниями, зеленым - с наименьшими (нулевыми).
Рассмотрим большую площадь перекрывания на примере 9 участка, используя команды:
ex_str -9 stacking.pdb step9.pdb
stack2img -cdolt step9.pdb step9.ps
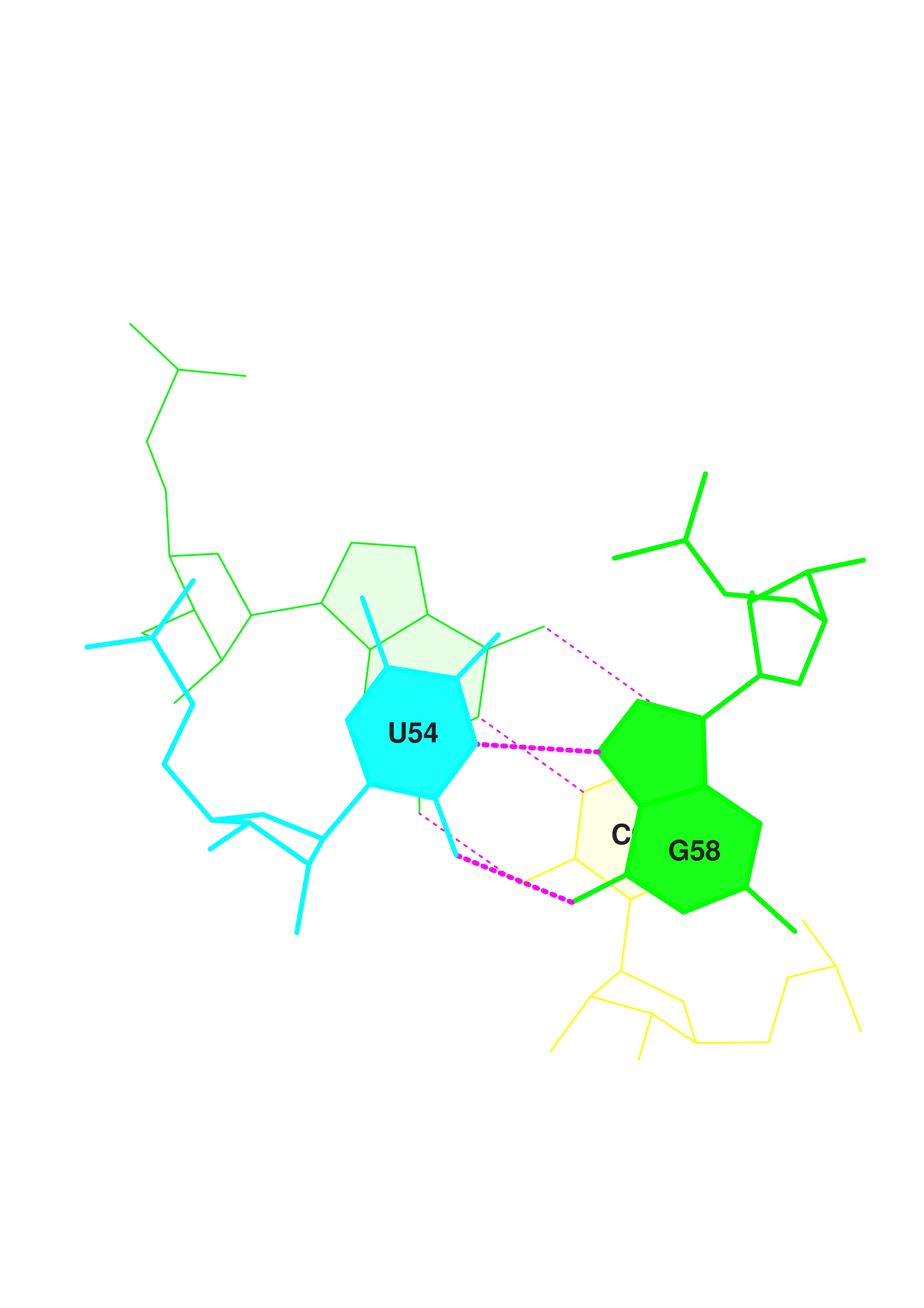
Перекрытия можно наблюдать и в Jmol.
Наименьшую площадь рассмотрим на примере 24 участка, видно, что участки совсем не перекрываются.