
 |
СЕМЕСТРЫ | ПРОЕКТЫ | ОБО МНЕ | ССЫЛКИ |
Для файла полученного командой "entret embl:D89965" нужно получить набор трансляций всех открытых рамок программой getorf. Рамки должны быть определены при использовании стандартного кода, начинаться со старт-кодона (или начала последовательности) и заканчиваться стоп-кодоном (или концом последовательности), их длина должна быть не менее 30 аминокислотных остатков.
Скрипт для получения: getorf d89965.entret -minsize 30 -find 1
Последовательность полностью соответствует пятой рамке:
>D89965_5 [163 - 432] Rattus norvegicus mRNA for RSS, complete cds. MALMHFQFTFKQFEQRKSIRSTARKARDDFVVVQTADLFHVAFHYGIAQRGLTITSDDHMAVTAYAYYSCHELTPWLRIQSTNPVQKYGA
Данная запись embl ссылается на последовательность, принадлежащую Escherichia coli (strain K12), а не Rattus norvegicus. Эта последовательность из Swiss-Prot полностью выравнялась с 9 рамкой считывания.
Query 7543 MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR 7602
MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR
Sbjct 1 MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR 60
Query 7603 MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGS 7640
MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGS
Sbjct 61 MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGS 98
Скрипт для получения выравнивания: blastp -query hslv_ecoli.entret -subject d89965.orf -out blastp.out
Такой странный результат (совпадение последовательностей крысы и кишечной палочки) можно объяснить тем, что была взята ткань желудка крысы, и при сиквенировании произошла ошибка - данные кишечной палочки случайно попали в крысиный геном. База embl может содержать ошибки в аннотациях, в отличие от swissprot, где проверка данных намного строже, нужно быть внимательными при использовании embl.
С помощью программ пакета EMBOSS: нужно скачать все доступные в Swissprot последовательности алкогольдегидрогеназ:
Скрипт: seqret sw:adh*_* -out adh.fasta
Получили файл с универсальными адресами (USA) этих последовательностей, результат - пример файла-списка.
infoseq adh2_soltu.fasta -only -usa -out adh2.out
Получили из этого файла-списка другой, меньший, с адресами только тех последовательностей, которые взяты из
infoseq adh2_soltu.fasta -only -usa -out adh2.out
На основе нового файла-списка получили fasta-файл с последовательностями дегидрогеназ ваших организмов
grep adh2.out -f organisms.txt > organisms_list.txt
seqret @organisms_list.txt
Портал EnsEMBL предназначен для визуализации известной информации о геномах человека и животных.
Получили последовательность всего гена hTPK1, чьим продуктом является тиамин пирофосфокиназа.
Команда: seqret ab028138.entret
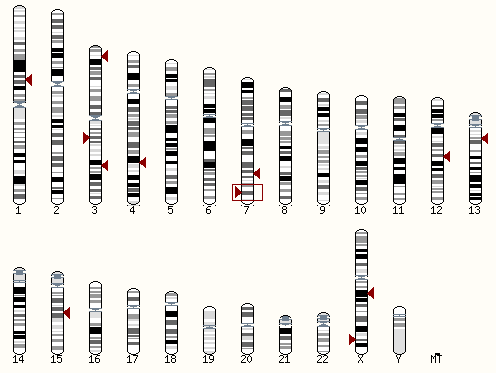
Нашли 22 выравнивания, на первой странице поиска можно увидеть: расположение гомологичных участков на хромосомах
выравнивание с запросом
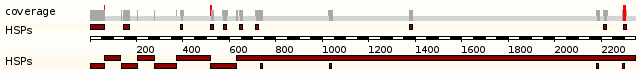
и краткое содержание выравнивания
Можно пройти по ссылке [A] - allignment, открылось выравнивание, ниже приведен отрывок:
Query: 2345 gataagtatccatttattaacactggtaacattttcagggcacacagaacatgcattctt 2286
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct: 144149043 gataagtatccatttattaacactggtaacattttcagggcacacagaacatgcattctt 144149102
Query: 2285 tggtagcaaaattttaaaatcacagatatatttcttttatatattatgaatatatttcta 2226
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct: 144149103 tggtagcaaaattttaaaatcacagatatatttcttttatatattatgaatatatttcta 144149162
А также можно посмотреть интрон-экзонную структуру, перейдя по [G].
Прошли по гиперссылке "Contig view". Открылась страница "Region in detail", посвящённая участку человеческого генома. Первый участок находится на 7 хромосоме с координатами: 144,147,043-144,152,756.
Показано более точное расположение гена на хромосоме.

Есть детальное рассмотрение участка и выравненные отноосительно запроса гены.
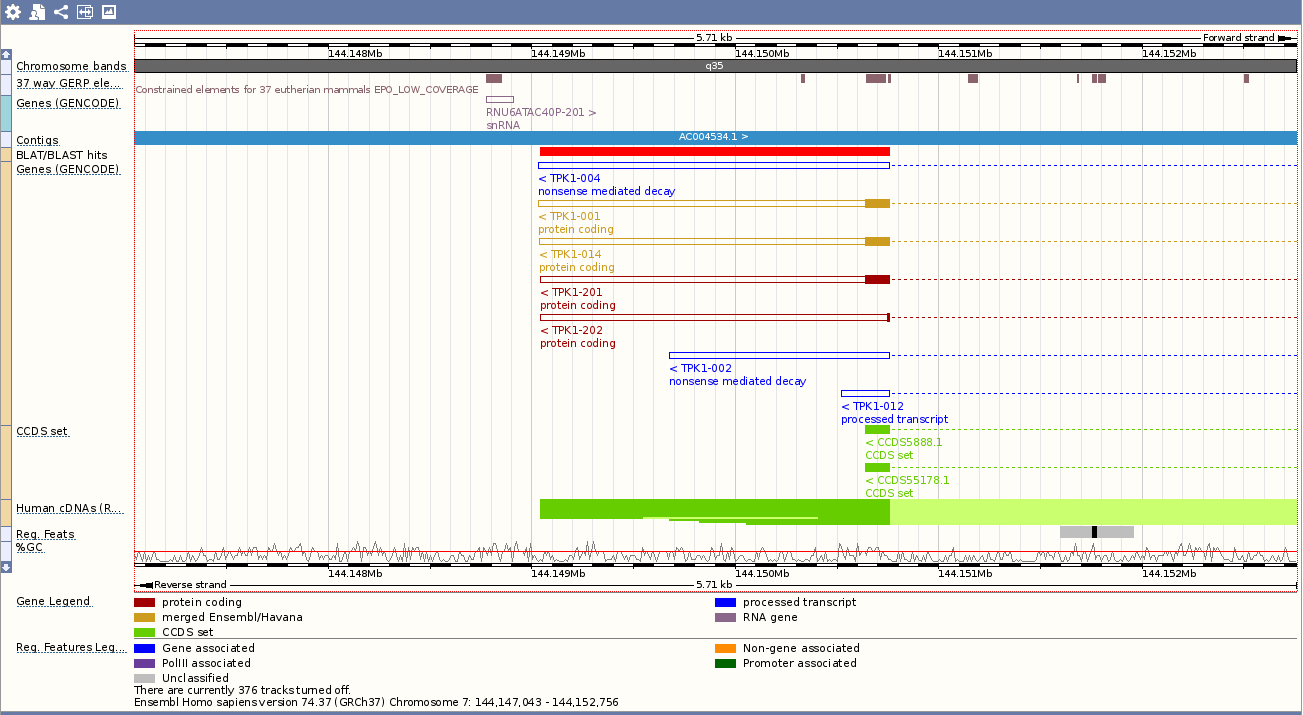
Параметры отбражения можно изменять уже в результатах.