
Пользуясь таксономическим сервисом NCBI, определим, к каким таксонам относятся отобранные бактерии.
Таблица 1. Таксоны отобранных бактерий
| Название | Мнемоника | Таксономия |
| Bacillus subtilis | BACSU | Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus subtilis group |
| Clostridium tetani | CLOTE | Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium |
| Enterococcus faecalis | ENTFA | Firmicutes; Bacilli; Lactobacillales; Enterococcaceae; Enterococcus |
| Finegoldia magna | FINM2 | Firmicutes; Clostridia; Clostridiales; Clostridiales incertae sedis; Clostridiales Family XI. Incertae Sedis; Finegoldia |
| Geobacillus kaustophilus | GEOKA | Firmicutes; Bacilli; Bacillales; Bacillaceae; Geobacillus |
| Staphylococcus aureus | STAA1 | Firmicutes; Bacilli; Bacillales; Staphylococcaceae; Staphylococcus |
| Streptococcus pyogenes | STRP1 | Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Streptococcus |
| Listeria monocytogenes | LISMO | Bacteria; Firmicutes; Bacilli; Bacillales; Listeriaceae; Listeria |
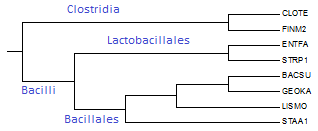
Рисунок 1. Настоящее дерево с подписями крупных таксонов на соответствующих ветвях.
Выберем функцию белков из списка - Фактор элонгации трансляции Ts, мнемоника EFTS. Реконструируем филогенетическое дерево по белкам с этой функцией.
Ниже приведено выравнивание с помощью muscle выбранных белков с заданной функцией.
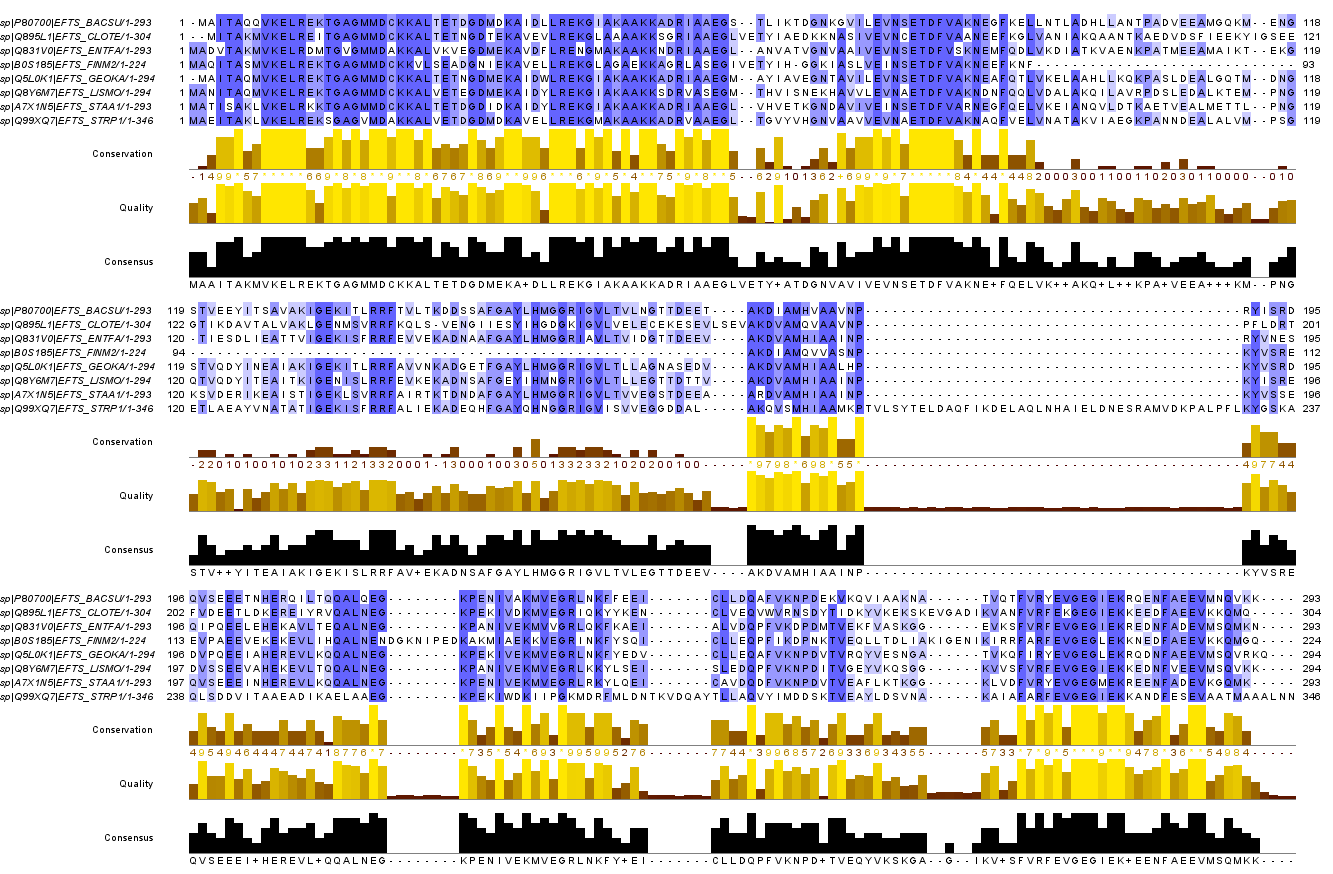
На выравнивнивании выделяются два белка: FINM2 (в середине у него отсутствует участок, соответствующий довольно большому количеству позиций, совпадающих у других белков) и STRP1 (эта последовательность длиннее всех остальных).
Реконструируем филогенетическое дерево четырьмя методами, доступными из JalView, и открываем файлы в Newick-формате с помощью MEGA. Сравним топологию полученных деревьев с топологией правильного дерева.
| Метод построения | Изображение |
| Настоящее | 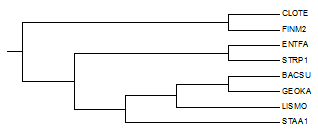 |
Ветви:(CLOTE, FINM2) vs (ENTFA, STRP1, BACSU, GEOKA, LISMO, STAA1) (ENTFA, STRP1) vs (CLOTE, FINM2, BACSU, GEOKA, LISMO, STAA1) (BACSU, GEOKA) vs (CLOTE, FINM2, ENTFA, STRP1, LISMO, STAA1) (BACSU, GEOKA, LISMO) vs (CLOTE, FINM2, ENTFA, STRP1, STAA1) (BACSU, GEOKA, LISMO, STAA1) vs (CLOTE, FINM2, ENTFA, STRP1) |
|
| average distance - identity | 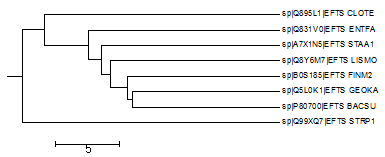 |
Ветви совпадают с реальными:
(BACSU, GEOKA) vs (CLOTE, FINM2, ENTFA, STRP1, LISMO, STAA1) |
|
Есть в новом, но нет в настоящем:
(BACSU, GEOKA, FINM2) vs (CLOTE, LISMO, ENTFA, STRP1, STAA1) (BACSU, GEOKA, LISMO, FINM2) vs (CLOTE, STAA1, ENTFA, STRP1) (BACSU, GEOKA, LISMO, FINM2, STAA1) vs (CLOTE, ENTFA, STRP1) (BACSU, GEOKA, LISMO, FINM2, ENTFA, STAA1) vs (CLOTE, STRP1) |
Есть в настоящем, но нет в новом:
(CLOTE, FINM2) vs (ENTFA, STRP1, BACSU, GEOKA, LISMO, STAA1) (ENTFA, STRP1) vs (CLOTE, FINM2, BACSU, GEOKA, LISMO, STAA1) (BACSU, GEOKA, LISMO) vs (CLOTE, FINM2, ENTFA, STRP1, STAA1) (BACSU, GEOKA, LISMO, STAA1) vs (CLOTE, FINM2, ENTFA, STRP1) |
| average distance - blossum | 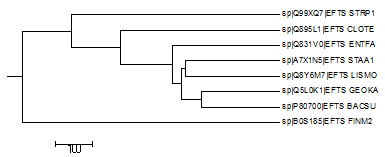 |
Ветви совпадают с реальными:
(BACSU, GEOKA) vs (CLOTE, FINM2, ENTFA, STRP1, LISMO, STAA1) (BACSU, GEOKA, LISMO, STAA1) vs (CLOTE, FINM2, ENTFA, STRP1) |
|
Есть в новом, но нет в настоящем:
(LISMO, STAA1) vs (ENTFA, STRP1, BACSU, GEOKA, CLOTE, FINM2) (BACSU, GEOKA, LISMO, STAA1, ENTFA) vs (CLOTE, FINM2, STRP1) (BACSU, GEOKA, LISMO, STAA1, ENTFA, CLOTE) vs (FINM2, STRP1) |
Есть в настоящем, но нет в новом:
(CLOTE, FINM2) vs (ENTFA, STRP1, BACSU, GEOKA, LISMO, STAA1) (ENTFA, STRP1) vs (CLOTE, FINM2, BACSU, GEOKA, LISMO, STAA1) (BACSU, GEOKA, LISMO) vs (CLOTE, FINM2, ENTFA, STRP1, STAA1) |
| neighbour-joining - identity | 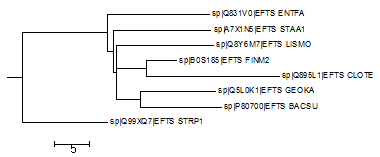 |
Ветви совпадают с реальными:
(BACSU, GEOKA) vs (CLOTE, FINM2, ENTFA, STRP1, LISMO, STAA1) (BACSU, GEOKA, LISMO, FINM2, CLOTE, STAA1) vs (ENTFA, STRP1) (CLOTE, FINM2) vs (ENTFA, STRP1, BACSU, GEOKA, LISMO, STAA1) |
|
Есть в новом, но нет в настоящем:
(BACSU, GEOKA, LISMO, FINM2, CLOTE) vs (STAA1, ENTFA, STRP1) (BACSU, GEOKA, LISMO, FINM2, CLOTE, STAA1, ENTFA) vs (STRP1) |
Есть в настоящем, но нет в новом:
(BACSU, GEOKA, LISMO) vs (CLOTE, FINM2, ENTFA, STRP1, STAA1) (BACSU, GEOKA, LISMO, STAA1) vs (CLOTE, FINM2, ENTFA, STRP1) |
| neighbour-joining - blossum | 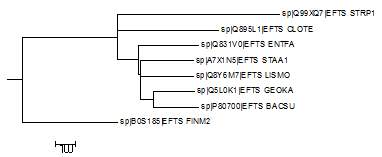 |
Ветви совпадают с реальными:
(BACSU, GEOKA) vs (CLOTE, FINM2, ENTFA, STRP1, LISMO, STAA1) (BACSU, GEOKA, LISMO, STAA1) vs (CLOTE, FINM2, ENTFA, STRP1) |
|
Есть в новом, но нет в настоящем:
(BACSU, GEOKA, LISMO, STAA1, ENTFA) vs (CLOTE, FINM2, STRP1) (BACSU, GEOKA, LISMO, STAA1, ENTFA, CLOTE, STRP1) vs (FINM2) |
Есть в настоящем, но нет в новом:
(CLOTE, FINM2) vs (ENTFA, STRP1, BACSU, GEOKA, LISMO, STAA1) (ENTFA, STRP1) vs (CLOTE, FINM2, BACSU, GEOKA, LISMO, STAA1) (BACSU, GEOKA, LISMO) vs (CLOTE, FINM2, ENTFA, STRP1, STAA1) |
Реконструируем дерево методом "Maximum Parsimony". Укоренили дерево в ветвь STRP1.

Ветви совпадают с реальными:
(BACSU, GEOKA) vs (CLOTE, FINM2, ENTFA, STRP1, LISMO, STAA1) |
|
Есть в новом, но нет в настоящем:
(ENTFA, LISMO) vs (CLOTE, STRP1, BACSU, GEOKA, FINM2, STAA1) (ENTFA, LISMO, STAA1) vs (CLOTE, STRP1, BACSU, GEOKA, FINM2) (ENTFA, LISMO, STAA1, BACSU, GEOKA) vs (CLOTE, STRP1, FINM2) (ENTFA, LISMO, STAA1, BACSU, GEOKA, CLOTE) vs (STRP1, FINM2) (ENTFA, LISMO, STAA1, BACSU, GEOKA, STRP1, FINM2) vs (CLOTE) |
Есть в настоящем, но нет в новом:
(CLOTE, FINM2) vs (ENTFA, STRP1, BACSU, GEOKA, LISMO, STAA1) (ENTFA, STRP1) vs (CLOTE, FINM2, BACSU, GEOKA, LISMO, STAA1) (BACSU, GEOKA, LISMO) vs (CLOTE, FINM2, ENTFA, STRP1, STAA1) (BACSU, GEOKA, LISMO, STAA1) vs (CLOTE, FINM2, ENTFA, STRP1) |
Как ни странно, укоренение не разделило два класса: Bacilli и Clostridia, но отделило ветвь (ENTFA, LISMO, STAA1, BACSU, GEOKA) vs (CLOTE, STRP1, FINM2). Данное дерево нельзя из-за этого считать достоверным.