
С помощью сервиса PDBeFold нашли четыре структурных гомолога цепи A белка 4j3c из разных организмов (4e8b, 1vhy, 3kw2 и 1z85, для всех тоже взяли цепи А). Построили пространственное выравнивание (рис. 3.1).
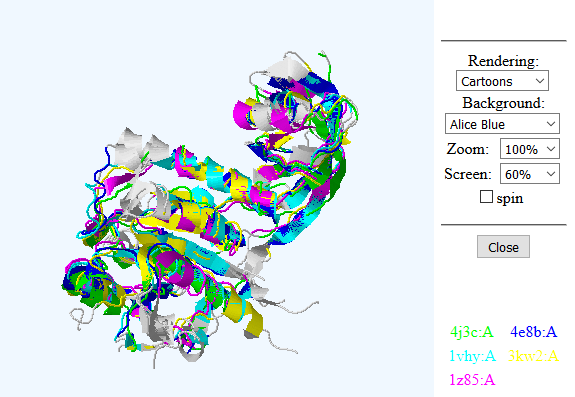
Рис. 3.1. Пространственное выравнивание для 4j3c и гомологов в Jmol, полученное с помощью PDBeFold.
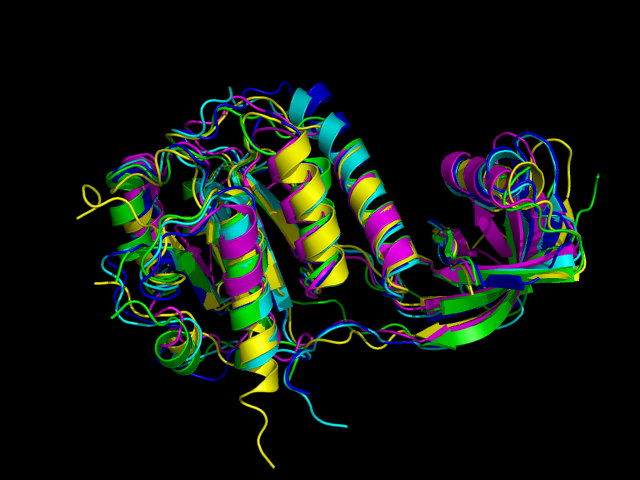
Рис. 3.2. Пространственное выравнивание для 4j3c и гомологов. Изображение получено в PyMOL, цвета структур соответствуют легенде на предыдущем рисунке.
Посмотрели полученное выравнивание в JalView. В получаемых выравниваниях по структуре с помощью PDBeFold только большие буквы соответствуют выровненным.
Рис. 3.3. Структурное выравнивание, полученное с помощью PDBeFold. Окраска по Clustalx.
Выровняли последовательности в JalView с помощью Muscle.
Рис. 3.4. Выравнивание, полученное с помощью алгоритма Muscle. Окраска по Clustalx.
Получили достаточно сильно различающиеся выравнивания. Есть участок (примерно 70-150 остатки 4j3c), где оба выравнивания показали большое сходство последовательностей, однако структурное выравнивание охватывает большее число остатков во всех структурах, чем множественное выравнивание с помощью Muscle.
С помощью PDBeFold (бывш. SSM) осуществили поиск структурных гомологов для домена 1cii A: 284-384. 1cii - это colicin Ia длиной 602 аминокислотных остатка. При стандартных параметрах поиска (искомые структуры должны быть похожи на запрос не менее, чем на 70%) поиск по домену [A: 284-384] длиной 100 аминокислотных остатка не дает 1cii, вероятно, из-за небольшого покрытия (~1/6). Но выбранный домен оказался связывающим доменом colicin Ia, поэтому поиск дает структуру колицинового рецептора E.coli CIR в комплексе с этим доменом.

Рис. 4.1. Результат поиска в PDBeFold.
Ссылка на связывающий домен colicin Ia с CIR рецептором
Выбрали два домена из SCOP, соответствующие альфа- и бета-цепочкам константного домена T-клеточного рецептора из структуры 2bnq (рис. 5.1).
Выделили участки командами, приведенными далее и сохранили участки в отдельном файле.
select alfa, chain d and resi 115-204 select beta, chain e and resi 114-242
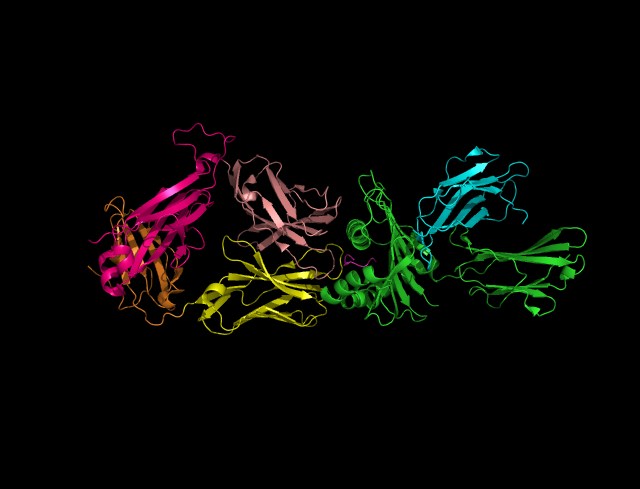
Рис. 5.1. Структура раскрашена по цепям, более ярко показаны выбранные участки (альфа-цепочка d:115-204 - оранжевым, бета-цепочка e:114-242 - ярко-розовым).
Построили карты бета-листов при помощи SheeP (рис. 5.2-5.4). Бета-листу альфа-цепочки соответствует первый (или 0) бета-лист бета-цепочки (смотрим на окружение консервативного цистеина - в зеленой рамочке, в обоих случаях валин и лейцин, а во втором бета-листе - заряженый аргинин и глутамин, другие остатки из окружения тоже играют роль). Попробовали менять местами ряды и колонки (операции не изменяют смысла), считаем, что полученные ориентации бета-листов в цепочках альфа и бета относительно друг друга - правильные.
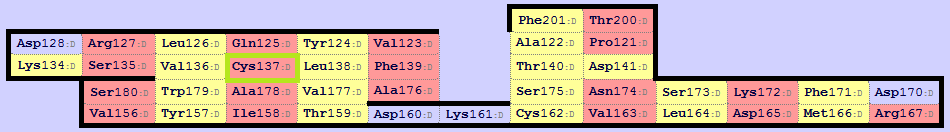
Рис. 5.2. Бета-лист из альфа-цепочки. Консервативный цистеин - в зеленой рамочке.
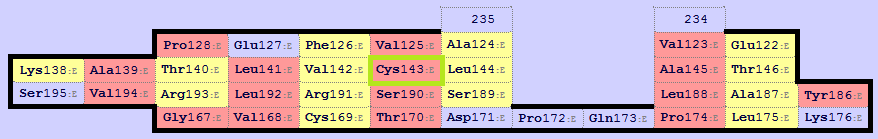
Рис. 5.3. Первый (или 0) бета-лист из бета-цепочки. Консервативный цистеин - в зеленой рамочке.
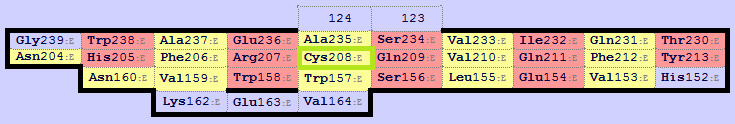
Рис. 5.4. Второй (или 1) бета-лист из бета-цепочки.
Применяли команду для PyMol, которая совмещает структуры цепочек по выбранным атомам:
select set1, alfa and name ca and (resi 136-138 or resi 124-126 or resi 177-179) select set2, beta and name ca and (resi 142-144 or resi 124-126 or resi 189-191) pair_fit set1, set2
Ниже представлены изображения сомещенных структур с разных ракурсов (рис. 5.5-5.7).
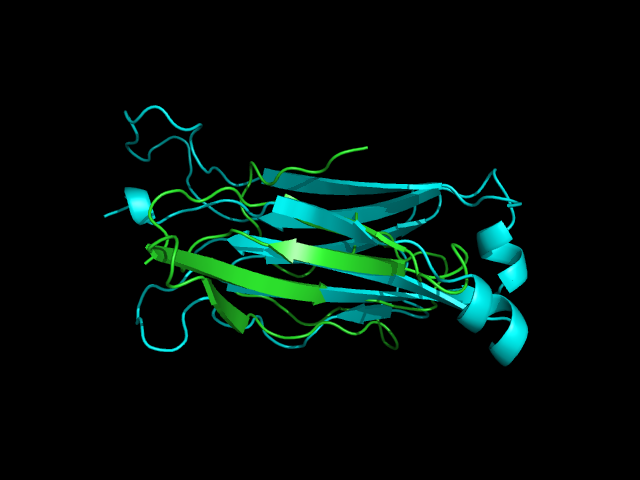
Рис. 5.5. Совмещение бета-листов по остаткам окружающим консервативный остаток цистеина в обоих листах. Здесь и далее: альфа-цепочка окрашена зеленым цветом, бета - голубым.
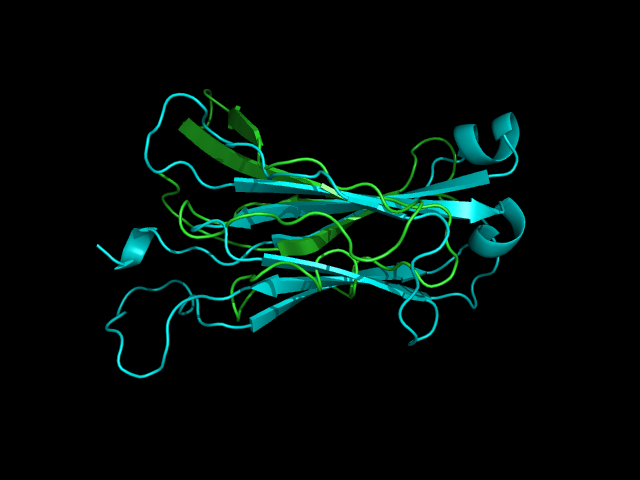
Рис. 5.6. Совмещение бета-листов.
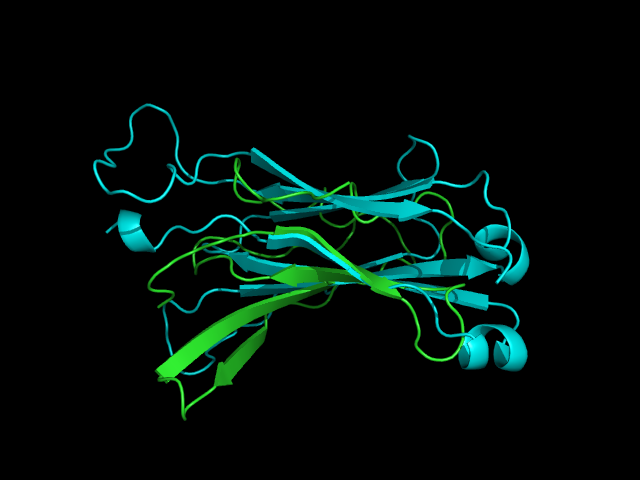
Рис. 5.7. Совмещение бета-листов.
В этих структурах хорошо совмещаются только первые два бета-тяжа, третьи похожи по длине, но не накладываются друг на друга полностью, вне этих листов структуры имеют мало совпадений. Можно, однако, заметить, что неупорядоченные части альфа-цепочки занимают почти то же пространство, что и второй бета-лист бета-цепочки. Направление совмещенных цепей совпадает.