
Выбор структуры: сервис EDS знает PDB-код и имеются подходящие структурные гомологи этого белка (проверили с помощью PDBeFold). Белок из предыдущих семестров не был обнаружен сервисом EDS.
Выбор объекта: функцией метилирования и других модификаций некоторых нуклеотидов является появление устойчивости бактерий к антибиотикам за счет изменения участка связывания антибиотика в рибосоме. рРНК метилтрансфераза RsmE (Ribosomal RNA small subunit methyltransferase E) - слабо изученный фермент, возможно, он играет важную функцию в процессе сборки рибосомы и обеспечении резистентности бактерий к антибиотикам.
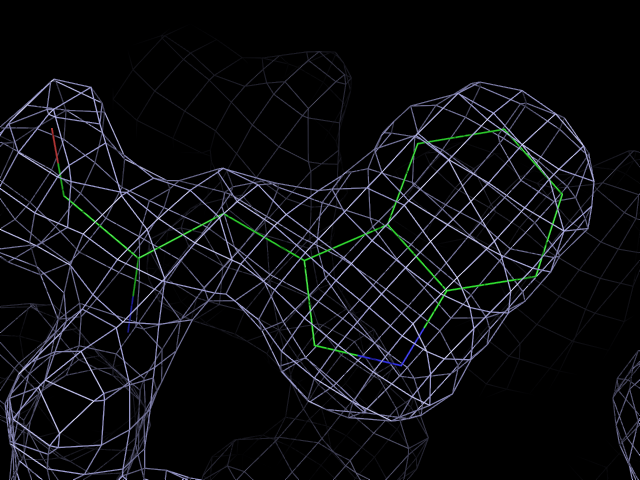
Загрузили PDB-файл и файл с электронной плотностью (ЭП) в PyMol. Использовали команду isomesh для визуализации ЭП цепи A, боковые цепи убрали.
isomesh new_map, 4J3C_map, 1.5, chain a, carve = 2,
где 1.5 - уровень, значения электронной плотности, по которым строится поверхность, carve = 2 - расстояние вокруг атома, на котором рассматривается плотность.
Рис. 1. Изображение электронной плотности вокруг полипептидной цепи.
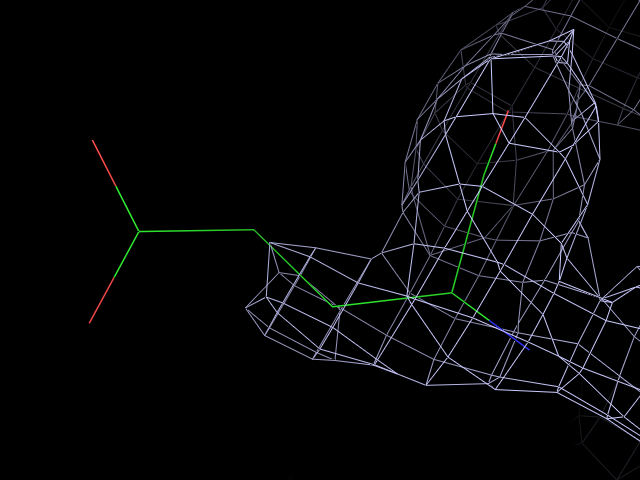
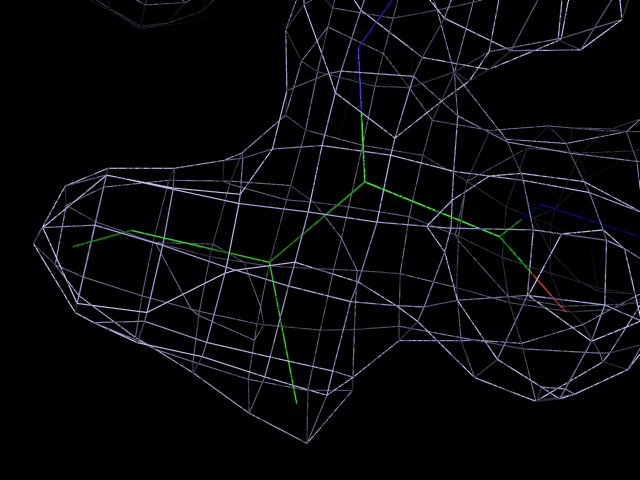
Выбрали остатки, различные по типу (триптофан, глутаминовая кислота и изолейцин). Рассматривали разные уровни подрезки: 0.5, 1.5 и 2.5σ на расстоянии вокруг атомов - 2 Å (carve).
 |
 |
 |
| Рис. 2. Изображение ЭП вокруг триптофана TRP-244 цепи A, уровень подрезки 0.5σ. | Рис. 3. Изображение ЭП вокруг триптофана TRP-244 цепи A, уровень подрезки 1.5σ. | Рис. 4. Изображение ЭП вокруг триптофана TRP-244 цепи A, уровень подрезки 2.5σ. |
Хорошое покрытие.
 |
 |
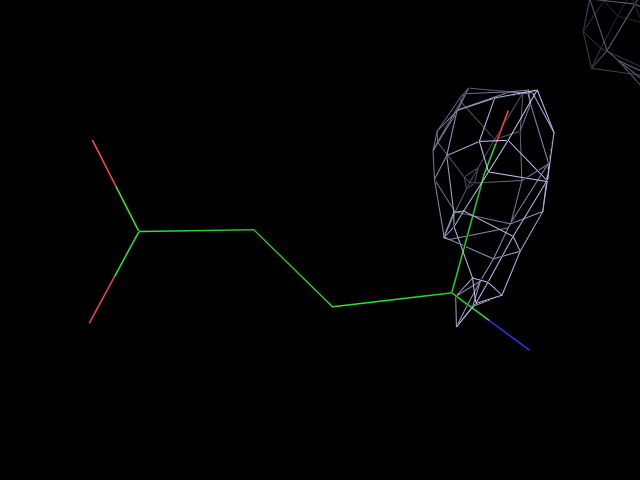 |
| Рис. 2. Изображение ЭП вокруг триптофана GLU-40 цепи A, уровень подрезки 0.5σ. | Рис. 3. Изображение ЭП вокруг триптофана GLU-40 цепи A, уровень подрезки 1.5σ. | Рис. 4. Изображение ЭП вокруг триптофана GLU-40 цепи A, уровень подрезки 2.5σ. |
Недостаточное покрытие на высоких уровнях подрезки.
 |
 |
 |
| Рис. 2. Изображение ЭП вокруг триптофана ILE-133 цепи A, уровень подрезки 0.5σ. | Рис. 3. Изображение ЭП вокруг триптофана ILE-133 цепи A, уровень подрезки 1.5σ. | Рис. 4. Изображение ЭП вокруг триптофана ILE-133 цепи A, уровень подрезки 2.5σ. |
Очень хорошее покрытие.
К сожалению, на данный момент в базе каталитических сайтов CSA не получилось найти остатки каталитического центра данного фермента, поэтому выбирались остатки, визуально расположенные ближе к центру молекулы и дальше от него. Хорошее качество покрытия соответствует координатам атомов аминокислотных остатков, расположенных ближе к центру молекулы. Для остатков, расположенных дальше от центра покрытие чуть хуже, однако качество структуры очень хорошее, так как электронная плотность на уровне 1.5σ хорошо описывает положение большинства атомов полипептидной цепи.