При выполнении этого практикума я использовала аминокислотную последовательность белка beta-lactamase domain-containing protein бактерии Cyanothece sp. PCC 8801. Я искала гомологов этого белка в базе данных Refseq_protein с помошью программы Blast. Для того чтобы найти все все возможные гомологи, в дополнительных параметрах программы Blast я изменила количество находок со 100 на 20000.
Общее число находок — 10151.
Гомологами исходной последовательности можно счиать 7913 находки. Для определения этого числа я использовала
такой условный критерий - найден гомолог всей последовательности, если E-value < 1e-3 и не менее 70% запроса
вошло в полученное выравнивание (Query cover). Сначала я отобрала находки с E-value < 1e-3, затем сохранила
полученные находки и с помошью linux отобрала те, у которых Query cover не менее 70%.
Графическое представление результатов поиска можно увидеть на рис.1.
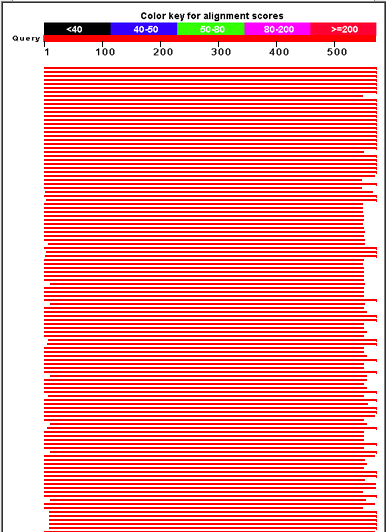
Рис.1 Графическое представление результатов поиска
В таблице 1 представлена информация о лучшей накодке (второй с начала, так как первая — идентичная), о находке
из середины списка и о худшей (последней) находке.
Таблица 1. Характеристика выравниваний.
| Название белка | Организм | Длина выравнивания | Bit score | Процент идентичных остатков, % | Процент сходных остатков, % | E-value | |
| Лучшая находка | Ribonuclease J | Cyanobacterium endosymbiont of Epithemia turgida | 590 | 1019 | 83 | 92 | 0.0 |
| Средняя находка | Zn-dependent hydrolase | Bavariicoccus seileri | 558 | 334 | 33 | 55 | 4e-103 |
| Худшая находка | Integrator complex subunit 11 | Bos taurus | 109 | 39.3 | 28 | 47 | 10.0 |
Ниже на рисунках 2-4 приведены выравнивания, которые были простноены в программе Blast.
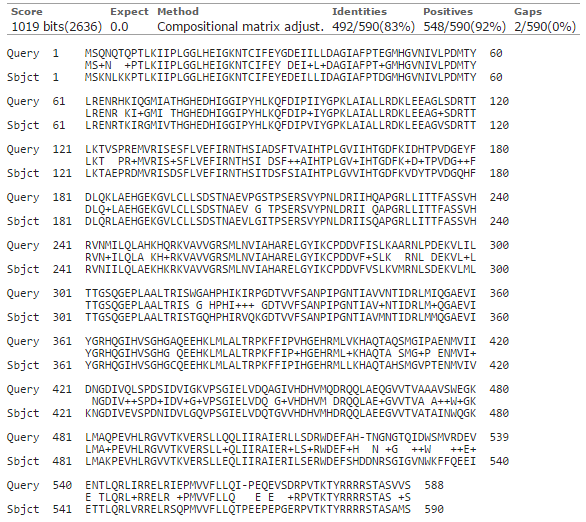
Рис.2 Выравнивание лучшей находки

Рис.3 Выравнивание находки из середины списка
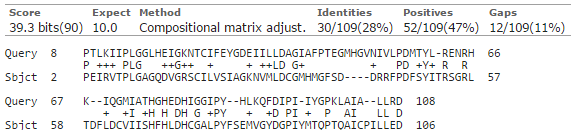
Рис.4 Выравнивание худшей находки
Затем я провела поиск сходных последовательностей среди белков из таксономической группы Bacilli (taxid:91061).
Общее количество находок — 3179. Для сравнения находок из двух поисковых вопросов я выбрала белок
ribonuclease J огранизма Bacillus halodurans. WP_010898811.1. Все параметры, кроме E-value, совпадают.
Выравниваение и score (506) одинаковые. E-value в запросе без указания таксона — 1e-169, а в запросе с
указанием таксона — 9e-171. Скорее всего, это можно объяснить тем, что E-value это параметр, описывающий
количество сходных находок, которые можно случайно найти при поиске в базе данных определенного размера, поэтому чем
больше база данных, тем больше вероятность случайно найти похожую последовательность.
Для построения карты локального сходства (рис. 5) для последовательностей исходного белка и белка hypothetical protein организма
Corynebacterium durum я построила их парное выравнивание в программе Blast. Длина выравнивания 554, Score - 339,
процент идентичных остатков - 36%, а схожих - 56%, E-value - 6e-111. На карте локального сходства прямой линией обозначены
выровненные участки последовательности, места прерываний линии - гэпы. По карте видно, что последовательности
довольно хорошо выровняны.
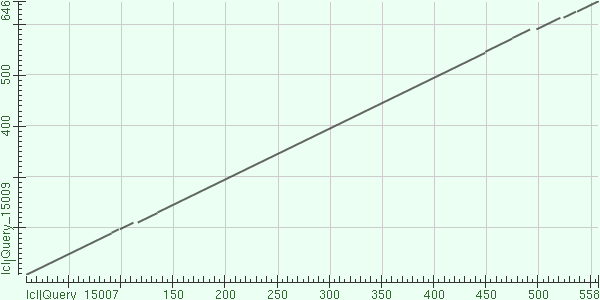
Рис.5 Карта локального сходства белка beta-lactamase domain-containing protein бактерии Cyanothece sp. PCC 8801 и белка hypothetical protein организма Corynebacterium durum.

Рис.6 Выравниванеи белка beta-lactamase domain-containing protein бактерии Cyanothece sp. PCC 8801 и белка hypothetical protein организма Corynebacterium durum.
Для поиска в своей базе данных я использовала Blast. Для начала нужно было создать свою базу данных. Для этого я удалила все гэпы из множественного выравнивания align_03.fasta и проиндексировала его. Затем я осуществила поиск в полученной базе случайно выбранной последовательности (последовательности белка beta-lactamase domain-containing protein бактерии Cyanothece sp. PCC 8801). Нашлось три выравнивания (рис.7).
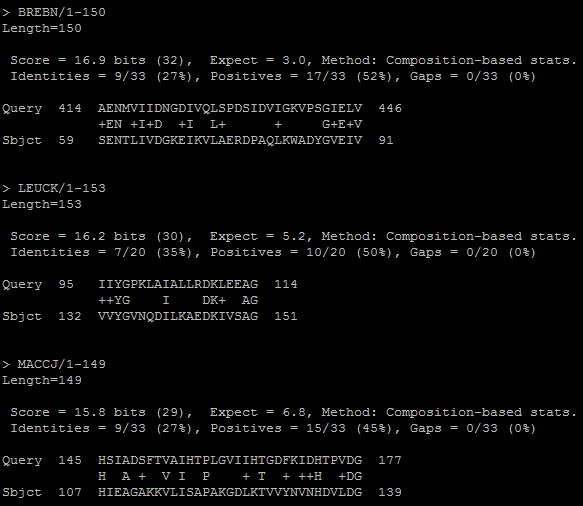
Рис.7 Выравниванеи белка beta-lactamase domain-containing protein бактерии Cyanothece sp. PCC 8801 в новой базе данных.
В таблице 2 предоставлены данные о лучшем выравнивании. Длина выравнивания небольшая, проценты совпадения идентичных и сходных последовательностей низкие, E-value выше порога 0.01. Все эти данные свидетельствуют об отстутствии гомологии между данными последовательностями.
Таблица 2. Данные о лучшем выравнивании.
| Выравнивание | Длина выравнивания | Bit score | Процент идентичных остатков, % | Процент сходных остатков, % | E-value |
| BREBN | 33 | 16.9 | 27 | 52 | 3.0 |
.jpg)