Я строила карту локального сходства бактерии Brucella pinnipedialis B2/94, которая содержит две хромосомы (AC 1 хромосомы: NC_015857; AC 2 хромосомы: NC_015858), и бактерии Brucella melitensis biovar Abortus 2308, которая так же содержит две хромосомы (AC 1 хромосомы: NC_007618; AC 2 хромосомы: NC_007624). Для построения я использовала blast2seq, алгоритм blastn, на сайте NCBI. Полученные карты локального сходства представлены на рис.1-4.
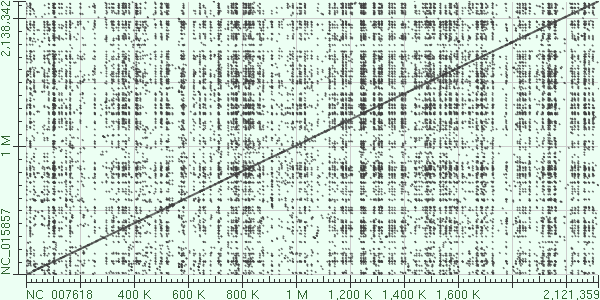
Рис.1 Карта локального сходства первых хромосом бактерий Brucella pinnipedialis B2/94 и Brucella melitensis biovar Abortus 2308.
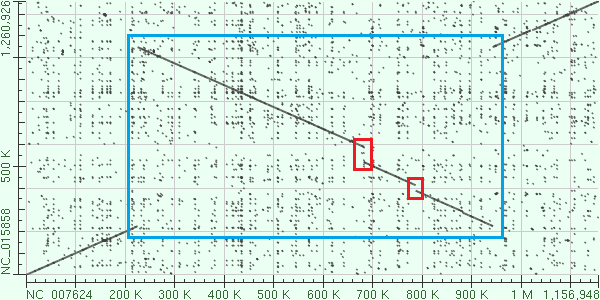
Рис.2 Карта локального сходства вторых хромосом бактерий Brucella pinnipedialis B2/94 и Brucella melitensis biovar Abortus 2308.

Рис.3 Карта локального сходства первой хромосомы бактерии Brucella pinnipedialis B2/94 и второй хромосомы бактерии Brucella melitensis biovar Abortus 2308.

Рис.4 Карта локального сходства второй хромосомы бактерии Brucella pinnipedialis B2/94 и первой хромосомы бактерии Brucella melitensis biovar Abortus 2308.
Из рис.1 видно, что крупных эволюционных событий в первых хромосомах не наблюдалось. По карте локального сходства вторых хромосом можно судить о том, что произошло три крупных эволюционных события. Синей рамкой выделена крупная инверсия, красными рамками выделены делеции, которые, вероятно, произошли во второй хромосоме Brucella melitensis biovar Abortus 2308. Рис.3-4 показывают, что обмена участками между хромосомами не происходило.
Опишисание сходства и различий геномов близкородственных бактерийЯ осуществила построение нуклеотидного пангенома (NPG) с помощью пакета NPG-explorer. Нуклеотидный пангеном - специальный формат для множественного выравнивания геномов, ориентированный на геномы близкородственных бактерий или архей.
В качестве материала я использовала геномы 3-х разных штаммов одного вида Rickettsia rickettsii. Полное название штаммов: Rickettsia rickettsii str. Colombia, Rickettsia rickettsii str. Arizona, Rickettsia rickettsii str. Brazil, Rickettsia rickettsii str. Hauke. Затем я создала файл с информацией откуда брать последовательности геномных ДНК и аннотации генов: genomes.tsv в папке Rickettsia_rickettsii_npg.
Я создала файл npge.conf с параметрами, в котором можно изменять значения параметров, с помощью команды npge -g npge.conf. Затем я скачала и переименовала геномные ДНК при помощи команды npge Prepare. После этого я создала файл examine/identity_recommended.txt с оценкой сходства геномов командой npge Examine. Я заменила параметры WORKERS на 1 и MIN_IDENTITY на 0.899 в соответствии с рекомендацией. Затем я создала нуклеотидный пангеном в файле pangenome/pangenome.bs с помощью команды npge MakePangenome и много файлов с аналитической информацией о пангеноме с помощью команды npge PostProcessing.
Идентификатор блока, например r34x1201, устроен так: r - тип блока (от repeat); 34 фрагмента в блоке; 1201 позиций в выравнивании блока; иногда приходится добавлять "n1", "n2" и т.п. на конце, чтобы сохранить уникальность имен.
Список глобальных блоков - синтений я нашла в global-blocks/blocks.gbi. g-блоки (глобальные блоки) состоят из последовательно идущих во всех геномах s-блоков, перемежающихся блоками других типов (r-, h-, u- и m-).Последовательность глобальных блоков в каждом геноме находится в файле global-blocks/blocks.blocks. Число g-блоков: 2 (g4x1180395, g4x86606). Число промежуточных i-блоков: 3 (i3x3572, i1x572, i1x345). Порядок g-блоков и i-блоков в каждой хромосоме для всех геномов можно увидеть на рис.5. g-блоки g4x1180395 и g4x86606 расположены во всех геномах. Геном штамма Brazil представлен в обратном направлении, поэтому в нем блок g4x86606 находится перед g4x1180395, а не наоборот, как у остальных штаммов. Расположение g- и i-блоков в штаммах Colombia, Arizona, Hauke полностью совпадает.

Рис.5 Порядок g-блоков и i-блоков в каждой хромосоме для всех геномов.
Информацию о s-блоках я нашла в папке pangenome/pangenome.info. s-блоки - стабильные (коровые) блоки, по одному фрагменту из каждого генома. Число s-блоков: 104. Суммарная длина и процент от длины генома в среднем: 1241196 и 98.41%. Сходство геномов - процент консервативных позиций в объединенном выравнивании s-блоков: 0.99865.
R-блоки - блоки с повторами, по крайней мере, в одном геноме. Всего содержится 55 r-блоков. Самый часто повторяющийся r-блок это r21x107. Он повторяется 21 раз: 1 в Arizona, 2 в Brazil, 11 в Colombia и 7 в Hauke. Длина этого блока: 107 позиций. Каждый фрагмент содержит 1 ген. Процент идентичности: 91,58%. Фрагмент выравнивания этого r-блока прелставлен на рис.6.
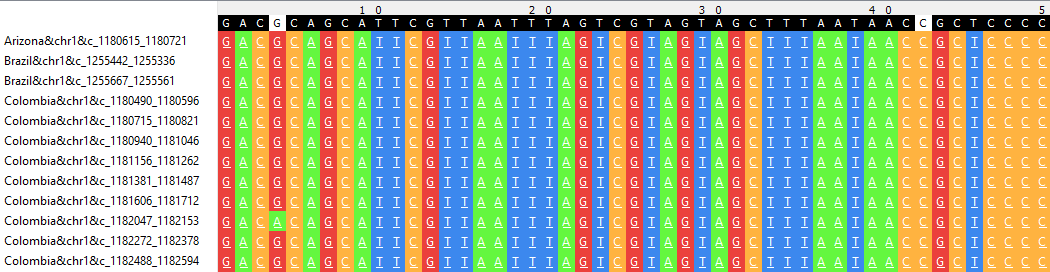
Рис.6 Фрагмент выравнивания r21x107.
H-блоки - "полустабильные" блоки - по одному фрагменту из части геномов, показывают крупных делеции. Самый большой h-block - h3x107. Он есть в штаммах Arizona, Colombia и Hauke и отсутствует в Brazil, это значит, что делеция произошла в штамме Brazil. Процент идентичности этого блока 100%. Фрагмент выравнивания данного h-блока прелставлен на рис.7.

Рис.7 Фрагмент выравнивания h3x107.
U-блоки - это не блоки, а уникальные последовательности из одного генома, у которых нет гомологов среди всех геномов, кроме самой себя. Всего таких блоков два: u1x102 и u1x115. U-блок u1x102 содержится в штамме Brazil, а блок u1x115 содержится в штамме Hauke. С помощью blastn я нашла гомологов последовательности из блока u1x102, находки представлены на рис.8. Странно, что были найдены находки из изучаемых штаммов в качестве гомологов индивидуальной последовательности. На основе полученных находок я не могу сделать вывод в пользу того, что эта последовательность является результатом горизонтального переноса из бактерий другого вида. Последовательность из блока u1x115 выглядит довольно странно: TTT TAC AAG ATT TTT ATC AGT TTT TTT CAT TTT TTG GCA AAA ATA ACC TCA CAG AAT CAA TAG GGG GGC TAT GCG GTA TAA AAT CAA GTA TAG GGG GCT TTT TGT AAA CTT TTTT. Очень много повторов одного нуклеотида. С помощью blastn я нашла гомологов последовательности из блока u1x115. Они представлены на рис.9. Видно, что данная последовательность встречается только у вида Rickettsia rickettsii в разных штаммах, что может говорить о том, что это новая вставка, которая не встречается у других организмов, а штаммы, ее содержащие, возможно, являются самымми ближними родственниками. Лтбо это ошибка секвенирования.
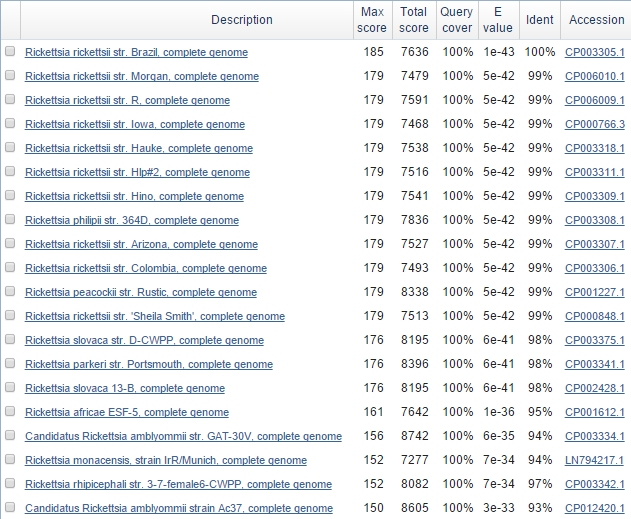
Рис.8 Находки blastn для последовательности из блока u1x102.
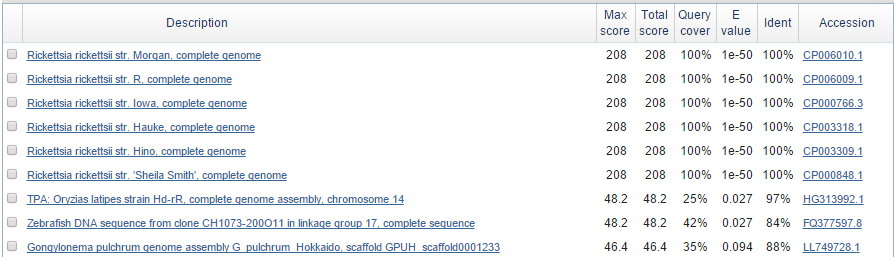
Рис.9 Находки blastn для последовательности из блока u1x115.
.jpg)