Для построения моделей структур A-, B- и Z-формы ДНК я использовала инструменты пакета 3DNA.
Пакет 3DNA один из популярных пакетов программ для анализа и простейшего моделирования структур
нуклеиновых кислот, работающий под операционной системой LINUX. С помощью программы fiber я построила
A-, B- и Z-форму дуплекса ДНК, последовательность одной из нитей которого представляет собой 5 раз
повторенную последовательность "gatc". Результаты работы можно увидеть на рис. 1-3. и в фалах:
gatc_a.pdb,
gatc_b.pdb,
gatc_z.pdb.
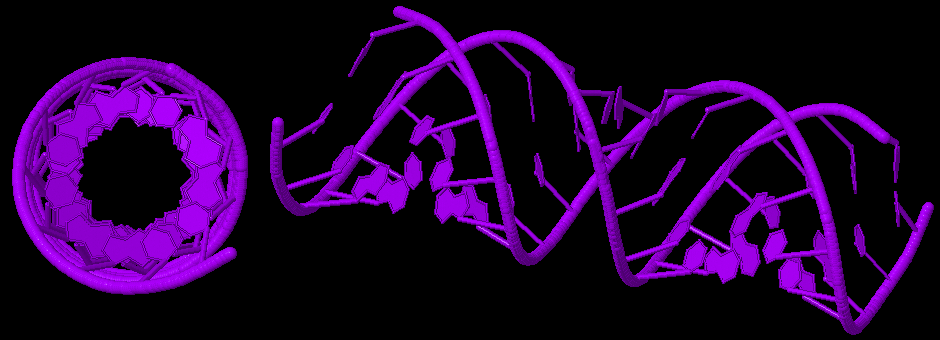
Рис.1 A-форма ДНК.

Рис.2 B-форма ДНК.

Рис.1 Z-форма ДНК.
Для выполнения следующего упражнения я использовала файл А-формы (gatc-a.png). Результат представлен на рис.4.
Салатовым и голубым цветом выделен сахарофосфатный остов ДНК, фиолетовм — азотистые основания всех нуклеотидов,
красным — азотистое основание аденин, зеленым — атом N7 во всех гуанинах.
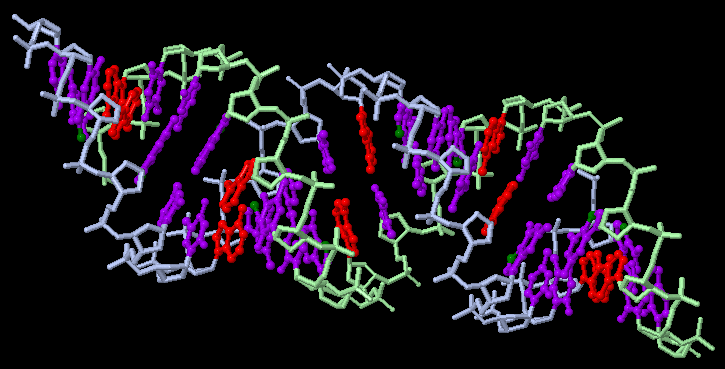
Рис.4 А-форма ДНК. Салатовым и голубым цветом выделен сахарофосфатный остов ДНК, фиолетовм — азотистые основания всех нуклеотидов, красным — азотистое основание аденин, зеленым — атом N7 во всех гуанинах.
Затем я получила файлы PDB тРНК (PDB ID — 1o0b) и ДНК-белкового комплекса (PDB ID — 1p47) (рис.5-6).
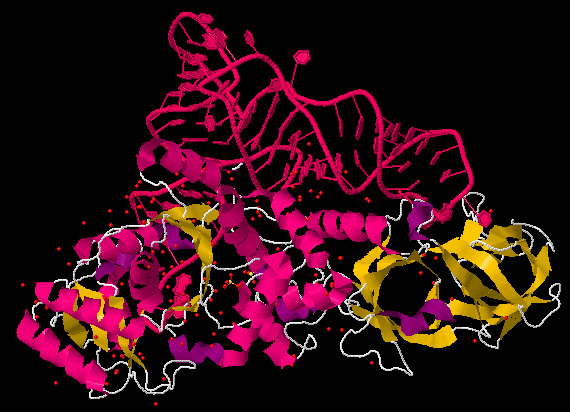
Рис.5 ТРНК (PDB ID — 1o0b).
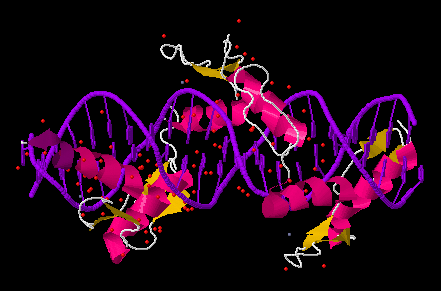
Рис.6 ДНК-белковый комплекс (PDB ID — 1p47).
Наличие разрывов в структурах ДНК и РНК не обнаружено (рис.7-8). Координаты атомов только
РНК и ДНК.

Рис.7 ТРНК.
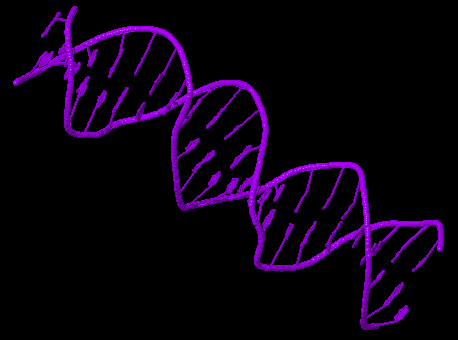
Рис.8 ДНК из ДНК-белкового комплекса.
У тимина (Т31) из В-формы ДНК (из файла gatc-b.pdb) я определила, какие атомы явно обращены в сторону большой бороздки, а какие в сторону
малой. Оказалось, что в сторону большой бороздки обращены атомы C6, C5, C4, O4, C5M; в сторону малой бороздки обращены атомы O2, C2, N1;
оставшийся атом основания N3 (рис. 9). С помощью программы ChemSketch я получила изображение тимина, красным цветом выделены атомы, смотрящие
в сторону большой бороздки, синим — в сторону малой, черным — оставшиеся (рис. 10). В A-форме ДНК ситация обратная: в сторону
большой бороздки обращены атомы O2, C2, N1; в сторону малой бороздки обращены атомы C6, C5, C4, O4, C5M;
оставшийся атом основания N3. В Z-форме нет тимина.
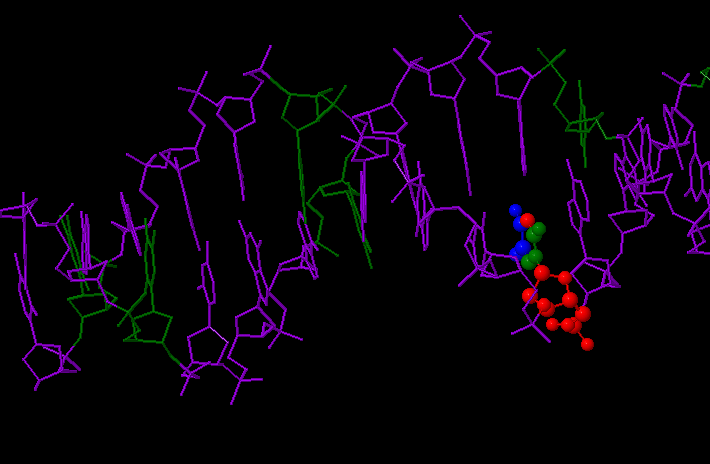
Рис.9 Тимин (Т31) из В-формы ДНК (из файла gatc-b.pdb). Синим цветом выделены атомы, смотрящие в сторону большой бороздки, зеленым — в сторону малой, красным — оставшиеся.
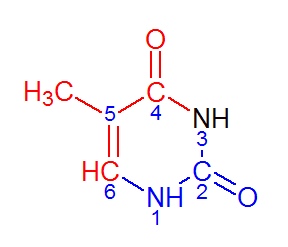
Рис.10 Тимин (Т31) из В-формы ДНК (из файла gatc-b.pdb). Красным цветом выделены атомы, смотрящие в сторону большой бороздки, синим — в сторону малой, черным — оставшиеся.
Результаты сравнения основных спиральных параметров разных форм ДНК приведены в таблице 1. Процесс измерения представлен на рис. 11-12.
Таблица 1. Сравнение основных спиральных параметров разных форм ДНК.
| Форма ДНК | A-форма | B-форма | Z-форма |
| Тип спирали (правая или левая) | Правая | Правая | Левая |
| Шаг спирали, Å | 28,03 | 33,75 | 43,50 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 7,98 ([C]28:B.P-[T]7:A.P) | 17,21 ([T]35:B.P-[A]10:A.P) | 9,87 ([G]11:A.P-[C]34:B.P) |
| Ширина малой бороздки | 16,81 ([T]15:A.P-[C]28:B.P) | 11,96 ([A]10:A.P-[C]28:B.P) | 16,08 ([C]12:A.P-[C]28:B.P) |
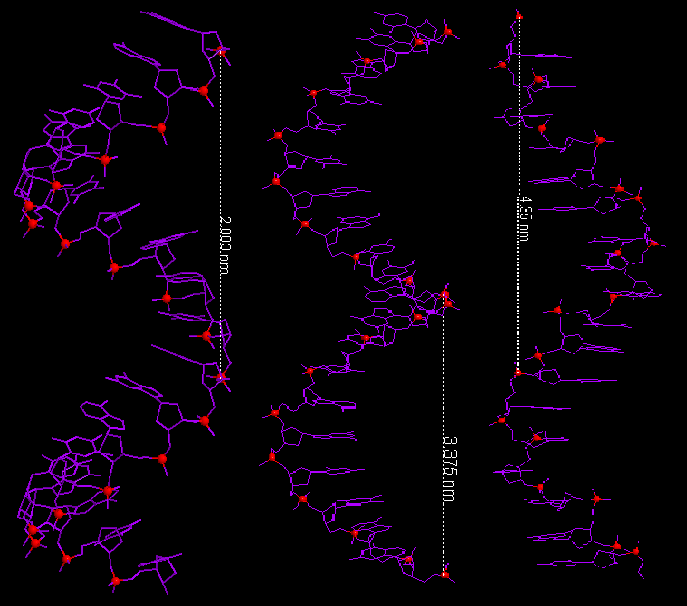
Рис.11 Измерение шага спирали в A-форме (слева), в В-форме (посередине) и Z-форме (справа)
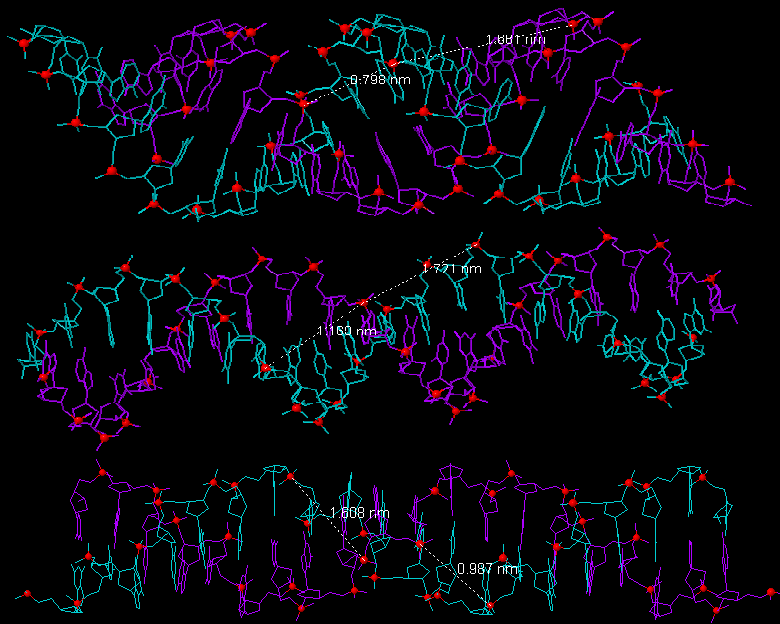
Рис.12 Измерение большой и малой бороздки в A-форме (сверху), в В-форме (посередине) и Z-форме (снизу)
Для сравнения торсионных углов тимина (Т31) в структурах А- и В-форм я использовала поманду Settings->Torsion JMol. Сравнение значений углов в А- и В-форме со значениями, приведенными в презентации, представлено в таблице 2. Процесс измерения торсионных углов изобажен на рис.13.
Таблица 2. Результаты измерения торсионных углов тимина (Т31) в А- и В-форме.
| Форма ДНК | α | β | γ | δ | ε | ζ | χ |
| A-форма (презентация) | 62 | 173 | 52 | 88/3 | 178 | -50 | -160 |
| А-форма (измерение) | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| B-форма (презентация) | 63 | 171 | 54 | 123/131 | 155 | -90 | -117 |
| B-форма (измерение) | -29.9 | 136.3 | 31.2 | 143.3 | -140.8 | -160.5 | -98.0 |
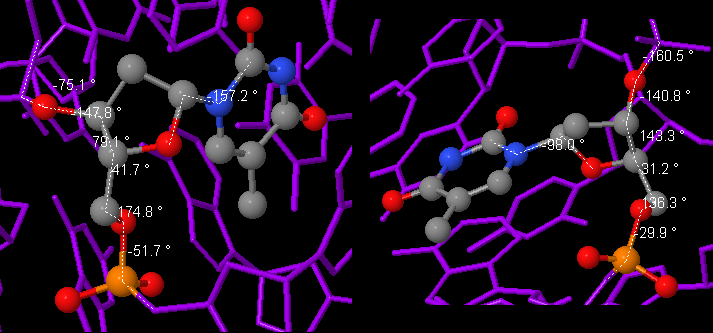
Рис.13 Измерение торсионных углов у тимина (Т31) в A-форме (слева) и в В-форме (справа).
Параметры структур нуклеиновых кислот я определяла с помощью программ пакета 3DNA. Пакет 3DNA пока работает только со старым форматом PDB. Для перевода файлов в старый формат я использовала программу remediator, установленную на kodomo. Синтаксис: remediator --old ''XXXX.pdb'' > ''XXXX_old.pdb.
Для анализа структур нуклеиновых кислот z использовала программы find_pair и analyze. Программа find_pair определяет спаренные основания и положения спиралей в структуре. Синтаксис: find_pair -t XXXX.pdb XXXX.fp. Полученные данные необходимы для работы analyze. Я перенаправить результат работы find_pair на вход программе analyze: find_pair -t XXXX.pdb stdout | analyze. В результате был создан ряд файлов с описанием разных параметров структуры, в файле XXXX.out я нашла описание водородных связей и значения всех торсионных углов.
Значения торсионных углов в структурах А- и В-форм ДНК совпадают со значениями, полученными при измерении торсионных углов в программе Jmol. Значения торсионных углов в Z-форме ДНК сильно различаются для гуанина и цитозина (таблица 3).
Таблица 3. Значения торсионных углов гуанина и цитозина в Z-форме.
| Форма ДНК | α | β | γ | δ | ε | ζ | χ |
| Z-форма (G) | -139.5 | -136.7 | 50.9 | 137.6 | -96.5 | 81.9 | -154.3 |
| Z-форма (C) | 52.0 | 179.0 | -173.8 | 94.9 | -103.6 | -64.8 | 58.7 |
Для определения средних значений каждого из торсионных углов в заданной структуре ДНК и тРНК я использовала программу Excel. Краевые нуклеотиды и нуклеотиды, у которых есть не все значения торсионных углов, я не учитывала. Также я определила номер самого "деформированного" нуклеотида. Результат представлен в таблицах 4-5.
Таблица 4. Средние значения торсионных углов в ДНК-белковом комплексе (из 1p47).
| Нуклеотид | α | β | γ | δ | ε | ζ | χ |
| A | -58.05 | 161.3 | 49.7 | 104.95 | -175.2 | -86.3 | -130.45 |
| C | -15.18 | 67.71 | -1.74 | 129.31 | -95.16 | -99.2 | -122.22 |
| G | -58.22 | 21.29 | 38.84 | 141.25 | -115.39 | -97.03 | -115.64 |
| T | -40.4 | 159.4 | 24.45 | 138.55 | -129.3 | -172 | -105.25 |
| C[7] наиболее "деформированный" | 100.1 | -163.6 | -141.9 | 136.4 | -155 | -91.3 | -135.6 |
| Для всех нуклеотидов | -38.02 | 56.69 | 20.5 | 133.86 | -110.22 | -101.38 | -118.81 |
Полученные средние значения торсионных углов РНК мало похожи на значения углов в A-, В- и Z-форме ДНК, но больше всего напоминают на А-форму.
Таблица 5. Средние значения торсионных углов в тРНК (из 1o0b).
| Нуклеотид | α | β | γ | δ | ε | ζ | χ |
| A | -18.32 | 55.82 | 67.92 | 82.83 | -152.70 | -75.57 | -157.67 |
| C | -32.63 | 34.32 | 12.89 | 82.59 | -147.82 | -76.35 | -131.48 |
| G | 13.23 | 56.89 | 3.78 | 80.86 | -148.60 | -71.33 | -139.63 |
| U | -37.95 | 32.09 | 17.69 | 83.24 | -99.13 | -71.60 | -111.10 |
| A[26] наиболее "деформированный" | 174.8 | 170.5 | 172.2 | 90.5 | -119.7 | -62.8 | -170 |
| Для всех нуклеотидов | -16.44 | 44.70 | 19.76 | 82.19 | -139.65 | -73.70 | -134.51 |
| А-форма (измерение) | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| B-форма (измерение) | -29.9 | 136.3 | 31.2 | 143.3 | -140.8 | -160.5 | -98.0 |
Информацию о водородных связых между нуклеотидами тРНК можно посмотреть в файле, полученном с помошью программ find_pair и analyze. На рис.14 рамкой выделены те нуклеотиды, которые образуют стебли (stems) во вторичной структуре тРНК. Оставшиеся дополнительные водородные связи в тРНК, по-видимому, стабилизируют ее третичную структуру. Неканонические пары оснований в структуре тРНК выделены рамкой на рис.15.

Рис.14 Нуклеотиды, образующие стебли в тРНК.

Рис.15 Неканонические взаимодействия нуклеотидов.
В файл staking.pdb записаны преобразованные координаты всех динуклеотидных пар, их используют для получения общепринятого изображения стекинг-взаимодействий. Для получения такого изображения нужно в исходном файле PDB определить номера остатков, между которыми предполагается стекинг-взаимодействие. Я искала возможные стекинг-взаимодействия нуклеотидов тРНК. На рис.16 выделены оранжевым пары оснований с максимальной площадью перекрывания, а фиолетовым - с минимальной.
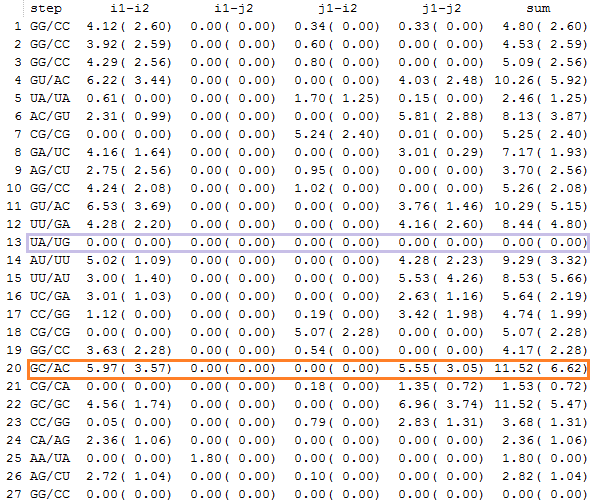
Рис.16 Площадь перекрывания пар оснований. Оранжевым выделены пары оснований с максимальной площадью
перекрывания, фиолетовым - с минимальной.
В файле staking.pdb я нашла номер структуры, описывающей координаты нуклеотидных остатков, короные имеют наибольшую и наиманьшую площадь перекрывания, затем я вырезала эту структуру в отдельный файл с помощью команды ex_str -Х stacking.pdb stepХ.pdb и построила изображение с помощью команды stack2img -cdolt stepХ.pdb stepХ.ps. Получившийся ps-файл я открыла с помощью программы Ghostscript и GSview. Результат представлен на рис.17-18.
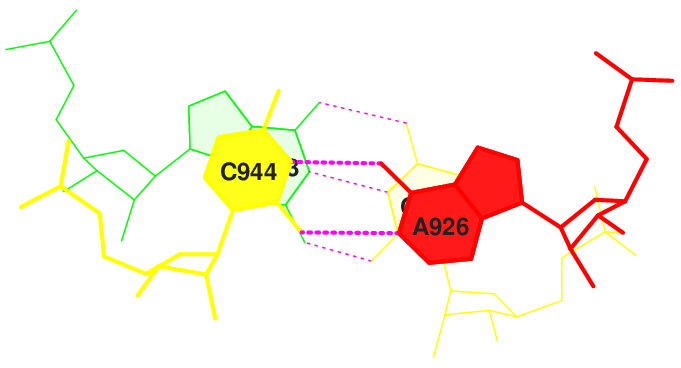
Рис.17 Изображение стекинг-взаимодействия между парами оснований с максимальной площадью перекрывания.

Рис.18 Изображение стекинг-взаимодействия между парами оснований с максимальной площадью перекрывания.
.jpg)