По результатам хроматограмм из капиллярного секвенатора по Сэнгеру и последовательности, сгенерированной программой,
нужно было получить последовательность фрагмента ДНК.
Капиллярный секвенатор по Сангеру выдает файлы с хроматограммой и последовательностью в формате XXX.ab1.
Для просмотра и редактирования этих файлов я использовала программу Chromas Lite.
Исходные файлы:
прямая цепь;
обратная цепь.
Я открыла файл с прямой последовательностью (файл WSWS2933_H3_F_C09_2013-06-11-22-39-58.ab1) в программе Chromas Lite,
вызвала еще раз Chromas Lite, открыла файл с обратной последовательностью (файл WSWS2933_H3_R_C10_2013-06-11-22-39-58.ab1)
и перешла к комплементарной цепочке с помощью "Reverse+Comlement". Затем я выровняла две хроматограммы с использованием
поиска подслов (Find...).
Границы не читаемого 5'-участка в прямой последовательности (координаты определены по прямой последовательности):
1-25.
Границы не читаемого 3'-участка в прямой последовательности (координаты определены по прямой последовательности):
379-384.
Границы не читаемого 3'-участка в обратной последовательности (координаты определены по прямой (перевернутой) последовательности):
1-5
Границы не читаемого 5'-участка в обратной последовательности (координаты определены по прямой (перевернутой) последовательности):
356-382.
26-я координата в прямой последовательности соответствует 56-ой координате в обратной перевернутой последовательности, а 325-я — 325-ой координате в обратной перевернутой последовательности. Значит, что начало и конец чтения одной последовательности соответствует прочтенному фрагменту в другой (обратной) последовательности (рис.1).
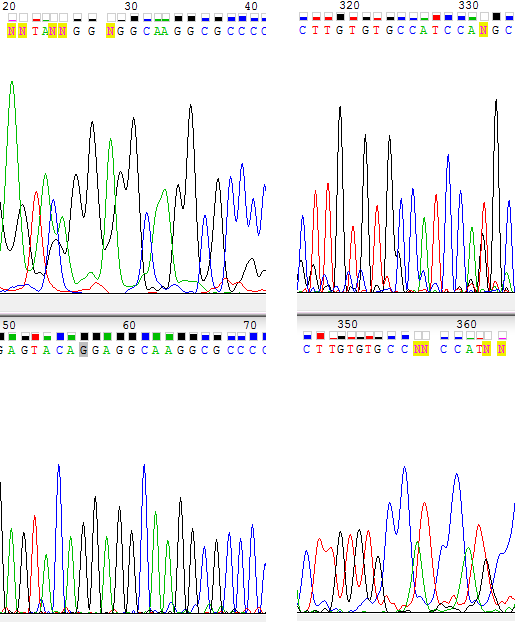
Рис.1 Соответствие начала (слева) и конца (справа) читаемых фрагментов последовательности в прямой (сверху) и обратной (снизу) цепей.
Оценка качества хроматограммы:
- прямая цепь:
- сигнал превосходит шум в среднем в 7 раз;
- средняя сила сигнала и шума вдоль оси X равномерна;
- сила у разных очевидных сигналов различается максимум в четыре раза, в основном самые сильные сигналы дают нуклеотиды A и G, но в среднем сила сигналов не зависит от нуклеотида;
- качество хроматограммы хорошее;
- обратная цепь:
- сигнал превосходит шум в среднем в 13 раз;
- средняя сила сигнала и шума вдоль оси X равномерна;
- сила у разных очевидных сигналов различается максимум в пять раза, в основном самые сильные сигналы дают нуклеотиды T и C, но в среднем сила сигналов не зависит от нуклеотида;
- качество хроматограммы хорошее
Для редактирования последовательности я включила опцию "Continuous edit". Затем я удалила не читаемые 5'- и 3'- концы, из-за этого изменилась нумерация нуклеотидов. Далее приведены измененные координаты нуклеотидов.
Я просмотрела прямую и обратную последовательность и отредактировала их. На рис. 2 представлен фрагмент последовательности, в которой нет проблем с прочтением последовательности нуклеотидов. На рис. 3 изображены основные проблемы, с которыми я столкнулась при исправлении полученной последовательности нуклеотидов. При возникновении проблемы в определении нуклеотида я смотрела на обратную последовательность, и, если сигнал был хорошим, выбирала тот нуклеотид, который на перевернутой. На рис. 3 А в 59-й координате шум выше среднего уровня или почти как сигнал. На рис. 3 С в 245-249-ых координатах (по прямой цепи) в обратной цепи вместо двух-трех пиков — один широкий. На рис. 3 В и D в 87-й и в 108-й координатах на обеих цепях одинаковый уровень сигнала Т и С, что может говорить о том, что в этой позиции последовательности полиморфизм. На рис. 3 Е в 346-й координате пик на нетипичном расстоянии от соседей - соседние пики нетипично удалены (обратной цепи на данном фрагменте нет).
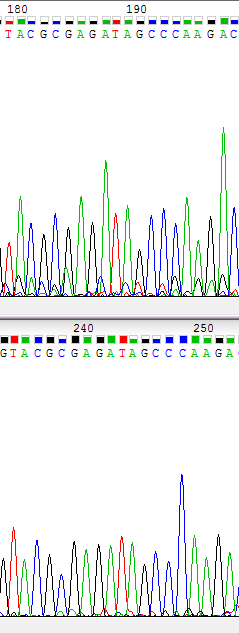
Рис.2 Фрагмент последовательности хорошего качества в прямой (сверху) и обратной (снизу) цепях.
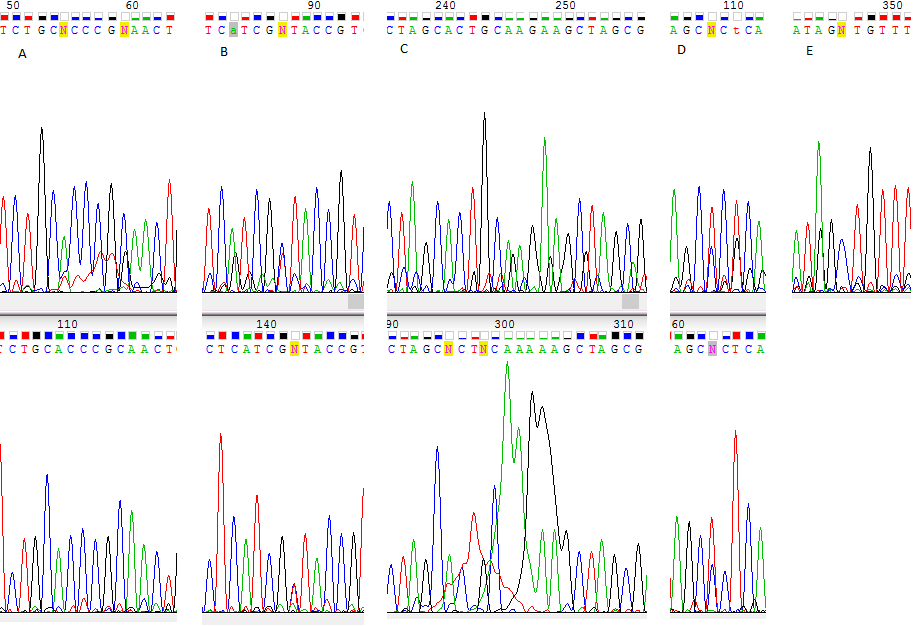
Рис.3 Проблемы в определении нуклеотида в прямой (сверху) и обратной (снизу) цепях.
Нуклеотиды, измененные мной по сравнению с последовательностью в исходных файлах, указаны маленькими буквами в выравнивании прямой и обратной последовательности.
Выравнивание прямой и комплементарной к обратной последовательности с отмеченными измененными нуклеотидами в проекте JalView:
align.jvp.
Файл в формате fasta с "чистой" прочтенной последовательностью:
WS2933.fasta.
"Чистая" - значит, проверен каждый нуклеотид и вынесено решение о том, какой он в анализируемой ДНК.
На рис. 4 представлен пример не читаеммой хроматограммы: NN_G10.ab1. Средний сигнал практически не превосходит сигнал шума.
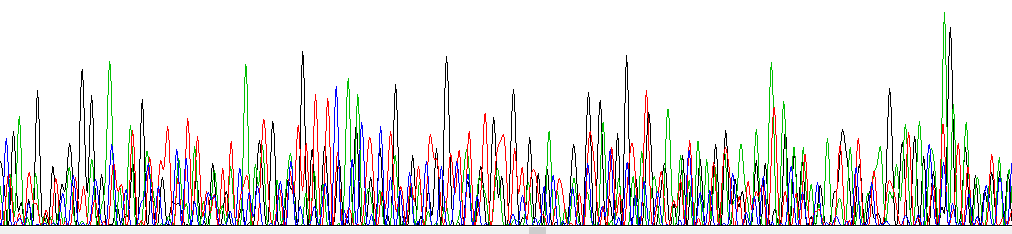
Рис.4 Не читаемая хроматограмма.
.jpg)