Для того, чтобы установить какому гену принадлежит последовательность, полученная в практикуме 6 (результат чтения хроматограммы), и таксономию организма, я воспользовалась программой BLASTN, которая ищет сходство последовательности нуклеиновых кислот по базе данных нуклеиновых кислот (в данном случае по базе Nucleotide collection (nr/nt)). Выбрала алгоритм Somewhat similar sequences (blastn), параметры поиска оставила стандартными, кроме Max target sequences, там выбрала 5000.
Часть результата работы программы представлена на рис.1. Резкий скачек в E-value наблюдается только у первой находки (между 1-ой и 2-ой находкой скачек E-value 1e-160 до 3e-105), аналогично score и процент идентичности резко падает только между 1-ой и 2-ой находкой. Из этого можно сделать вывод, что 1-ая находка является лучшей, а остальные примерно однаковы между собой. Для дальнейшего анализа я выбрала первые 3 находки.
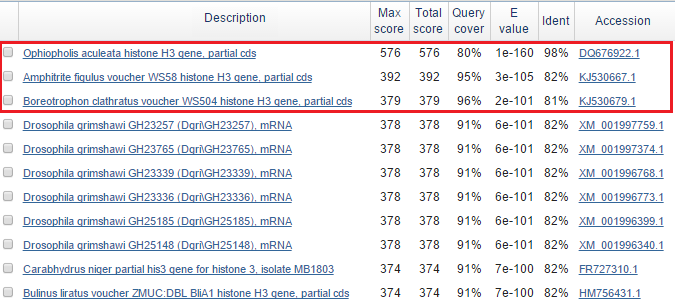
Рис.1 Результат работы программы BLASTN.
Из таблицы находок видно, что последовательность принадлежит гену, который кодирует 3 гистон (H3). Определить принадлежность последовательности определенному виду не удается. На рис.2 показаны таксоны, которым принадлежит наибольшее число находок. Наибольшее число находок принадлежит таксону Bilateria, в котором наибольшее количсетво находок разделено между таксонами Deuterostomia и Protostomia. Лучшая находка принадлежит организму Ophiopholis aculeata (рис.3). 2-ая находка принадлежит организму Amphitrite figulus (рис.4), а 3-ая - Boreotrophon clathratus (рис.5). Уровень сходства с лучшей находкой (вид Ophiopholis aculeata) - 2 замены на 100 п.н. и с лучшей находкой из другого вида (Amphitrite figulus) - 18 замен на 100 п.н.
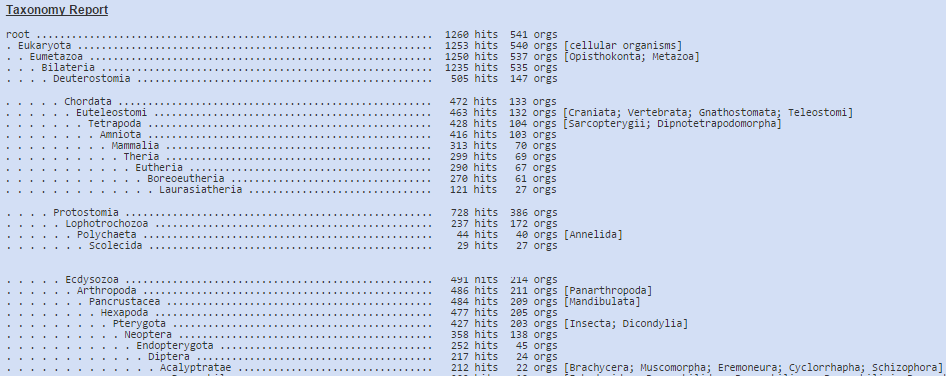
Рис.2 Таксономия организмов из результата работы программы BLASTN.

Рис.3 Ophiopholis aculeata.

Рис.4 Amphitrite figulus.
Рис.5 Boreotrophon clathratus.
В программе Jalview я построила выравнивание входной последовательности с 3-мя лучшими находками.
Для сравнения списка находок нуклеотидной последовательности 3-я разными алгоритмами blast я использовала последовательность из предыдущего задания. Я ограничила облать поиска таксоном Eleutherozoa (taxid:133551), к которому относится лучшая находка алгоритма blastn, так, чтобы находок blastn было от 10 до 1000 и схожство находок было заметно разным. Я осуществила поиск данной последовательности по очереди тремя алгоритмами: blastn, megablast, discontiguous megablast, остальные параметры поиска были одинаковыми. Сравнение результата поиска алгоритмов приведено в таблице 1. Верхние и нижние части результата выдачи алгоритмов представлены на рис.6-8.
Таблица 1. Сравнение результата поиска последовательности алгоритмами blastn, megablast и discontiguous megablast.
| Алгоритм | Число находок | E-value худшей находки | Сходство худшей находки |
| blastn | 399 | 3.0 | 91% |
| megablast | 11 | 1-е61 | 79% |
| discontiguous megablast | 393 | 4e-17 | 80% |
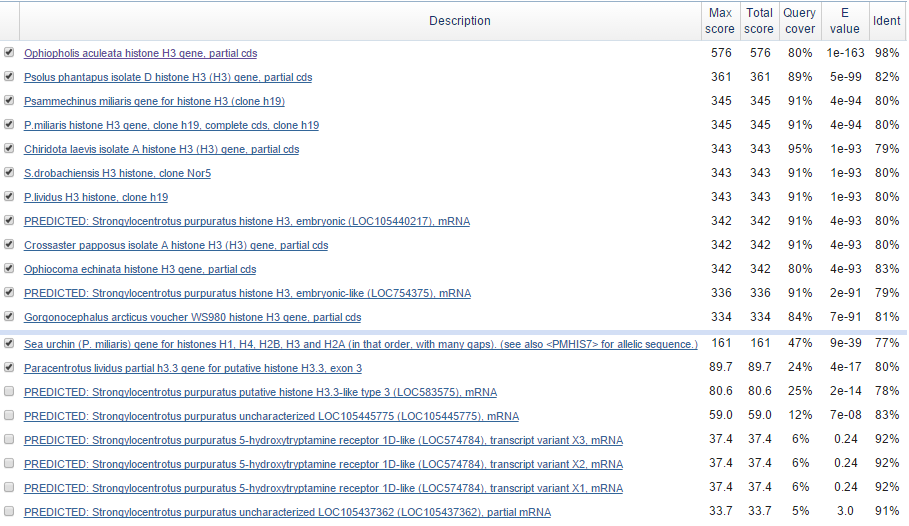
Рис.6 Результат работы алгоритма blastn.
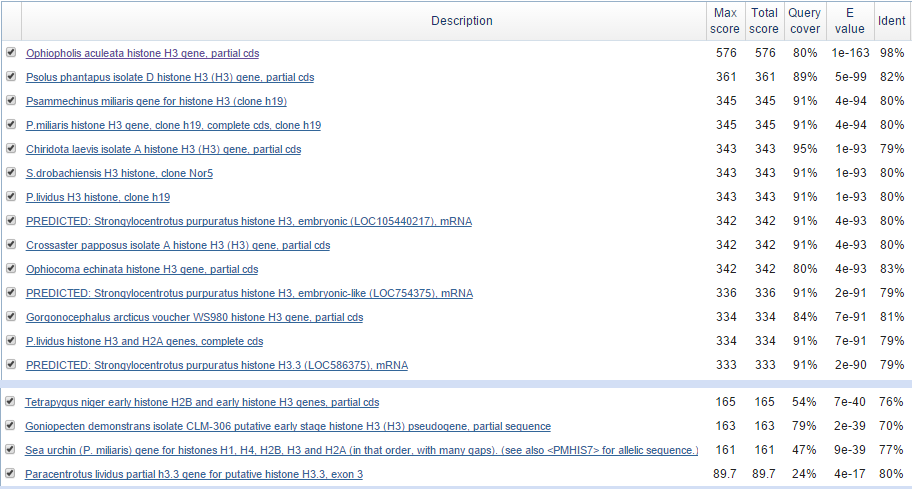
Рис.7 Результат работы алгоритма discontiguous megablast.
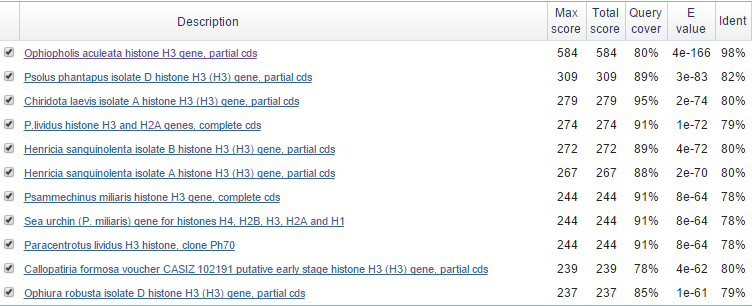
Рис.8 Результат работы алгоритма megablast.
Как видно из таблицы 1 и рис.6-8, алгоритмы выдали различающиеся находки. Алгоритм megablast нашел только самых близких гомологов (11 находок), так как его ХЭШ-таблица содержит слова длиной в 28 букв, в то время как в других алгоритмах содержатся слова диной в 11 букв. E-value в megablast самое низкое, что говорит о том, что все находки достоверны.
На рис.6-7 галочками обозначены находки, найденные и аглоритмом blastn, и алгоритмом discontiguous megablast. Оставшиеся 6 находок из алгоритма blastn не нашлись алгоритмом discontiguous megablast, веротно, потому что эти находки слишком короткие (покрытие менее 25%) и нет достаточно веских оснований считать их гомологами данной последовательности. Выравнивание двух из таких последовательностей с исходной представлены на рис.9.
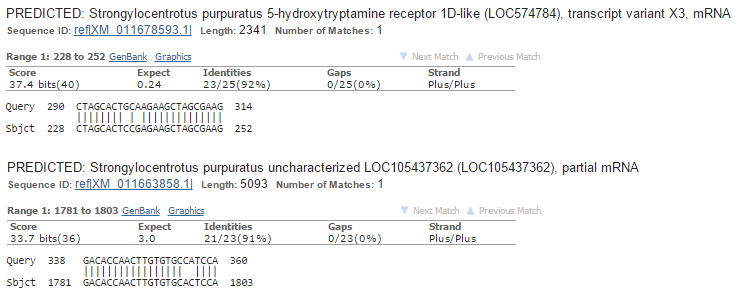
Рис.9 Находки, найденные алгоритмом blastn, но не найденные алгоритмомами discontiguous megablast и megablast.
Я проверила наличие некоторых белков в организме X5 (Amoboaphelidium) - сборка генома X5. Белки нужно было выбрать на свое усмотрение из тех, которые, по моему мнению, должны быть почти у всех эукариот. Белки, которые я выбрала, представлены в таблице 2.
Таблица 2. Название и краткое описание функций выбранных белков.
| Запись в UniProt | AC | Название белка | Функция |
| TERT_HUMAN | O14746 | Telomerase reverse transcriptase | Катализирующий синтез ДНК на матрице РНК в процессе, называемом обратной транскрипцией, что приводит к удлинению теломер. |
| H3_ENTHI | Q06196 | Histone H3 | Основной компонент нуклеосом. Компактизирует ДНК в хроматин, ограничивая доступ к ДНК клеточных механизмов, которым нужна ДНК в качестве матрицы. |
| H4_HUMAN | P62805 | Histone H4 | -"- |
| H2B_EUPCR | O97484 | Histone H2B | -"- |
| H2A_DROME | P84051 | Histone H2A | -"- |
Для того, чтобы проверить наличие этих белков в организме Amoboaphelidium, я использовала поиск белковой последовательности против трансляции нуклеотидного банка данных в шести рамках. Для этого я установила локальный бласт (standalone blast). С помощью программы makeblastdb я создала локальную базу данных. Команда для создания базы: makeblastdb -in X5.fasta -dbtype nucl.
Затем я использовала программу tblastn для поиска гомолога белка в формальной трансляции нуклеотидного банка. Программа приняла файл all.fasta, в котором находятся последовательности выбранных белков в fasta-формате. Команда: tblastn -query all.fasta -db X5.fasta -out tblastn.out -outfmt 7. Полученный файл: tblastn.out. Результаты приведены в таблице 3. Хорошими находками я назвала те, которые предположительно свидетельствуют о гомологии (E-value < 1e-05).
Таблица 3. Результаты поиска tblastn.
| Запись в UniProt | Общее число находок | Число хороших находок | Запись лучшей находки | E-value лучшей находки | Процент идентичности лучшей находки, % | Покрытие входной последовательности лучшей находки, % |
| TERT_HUMAN | 3 | 2 | scaffold-17 | 8e-23 | 26.58 | 50.18 |
| H3_ENTHI | 5 | 5 | unplaced-984 | 2e-35 | 76.84 | 70.37 |
| H4_HUMAN | 9 | 6 | unplaced-368 | 1e-48 | 93.90 | 79.61 |
| H2B_EUPCR | 7 | 2 | scaffold-57 | 3e-43 | 66.09 | 98.23 |
| H2A_DROME | 8 | 6 | scaffold-57 | 3e-55 | 82.57 | 87.90 |
.jpg)