Я построила филогенетическое дерево тех же бактерий, что в предыдущих заданиях, используя последовательности РНК малой субъединицы рибосомы (16S rRNA). Для этого я взяла последовательности 16S рибосомальной РНК каждой из бактерий из базы полных геномов NCBI. Я брала геном случайно выбранного штамма нужного вида, названия штаммов приведены в таблице 1. В файле с расширением .frn я находила нужную мне последовательность 16S rRNA и добавляла ее в единый файл в fasta-формате с отредактированными названиями. Полученный файл я открыла в Jalview и выровняла программой Muscle. Файл с выравниванием я импортировала в программу MEGA (указав Analyze при импорте) и реконструируировала дерево методом "Neighbor Joining Using % Identity". Изображение полученного дерева на рис.1. Данное дерево очень похоже на "правильное" дерево, но есть небольшое различие. Дерево содержит ветвь {ENT38, ECOLI, SALTY, YERPS, VIBFM} против {NEIMA, HAEIN, PASMU} вместо ветви {ENT38, ECOLI, SALTY, YERPS, HAEIN, PASMU} против {NEIMA, VIBFM} в правильном дереве. Деревья, построенные по белкам, лечше отражают филогению организмов, так как аминокислотные последовательности более консервативны. Поэтому дерево, построенное по белкам, получилось достовернее дерева, построенного по последовательности РНК.
Таблица 1. Штаммы отобранных бактерии.
| Название | Мнемоника |
| Neisseria meningitidis 053442 uid58587 | NEIMA |
| Enterobacter 638 uid58727 | ENT38 |
| Escherichia coli DH1 uid161951 | ECOLI |
| Salmonella typhimurium DT104 uid223287 | SALTY |
| Yersinia pseudotuberculosis IP 31758 uid58487 | YERPS |
| Vibrio fischeri ES114 uid58163 | VIBFM |
| Haemophilus influenzae 10810 uid86647 | HAEIN |
| Pasteurella multocida 3480 uid161955 | PASMU |
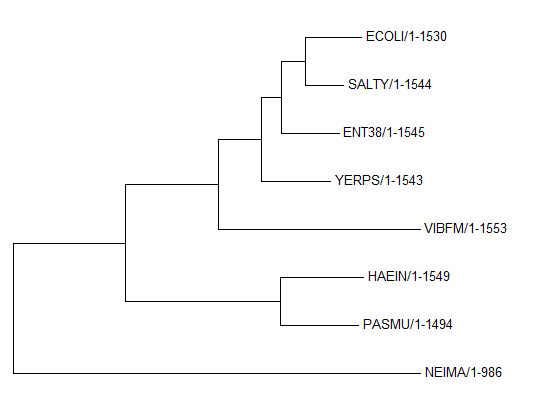
Рис.1 Филогенетическое дерево выбранных бактерий, полученное по последовательностям РНК малой субъединицы рибосомы (16S rRNA).
Построение и анализ дерева, содержащего паралогиДля того чтобы найти достоверные гомологи белка CLPX_ECOLI, я провела поиск программой blastp с порогом E-value 0,001 по протеомам отобранных мной бактерий. Я объединила протеомы нужных бактерий в один файл командой cat file1 >> file2, которая добавляет содержимое файла file1 в конец файла file2. Для запуска поиска я использовала команды: makeblastdb -in data.fasta -dbtype prot -out db.fasta и blastp -query CLPX_ECOLI.fasta -evalue 0.001 -db db.fasta -outfmt 6 -out out.fasta. Полученный файл: out.fasta. Среди полученных находок я считала бы достоверными гомологами те, которые имеют e-value меньше 1e-018. Для полученных белков я построила выравнивание программой MUSCLE. По полученному выравниванию я реконструируировала дерево методом "Neighbor Joining Using % Identity" в программе MEGA, филогенетическое дерево представлено на рис. 2.
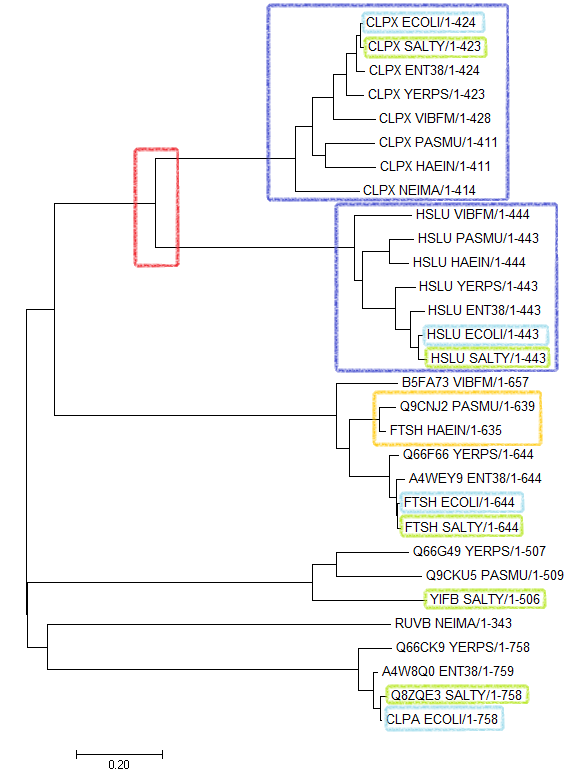
Рис.2 Филогенетическое дерево белков выбранных бактерий, гомологичных белку CLPX_ECOLI.
Пары паралогов (гомологичных белков из одного организма) выделены на рисунке салатовым и голубым цветом. Группа попарно ортологичных белков выделена синим цветом. Гомологичные белки будем называть ортологами, если они: а) из разных организмов; б) разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования. На рисунке выделены примеры отражённых на дереве эволюционных событий двух типов: дупликация гена (красныя рамка); разделение путей эволюции белков в результате видообразования (оранжевая рамка).
.jpg)