Для изучения структуры трансмембранных белков была использована цепь А белка мембранной натриевой фосфотазы из организма Thermotoga Maritima (идентификатор в PDB: 4AV3).
Была составлена репрезентативная выборка гомологов данного белка при помощи поиска BLAST на сайте EMBL EBI по кластерам UniRef50. Поиск велся по базе данных UniProt Clusters на уровне идентичности 50%. Число желаемых находок было увеличено до 200 (параметры SCORES и ALIGNMENTS). В итоге было отобрано 200 находок, e-value худшей отобранной находки равняется 8,5E-54. Было построено множественное выравнивание найденных белков и исходного белка с помощью программы Muscle. Полученное выравнивание в формате fasta: alignment.fasta.
Из полученного выравнивания были удалены следующие короткие последовательности: X1EKZ3, X1FQU8, R5Q2Z4, X1F0L6, A0A0G1KLG3, T0Y9Z3, X1HCK3, X0U0E6, A0A0G9I1G6, Q93NB7, X1PZP9, A0A0A9VWD8, Q8VNJ8, A0A090VAB9.
Множественное выравнивание было открыто в программе JalView, где была добавлена строка аннотации, названная "TM_REAL", в которой были отмечены буквой "М" участки выравнивания, отвечающие трансмембранным спиралям в белке со структурой. Для этого белок с известной структурой (идентификатор в PDB: 4AV3) был перемещен в первую строку выравнивания, и к нему была добавлена его трехмерная структура. Проект в JalView: project.jar.
Для одного из гомологов исследуемого белка (гомолог A0A0L6JS98) было получено предсказание трансмембранных спиралей, выдаваемых программой TMHMM. Графическое представление результата можно посмотреть на рис.1. В проекте JalView была добавлена строка аннотации "TM_PREDICTED", в которой буквой "Р" были отмечены участки, предсказанные TMHMM как трансмембранные, для белка A0A0L6JS98.
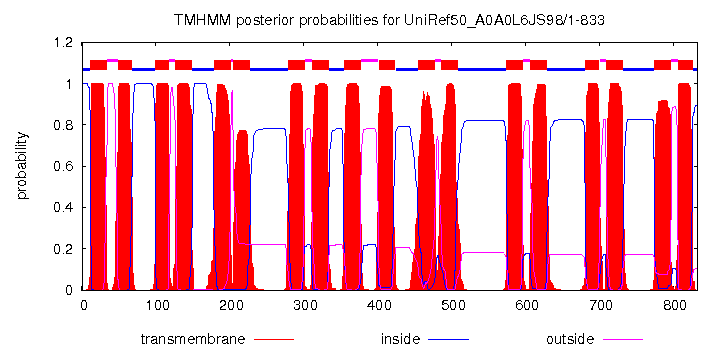
Рис.1 Результат предсказания трансмембранных участков для белка A0A0L6JS98 с помощью сервиса TMHMM.
Позиции, соответствующие трансмембранным спиралям, в основном представлены гидрофобными аминокислотами и являются функционально консервативными. Однако в центре белка на трансмембранных спиралях встречаются консервативные заряженные или просто полярные аминокислотные остатки, возможно, они нужны для связывания субстрата или других молекул или ионов. Участки между спиралями малоконсервативны и довольно сильно различаются по длине, меньше всего консервативны и больше всего различаются по длине крайние участки.
На рисунке 2 представлен белок, остатки которого покрашены с использованием цветовой схемы Hydrophobicity. Белок изображен так, чтобы ориентированная в n-сторону мембраны часть оказалась сверху, а ориентированная в p-сторону - снизу.
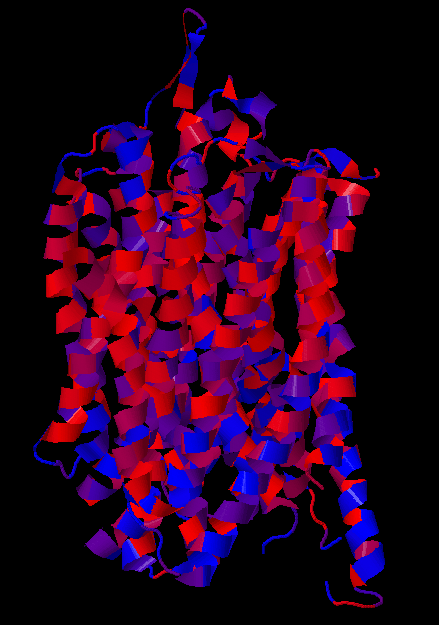
Рис.2 Белок 4AV3 с окраской по гидрофобности (JalView Hydrophobicity). Часть белка, обращенная к p-стороне мембраны, находится снизу.
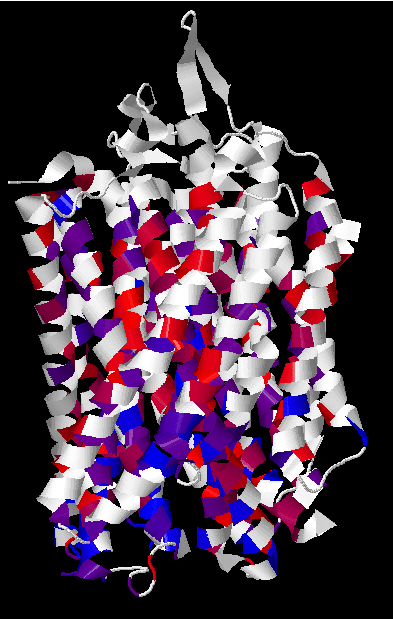
Рис.2 Белок 4AV3 с окраской по гидрофобности (JalView Hydrophobicity) и порогом идентичности 20%. Часть белка, обращенная к p-стороне мембраны, находится снизу.
В большинстве случаев предсказанные и реальные трансмембранные участки расположены в одном месте, но не совпадают по длине и своим границам. В целом, предсказанные спирали короче. Были найдены лишние спирали в начале и в конце последовательности. Данные различия связаны, скорее всего, с тем, что это гомологичные, но разные белки.
.jpg)