Учебный Сайт Николая Николаева
Мини-обзор генома Deinococcus peraridilitoris
Николай Николаев
Факультет биоинженерии и биоинформатики, Московский государственный университет имени М. В. Ломоносова, Ленинские горы д. 1 стр. 73, 119234, Москва, Россия
12 декабря 2020 года
Pdf-версияДополнительные материалыРЕЗЮМЕ
В данной работе был проведён анализ генома и протеома бактерии Deinococcus peraridilitoris с помощью электронных таблиц и ряда программ расширения emboss. Были получены данные о встречаемости k-меров в геноме, длине белков, вероятном расположении ориджина и терминатора репликации, распределении генов на прямой и обратной цепях хромосомы и плазмид.
Ключевые слова: геном, протеом, Excel, Deinococcus peraridilitoris.
1 ВВЕДЕНИЕ
Deinococcus peraridilitoris – аэробный грамположительный неподвижный кокк, не образующий спор, изолированный из почвы прибрежной пустыни в Чили в 2007 году1 и интересный своей исключительной устойчивостью к гамма-излучению: бактерия способна переносить дозы радиации свыше 10 кГр.
Геном D. Peraridilitoris представлен хромосомой и двумя плазмидами размером 3881839, 556630 и 75245 п.н. соответственно.
В данной работе главным образом с помощью электронных таблиц был проведён анализ некоторых характеристик генома и протеома бактерии, а именно: определён GC-состав генома, проведён его k-мерный анализ; исследовано соотношение G/C на разных участках бактериальной хромосомы для определения положения ориджина и терминатора репликации; составлена таблица количества генов различных типов на молекулах ДНК; вычислены средняя и медианная длина белка.
2 МАТЕРИАЛЫ И МЕТОДЫ
Данные о геноме и протеоме D. Peraridilitoris были получены с портала NCBI. Информация о генах была импортирована в Excel 2016. Путём поиска по тегам локусов (команда ВПР) была получена сводная таблица генов бактерии, содержимое которой было затем отсортировано по принадлежности к той или иной молекуле ДНК и расположению на ней.
k-мерный анализ генома проводился с помощью программы wordcount. Результаты были импортированы в Excel, где с по-мощью команд ВПР и ПСТР для каждого k-мера при k от 2 до 4 была подсчитана ожидаемая частота встречаемости и Compositional Bias (cb, отношение наблюдаемого значения к ожидаемому) на основании частот встречаемости нуклеотидов в гено-ме (результата для k=1).
Поиск ориджина и терминатора репликации был выполнен с помощью программы GenSkew, доступной по ссылке http://genskew.csb.univie.ac.at. Программа вычисляет GC-skew по формуле (G-C)/(G+C), где G, C – количество гуанинов и цитозинов в окне заданного размера. GC-skew cumulative (далее-GCsc) определяется сложением значений в данном окне и в нескольких предыдущих.
Как правило, у бактерий на лидирующей цепи преобладает гуанин, а на отстающей – цитозин2. Поскольку программа читает последовательность в направлении 5’-3’, в ориджине репликации значение GCsc будет наиболее характерным для отстающей цепи, т.е. минимальным, а в точке терминации репликации, наоборот, максимальным.
Подсчёт генов разных типов на «+» и «-»-цепях ДНК был выполнен с помощью формулы Excel СЧЁТЕСЛИМН.
Белок-кодирующие гены были выбраны из плоской таблицы всех генов формулой ЕСЛИ. Их количество было определено формулой СЧЁТЕСЛИ, средняя длина – СРЗНАЧ, медианная длина – МЕДИАНА.
Кроме вышеперечисленных действий, в электронных таблицах использовались арифметические операции, абсолютные и относительные ссылки, распространение формул и другие методы работы.
3 РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
3.1 K-мерный анализ генома и GC-состав
Для расчёта встречаемости k-меров были определены частоты встречаемости нуклеотидов (Таблица 1).
| Нуклеотид | Частота |
|---|---|
| A | 18.1% |
| C | 31.8% |
| G | 31.9% |
| T | 18.2% |
Видно соблюдение второго правила Чаргаффа – частоты комплементарных нуклеотидов примерно равны.
На Рис. 1 приведены гистограммы встречаемости k-меров для k=2, 3; в Таблице 2 – значения cb для некоторых тетрамеров (точные значения и полные результаты - см. дополнительные материалы, таблица «K-mers»).
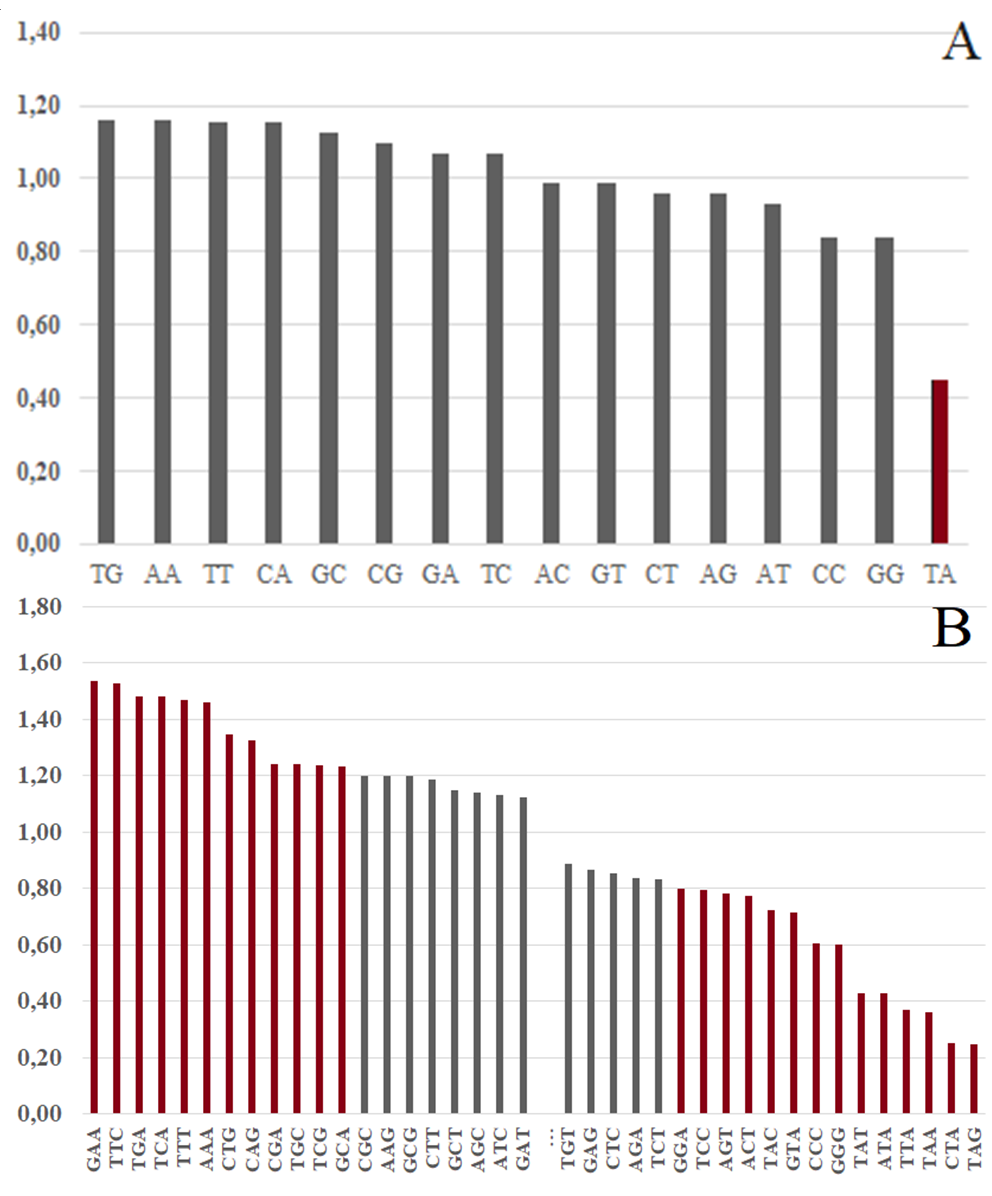
Наиболее сильно отклоняются от случайной встречаемости некоторые тетрамеры. Причину этого установить пока не удалось.
| Тетрамер | cb |
|---|---|
| TTTT | 2.06 |
| TGAA | 2.04 |
| AAAA | 2.03 |
| TTCA | 2.01 |
| TTAG | 0.11 |
| CTAA | 0.10 |
| CTAG | 0.06 |
3.2 Определение сайтов начала и терминации репликации по GC-skew
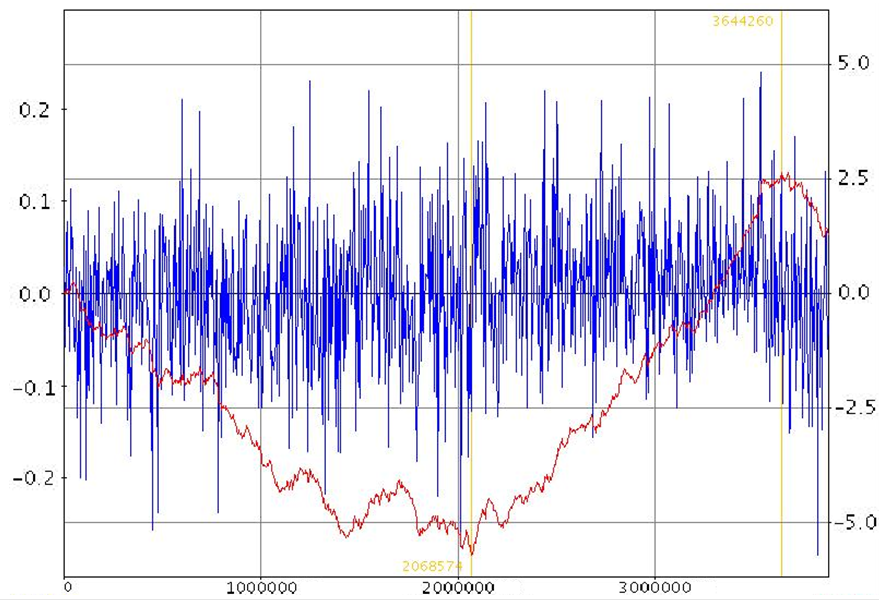
На Рис. 2 представлен результат работы программы GenSkew для хромосомы бактерии. Определить точки начала и конца репликации плазмид не удалось из-за их небольшого размера.
В то время как положение конечной точки репликации (около 3644 тыс. п.н. от начала последовательности) не вызывает сомнений, для ориджина есть три сайта со схожим GCsc с коор-динатами около 1.43, 1.82 и 2.04 млн. п.н. В то время как по-следний обладает наибольшим GCsc, второй ближе к положению, противоположному терминатору (1.70 млн. п.н.).
Для уточнения положения ориджина, возможно, стоит использовать другую программу с охватом большего числа окон при вычислении GCsc, чтобы ликвидировать случайные пики. Можно также провести поиск консервативных последователь-ностей точки начала репликации.
3.3 Распределение генов по «+» и «-» - цепям хромосомы и плазмид
Было исследовано, как гены различных типов распределены по молекулам ДНК в бактерии. Результаты (см. дополнительные материалы, таблица «Genes_distribution») свидетельствуют о том, что они в целом расположены равномерно. Значительно отклоняются от общего уровня в 1 ген примерно на каждые 2000 нуклеотидов лишь «+» и «-» - цепи меньшей плазмиды, что, вероятно, связано с её размером – 75.2 тыс. п.н. и 74 гена.
Пожалуй, наиболее интересным является наличие трёх генов рРНК на большей плазмиде, что, предположительно, является результатом её рекомбинации с хромосомой.
3.4 Анализ длин белков
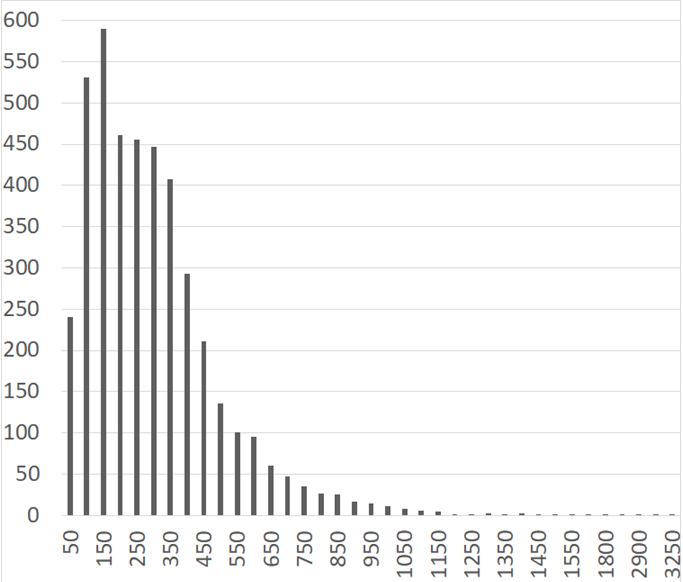
D. Peraridilitoris имеет 4246 белков, распределение которых по длине показано на Рис. 3. Наиболее часто встречаются белки с длиной 125-175 аминокислотных остатков. Медианная длина белка – 256 аминокислотных остатков; средняя – 298, что несколько меньше средней длины для бактерий - 320 аминокислотных остатков, к которой, к примеру, близка средняя длина белка у родственного Deinococcus deserti (314 остатков)3.
4 ЗАКЛЮЧЕНИЕ
В данной работе был проанализирован геном и протеом D. Peraridilitoris. Были построены таблицы и диаграммы встречаемости k-меров, выделены наиболее перепредставленные и недопредставленные; определены координаты сайта терминации репликации, три возможные координаты ориджина и предложены способы его точного нахождения в дальнейшем; построена таблица расположения генов на различных молекулах ДНК и показана равномерность их распределения по составным частям генома; построена гистограмма длин белков, определена средняя и медианная длина белка.
ДОПОЛНИТЕЛЬНЫЕ МАТЕРИАЛЫ
Электронная таблица Nikolaev_supple_fin:
- Лист «K-mers» - Compositional bias для всех k-меров с k=2,3,4.
- Лист «Genes_distribution» - Количество кодирующих белки генов, псевдогенов и генов рибосомальных РНК на «+» и «-» - цепях хромосомы и двух плазмид.
ИСТОЧНИКИ
- Rainey FA, Ferreira M, Nobre MF, Ray K, Bagaley D, Earl AM, и др. Deinococcus peraridilitoris sp. nov., isolated from a coastal desert. International Journal of Systematic and Evolutionary Microbiology. 1 июля 2007 г.;57(7):1408–12.
- Tillier ERM, Collins RA. The Contributions of Replication Orientation, Gene Direction, and Signal Sequences to Base-Composition Asymmetries in Bacterial Genomes. J Mol Evol. март 2000 г.;50(3):249–57.
- Tiessen A, Pérez-Rodríguez P, Delaye-Arredondo LJ. Mathematical modeling and comparison of protein size distribution in different plant, animal, fungal and microbial species reveals a negative correlation between protein size and protein number, thus providing insight into the evolution of proteomes. BMC Res Notes. декабрь 2012 г.;5(1):85.