Учебный Сайт Николая Николаева
Назад на страницу семестра
Сборка генома de novo.
Файлы TruSeq3-SE.fa и TruSeq2-SE.fa из папки adapters были скопированы в рабочую директорию и объединены в файл TruSeq-SE.fa.
Геном собирался следующей последовательностью программ:
- java -jar /usr/share/java/trimmomatic.jar SE SRR*fastq.gz trim1.fastq.gz ILLUMINACLIP:./TruSeq-SE.fa:2:7:7 &> trim1.log
- java -jar /usr/share/java/trimmomatic.jar SE trim1.fastq.gz trim2.fastq.gz MINLEN:32 TRAILING:20 &> trim2.log
- velveth . 31 -short -fastq trim2.fastq.gz &> velveth.log
- velvetg . -scaffolding no &> velvetg.log
После первого запуска trimmomatic отбросил 97710 (1.87%) чтений - они оказались остатками адаптеров.
После второго запуска было удалено 75915 (1.48%) чтений; размер файла уменьшился с 110.8 мб (trim1.fq.gz) до 106.8 мб (trim2.fq.gz).
N50 сборки - 12382, среднее покрытие - 25.77. Список контигов, их длин и покрытий, а также вычисление среднего покрытия доступны в таблице contigs-info.xlsx
- Самые длинные контиги: Node_3 (длина: 25915, покрытие: 27.42); Node_20 (длина: 23850, покрытие: 24.78); Node_22 (длина: 23807, покрытие: 25.75)$
- Контиг с аномально высоким покрытием: Node_55 (длина: 934, покрытие: 130.49);
- Некоторые контиги с аномально низким покрытием: Node_228 (длина: 62, покрытие: 2.42); Node_106 (длина: 80, покрытие: 2.70).
Анализ сборки.
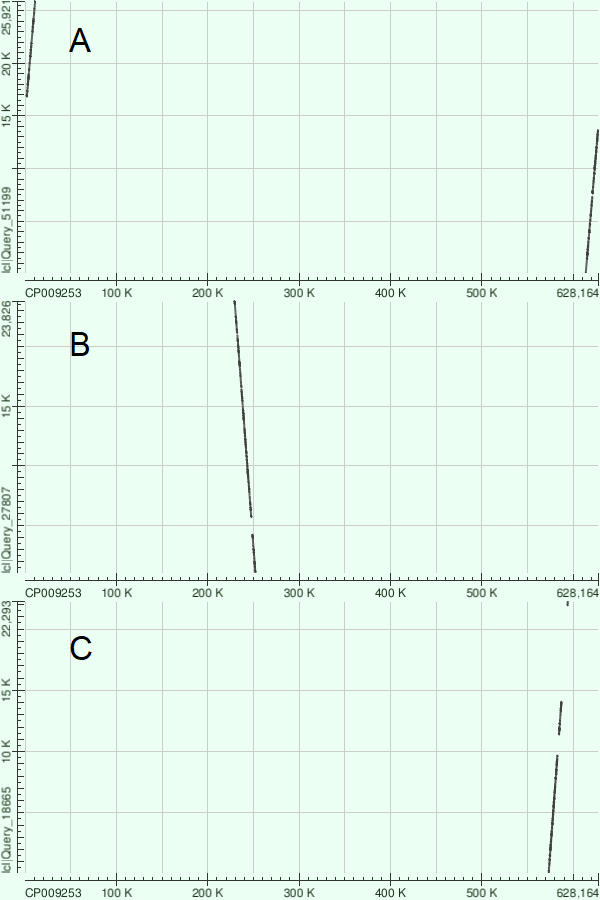
Контиги Node_3. Node_20 и Node_22 были выровнены на готовый геном с помощью megablast. Для каждого из них лишь одно выравнивание прошло порог E-value 0.05. Каждое имеет характеристики, позволяющие с высокой долей уверенности утверждать, что контиги найдены на хромосоме правильно (см. Табл.1).| Контиг | Координаты на хромосоме | E-value | Несовпадения нуклеотидов | Гэпы |
|---|---|---|---|---|
| Node_3 | 613658-11103 | 0.00 | 1748 | 244 |
| Node_20 | 252164-229411 | 0.00 | 2315 | 387 |
| Node_22 | 573092-594099 | 0.00 | 2147 | 463 |