Учебный Сайт Николая Николаева
Назад на страницу семестра
Паралоги, визуализация.
Составление списка гомологичных белков, включающих паралоги.
Протеомы были объединены в файл all.fasta. Поиск гомологов производился командой
blastp -query CLPX_ECOLI.fasta -db all.fasta -out all.txt -evalue 0.001
-outfmt "7 saccver qstart qend sstart send length qcovs pident gapopen evalue"
Список находок представлен по ссылке.
Из их названий был создан файл all-names.txt. Последовательности белков были получены командой
seqret @all-names.txt -outseq homologs.fasta
и выровнены программой Muscle.
Реконструкция и визуализация.
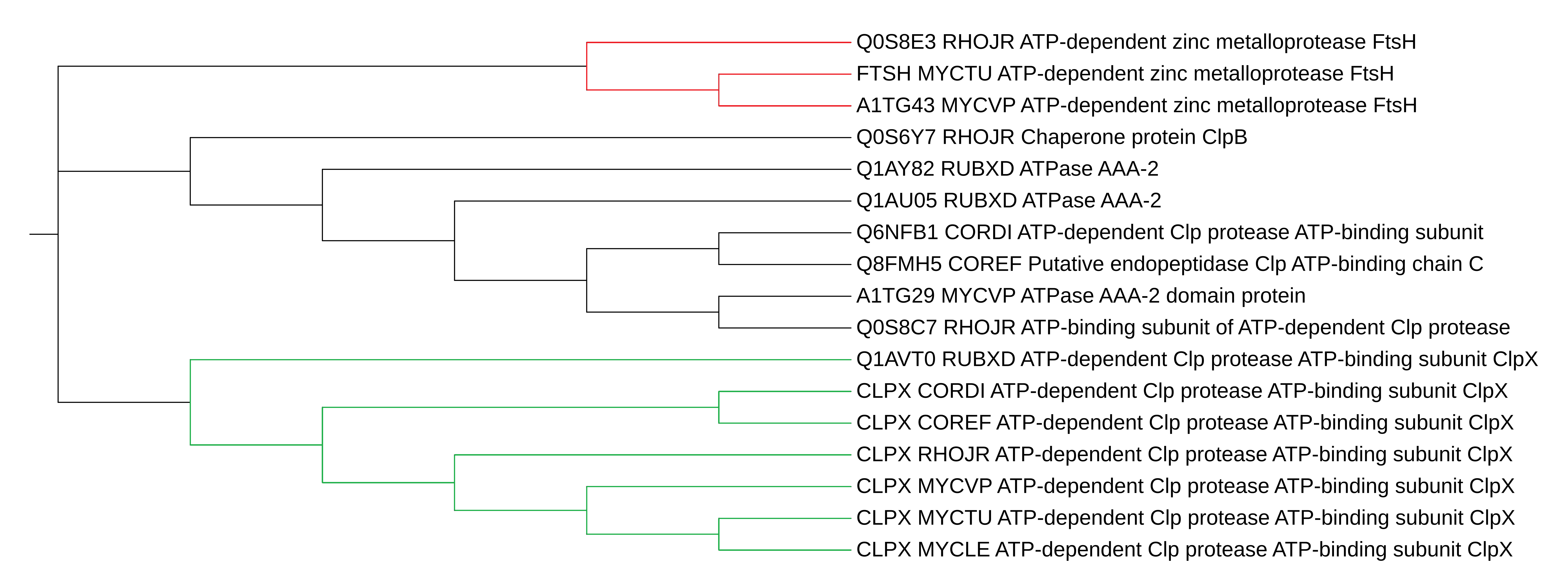
Дерево было реконструировано в MEGA методом Neighbor-joining. Оно доступно по ссылке.
Примеры ортологичных белков:
- CLPX COREF и CLPX CORDI;
- FtsH из MYCTU и MYCVP;
- CLPX MYCLE и CLPX CORDI.
Примеры паралогов:
- CLPX RHOJR и FtsH RHOJR;
- Putative endopeptidase Clp ATP-binding chain C COREF и CLPX COREF;
- Chaperone protein ClpB RHOJR и FtsH RHOJR.